中国圈养食蟹猴TrimCyp基因频率
2013-09-20蒙裕欢柳明玉陈军辉杜红丽王小宁
田 帅,蒙裕欢,柳明玉,孙 飞,陈军辉,杜红丽,王小宁
华南理工大学 生物科学与工程学院,广州 510006
TRIM5α是TRIM蛋白家族的一员,由TRIM5 初级转录产物经过可变剪切产生。人TRIM5α由493个氨基酸组成,具有 RING、B-box2、coiled-coil以及C'末端的B30.2/SPRY等4个结构域。TRIM5α蛋白作为逆转录病毒衣壳特异性的限制因子,具有抗HIV-1感染的作用,且由于它能够在病毒进入细胞后通过阻碍其反转录过程来限制病毒感染,故属于进入后限制作用 (Stremlau et al, 2004; Tang et al,2009)。研究表明,TRIM5α的病毒限制作用与各结构域的结构和功能紧密相关。RING结构域具有E3泛素连接酶功能,能够促进TRIM5α自身泛素化,增强蛋白与蛋白之间的相互作用(Mische et al,2005)。B-box2结构域为抑制病毒逆转录所必须,与TRIM5α同源多聚体的相互作用有关 (Mische et al, 2005),该区域在一定程度上能够通过调节TRIM5α高度有序的自我装配而增强对HIV-1的限制作用(Diaz-Griffero et al, 2009)。Coiled-coil功能域对TRIM5α蛋白二聚体的形成和稳定有重要作用,只有 TRIM5α同源二聚体才具有抗慢病毒的功能(Stremlau et al, 2004),且coiled-coil结构域也参与决定抗病毒特异性,对TRIM5α特异性识别和结合病毒衣壳有一定作用(Maillard et al, 2010)。SPRY/B30.2结构域的V1、V2和V3三个可变区域对病毒衣壳特异性具有重要作用。B30.2区域的这3个可变区域均可形成 Loop结构,该结构有利于TRIM5α蛋白与病毒特异性识别与结合 (Ohkura et al, 2006)。该区域的N端部分对抗HIV-1有决定作用 (Yap et al, 2005),C末端SPRY/B30.2结构域是抗逆转录病毒所必需的,也是识别结合HIV-1衣壳必不可少的关键区域 (Lim et al, 2010)。在HIV-1感染靶细胞的早期阶段,细胞内的TRIM5α蛋白通过B30.2结构域与入侵的HIV-1衣壳蛋白相互作用,并且在RING结构域E3泛素连接酶的作用下,通过促进HIV-1脱壳或加速衣壳蛋白降解来阻碍反转录过程的顺利进行,从而抑制HIV-1后期逆转录产物的产生 (Stremlau et al, 2006)。
TRIMCyp蛋白首先在鹰猴 (Aotus trivirgatus)中被发现,鹰猴体内表达的一种融合蛋白TRIMCyp能够限制HIV-1感染。TRIMCyp蛋白保留了TRIM蛋白家族N末端的RBCC三模序列,只是TRIM5α蛋白的B30.2结构域被亲环素CypA (Cyclophilin A)代替。在鹰猴中,CypA基因通过逆转座作用插入TRIM5基因的第 7和第 8外显子之间,形成TRIM5-CypA融合基因,TRIMCyp同样通过形成同源二聚体而发挥限制作用 (Nisole et al, 2004; Sayah et al, 2004)。鹰猴 TRIMCyp以与其他灵长类TRIM5α相似的机制限制 HIV-1感染,都能够在反转录病毒进入靶细胞后的早期阶段限制其感染。亲环蛋白CypA是一种肽基脯氨酰异构酶,能够结合HIV-1衣壳表面富含脯氨酸的回环结构,这种相互作用增加了人对HIV-1的易感性,同时也增强了非人类灵长类动物限制 HIV-1的活性 (Sokolskaja &Luban, 2006)。TRIMCyp除了在新大陆猴鹰猴体内被发现以外,也在旧大陆猴体内被发现,至少4种猴子体内有 TRIMCyp融合蛋白:印度恒河猴(Macaca mulatta)、平顶猴 (Macaca nemestrina)、熊猴(Macaca assamensis)以及食蟹猴(Macaca fascicularis)(Brennan et al, 2008; Cao et al, 2011;Liao et al, 2007; Newman et al, 2008; Virgen et al,2008; Wilson et al, 2008),这些融合基因均由 CypA基因通过L1介导的逆转座作用插入TRIM5 基因的3’UTR而形成,它们均能经转录和翻译产生由TRIM5α的2~6号外显子和完整CypA结构域组成的 TRIMCyp蛋白。恒河猴和平顶猴的 TRIMCyp不能限制HIV-1感染,却能够限制HIV-2和FIV的活性(Brennan et al, 2008; Liao et al, 2007; Newman et al, 2008; Virgen et al, 2008; Wilson et al, 2008)。最新研究表明,食蟹猴中的TRIMCyp多态性具有功能差异 (Dietrich et al, 2011),TRIMCyp主要单体型(DK)即TRIMCyp融合蛋白CypA结构域中的第66位天冬氨酸(D)和第 143位赖氨酸(K)可限制 HIV-1的作用,但不能限制HIV-2。TRIMCyp次要单体型(NE)即TRIMCyp融合蛋白CypA结构域中的第66位天冬酰胺(N)和第 143位谷氨酸(E)能够限制HIV-2,却不能限制HIV-1感染 (Saito et al, 2012a),两种单体型功能完全相反,提示TRIMCyp的多态性对抗病毒效应有一定的影响,因此携带TRIMCyp NE单体型的食蟹猴有可能构建成为HIV-1感染的动物模型。
TRIMCyp的等位基因频率存在地域偏差,虽然食蟹猴 TRIMCyp基因的频率在东南亚不同国家或地区已经被初步调查 (Berry et al, 2012; Dietrich et al, 2011; Saito et al, 2012a; Saito et al, 2012b)(表 1),但是,中国大陆食蟹猴养殖场的 TRIMCyp基因频率还未被明确阐明,本研究对中国5个省11个养殖场共 1 594个食蟹猴繁殖种群随机样本中的TRIMCyp基因频率进行了筛查研究,以期为进一步开展食蟹猴HIV-1动物模型和HIV-1发病机制研究奠定基础。
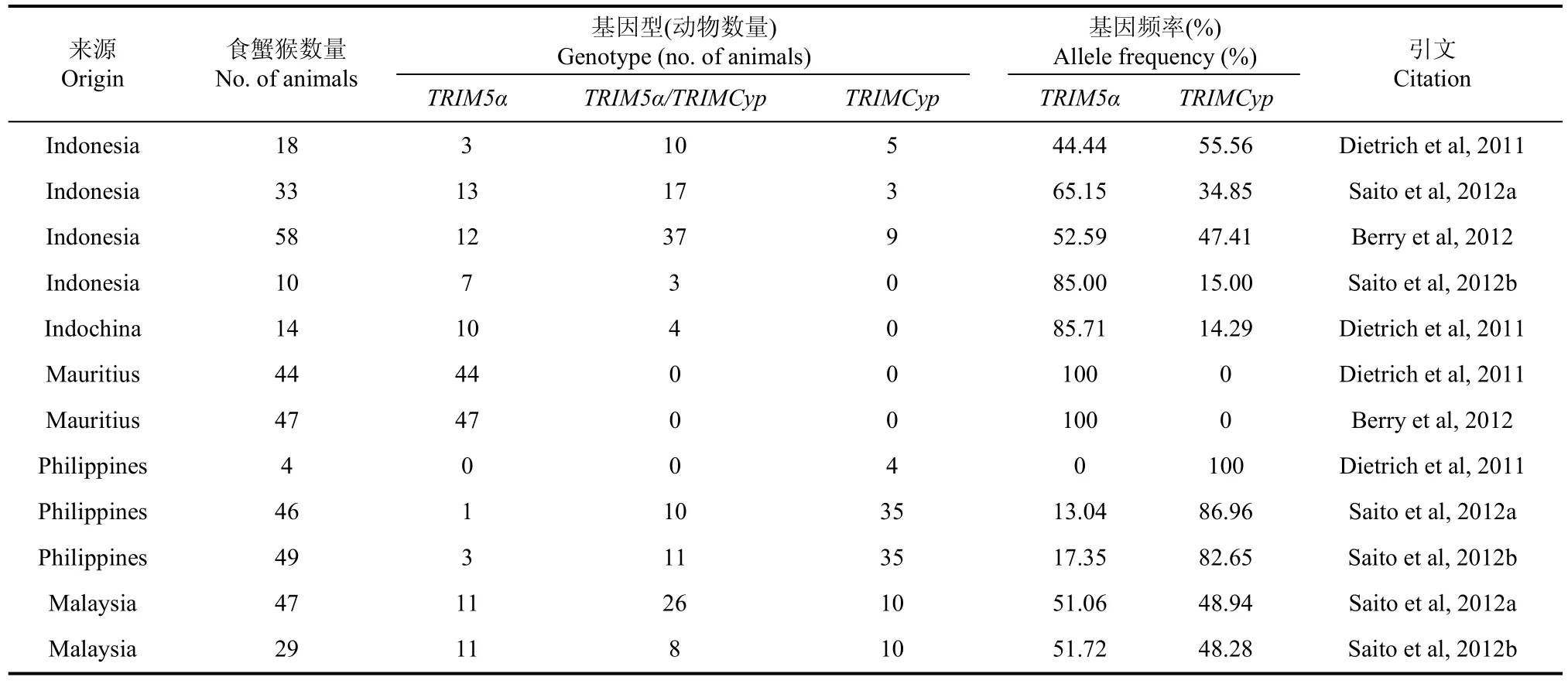
表1 东南亚不同地区食蟹猴TRIMCyp等位基因频率Table 1 Frequency of TRIMCyp alleles of cynomolgus macaques in Southeast Asia
1 材料与方法
1.1 样品来源与DNA抽提
本研究在广东、广西、云南、海南和江苏5个省 11个养殖场的食蟹猴繁殖种群中随机采集了 1 594个血样,EDTA抗凝于-20 ℃保存备用。基因组DNA抽提试剂盒购自北京普博生物科技有限公司,按照说明书进行基因组DNA提取。
1.2 引物设计与合成
分别根据TRIM5α第8外显子和3'UTR序列设计CypA筛查引物,引物为TrimcypF: ATGACTCTG TGCTCACCAAG,TrimcypR:AACTCTAGTCACC CTACTATG,由上海生工生物公司合成。
1.3 PCR扩增及测序
以基因组 DNA为模板,通过 PCR扩增筛查CypA插入情况,并对带有CypA个体进行测序。PCR扩增所用Taq酶为TaKaRa LA Taq酶(宝生物工程(大连)有限公司)。反应条件为:94 ℃ 5 min;94 ℃40 s,退火温度59 ℃ 30 s,72 ℃ 2 min,35个循环;72 ℃ 5 min。CypA插入片段的测序验证:回收1 000 bp和1 700 bp的PCR产物凝胶,送交深圳华大基因科技有限公司 (广州)进行上、下游引物测序。
2 结果
2.1 PCR和测序
PCR扩增结果见图1,PCR产物凝胶回收后的上、下游引物测序结果与预期序列一致。
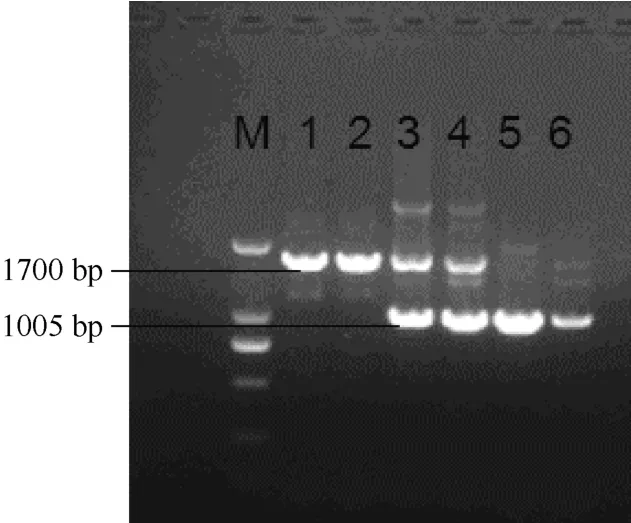
图1 PCR产物电泳情况Figure 1 Electrophoresis image of PCR product
2.2 基因分型和等位基因频率
从5个省11个养殖场共1 594个食蟹猴样本统计来看,共有379个个体含有TRIMCyp融合基因,占总数的 23.78%,其中纯合子47个(12.40%),杂合子332个(87.60%)。TRIM5α和TRIMCyp基因频率具体结果见表 2。在我们筛查的中国圈养食蟹猴中,TRIM5α基因频率显著高于TRIMCyp融合基因频率,分别是86.64%和13.36%;TRIMCyp融合基因频率(7.65%~19.79%)高于毛里裘斯食蟹猴的TRIMCyp融合基因频率(0%),显著低于印度尼西亚、菲律宾和马来西亚各国的 TRIMCyp融合基因频率(34.85%~100%),而与印度尼西亚 (Saito et al,2012b)和中印半岛食蟹猴 TRIMCyp融合基因频率无明显差异(表1)。
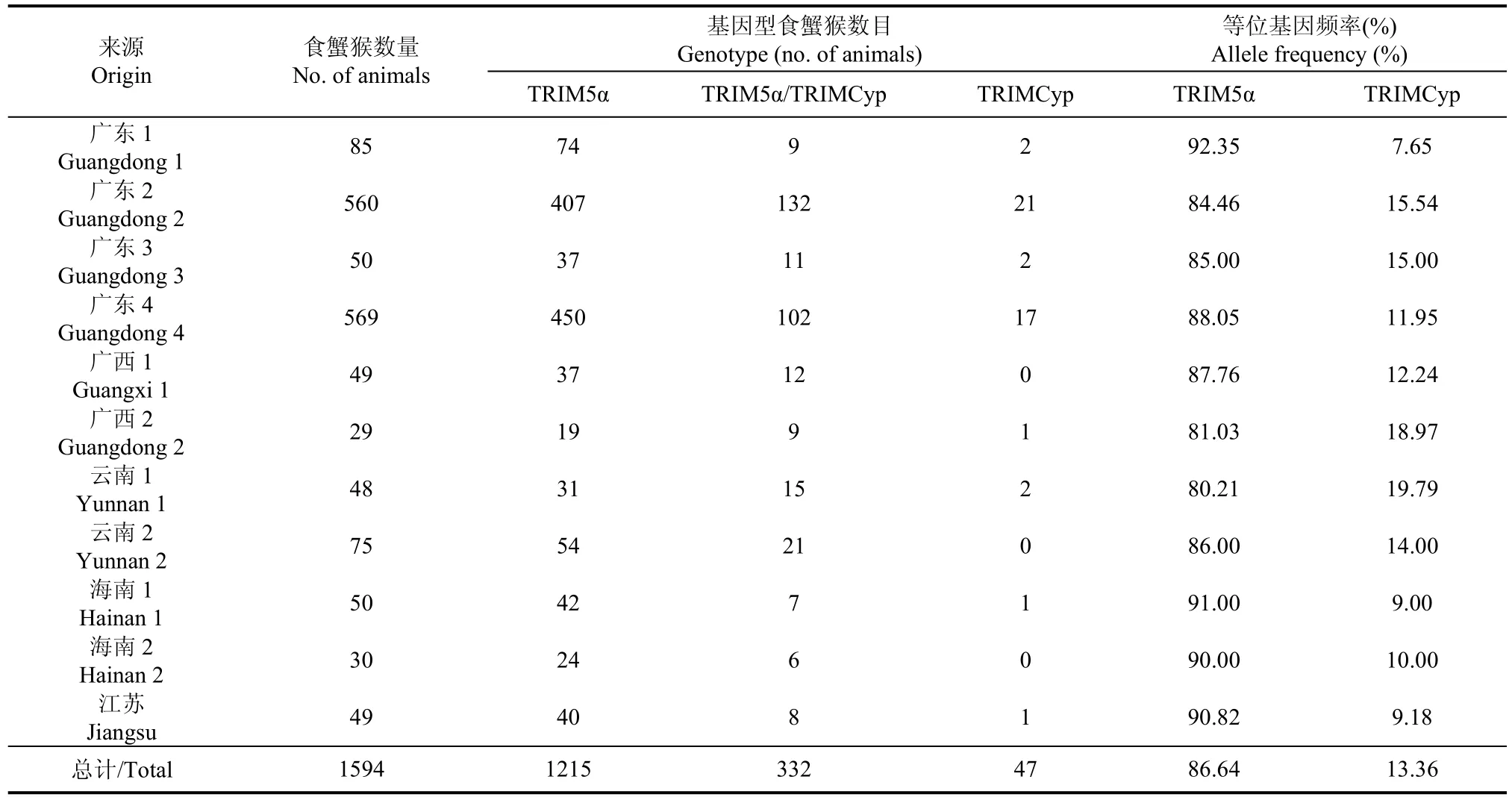
表2 中国圈养食蟹猴TRIM5α与TRIMCyp基因频率统计Table 2 Frequencies of cynomolgus TRIM5 alpha and TRIMCyp genes in China
2.3 TRIMCyp单倍型统计
将含有TRIMCyp融合基因的个体进行CypA测序,并对CypA第66位氨基酸(D/N)和143位氨基酸(K/E)的个体进行统计,结果显示NE单倍型食蟹猴个体很少,仅20个个体,其中只有一个是NE单倍型纯合子(表 3),且中国圈养食蟹猴 NE单倍型频率 (4.93%)也显著低于东南亚三个国家食蟹猴NE单倍型频率 (11.1%~14.3%)(Saito et al, 2012a)。
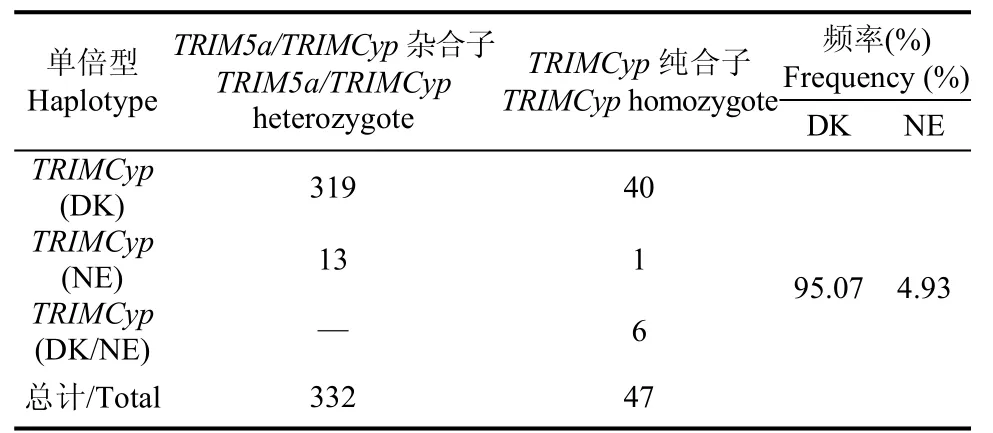
表3 中国圈养食蟹猴TRIMCyp DK和NE单倍型频率Table 3 Frequencies of cynomolgus TRIMCyp haplotypes DK&NE in China
3 讨论
本研究初步筛查了我国境内食蟹猴 TRIMCyp融合基因和 CypA单倍型的频率,而对食蟹猴TRIMCyp融合基因对不同逆转录病毒的抗毒效应未进一步研究。灵长类动物 TRIMCyp融合基因从发现至今,研究者除了对新大陆猴鹰猴 TRIMCyp融合蛋白限制HIV-1复制的作用机制有一定了解以外,旧大陆猴 TRIMCyp融合蛋白不能限制 HIV-1感染的具体机制到现在还没有确切结论。最近发现CypA上几个位点的多态性会影响TRIMCyp融合蛋白的限制逆转录病毒活性,其中CypA结构域中第54位氨基酸的单点突变(H54R)决定了食蟹猴TRIMCyp融合蛋白限制 HIV-1和 FIV的能力(Ylinen et al, 2010)。进一步研究除证实了该结论以外,还发现TRIMCyp融合蛋白CypA结构域中的第 66位和 143位氨基酸与印度尼西亚食蟹猴TRIMCyp融合蛋白的限制逆转录病毒活性密切相关,其中66位为D,143位为K的突变能够限制HIV-1复制 (Dietrich et al, 2011)。由于不同灵长类动物来源的TRIMCyp融合蛋白在其CypA结构域中的许多氨基酸位点存在差异,导致其 TRIMCyp融合蛋白与不同逆转录病毒衣壳蛋白Gag的识别和结合能力也存在差异,继而导致其抑制活性上的差异 (Cao et al, 2012)。在我国境内含TRIMCyp融合基因的食蟹猴,绝大部分是以TRIM5α/TRIMCyp杂合子的形式出现 (87.60%),在东南亚不同国家或地区的 TRIM5α/TRIMCyp杂合子频率是 53.16%;而TRIMCyp的基因频率甚至高达100%(Philippines),远远高于中国圈养的平均水平 (13.36%)。原因可能是由于前者是建立于1978年的封闭群。同时,Saito et al (2012a)对印度尼西亚食蟹猴的两次研究显示不同的 TRIMCyp融合基因频率 (15.00%和34.85%),而在毛里裘斯食蟹猴中则未发现TRIMCyp融合基因。另外,我们发现在中国圈养的NE单倍型食蟹猴个体极少,NE单倍型频率(4.93%)也显著低于东南亚三个国家的食蟹猴NE单倍型频率 (11.1%~14.3%)。因此,如果需要构建中国圈养食蟹猴HIV-1感染模型的资源群,需要大量筛查中国圈养携带 TRIMCyp融合基因的食蟹猴,并根据TRIMCyp单倍型来构建封闭群。另外,研究TRIMCyp不同单倍型与HIV-1和HIV-2感染的相关性,有助于进一步揭示HIV的感染机制。TRIMCyp融合基因的发现和研究为 HIV-1研究提供策略基础,为探讨TRIMCyp与其他抗HIV-1固有免疫分子的相互关系提供新思路,并为建立更理想的HIV-1/AIDS动物模型提供科学依据。
Berry NJ, Marzetta F, Towers GJ, Rose NJ. 2012. Diversity of TRIM5α and TRIMcyp sequences in cynomolgus macaques from different geographical origins. Immunogenetics, 64(4): 267-278.
Brennan G, Kozyrev Y, Hu SL. 2008. TRIMcyp expression in old world primates macaca nemestrina and macaca fascicularis. Proceedings of the Natlional Academy of Sciences of the United States of America, 105(9):3569-3574.
Cao G, Liu FL, Zhang GH, Zheng YT. 2012. The primate TRIMcyp fusion genes and mechanism of restricting retroviruses replication. Zoological Research, 33(1): 99-107. [曹光, 刘丰亮, 张高红, 郑永唐. 2012. 灵长类动物TRIMCyp融合基因模式及对逆转录病毒复制的限制作用. 动物学研究, 33(1): 99-107.]
Cao G, Nie WH, Liu FL, Kuang YQ, Wang JH, Su WT, Zheng YT. 2011.Identification of the TRIM5/TRIMcyp heterozygous genotype in macaca assamensis. Zoological Research, 32(1): 40-49. [曹光, 佴文惠, 刘丰亮,况轶群, 王金焕, 苏伟婷, 郑永唐. 2011. 熊猴存在TRIM45/TRIMCyp杂合子基因型. 动物学研究, 32(1): 40-49.]
Diaz-Griffero F, Qin XR, Hayashi F, Kigawa T, Finzi A, Sarnak Z, Lienlaf M, Yokoyama S, Sodroski J. 2009. A b-box 2 surface patch important for trim5alpha self-association, capsid binding avidity, and retrovirus restriction.Journal of Virology, 83(20): 10737-10751.
Dietrich EA, Brennan G, Ferguson B, Wiseman RW, O'Connor D, Hu SL.2011. Variable prevalence and functional diversity of the antiretroviral restriction factor trimcyp in macaca fascicularis. Journal of Virology, 85(19):9956-9963.
Liao CH, Kuang YQ, Liu HL, Zheng YT, Su B. 2007. A novel fusion gene,trim5-cyclophilin a in the pig-tailed macaque determines its susceptibility to hiv-1 infection. AIDS, 21(Suppl 8): S19-S26.
Lim SY, Chan T, Gelman RS, Whitney JB, O'Brien KL, Barouch DH,Goldstein DB, Haynes BF, Letvin NL. 2010. Contributions of Mamu- A*01 status and TRIM 5 allele expression, but not CCL3L copy number variation,to the control of sivmac251 replication in indian-origin rhesus monkeys.PLoS Genet, 6(6): e1000997.
Maillard PV, Ecco G, Ortiz M, Trono D. 2010. The specificity of TRIM5 alpha-mediated restriction is influenced by its coiled-coil domain. Journal of Virology, 84(11): 5790-5801.
Mische CC, Javanbakht H, Song B, Diaz-Griffero F, Stremlau M, Strack B,Si Z, Sodroski J. 2005. Retroviral restriction factor TRIM5alpha is a trimer.Journal of Virology, 79(22): 14446-14450.
Newman RM, Hall L, Kirmaier A, Pozzi LA, Pery E, Farzan M, O'Neil SP,Johnson W. 2008. Evolution of a TRIM5-cypa splice isoform in old world monkeys. PLoS Pathog, 4(2): e1000003.
Nisole S, Lynch C, Stoye JP, Yap MW. 2004. A TRIM5-cyclophilin a fusion protein found in owl monkey kidney cells can restrict hiv-1. Proceedings of the National Academy of Sciences of the United States of America, 101(36):13324-13328.
Ohkura S, Yap MW, Sheldon T, Stoye JP. 2006. All three variable regions of the TRIM5alpha b30.2 domain can contribute to the specificity of retrovirus restriction. Journal of Virology, 80(17): 8554-8565.
Saito A, Kono K, Nomaguchi M, Yasutomi Y, Adachi A, Shioda T, Akari H,Nakayama EE. 2012a. Geographical, genetic and functional diversity of antiretroviral host factor trimcyp in cynomolgus macaque (Macaca fascicularis). Journal of General Virology, 93(Pt 3): 594-602.
Saito A, Kawamoto Y, Higashino A, Yoshida T, Ikoma T, Suzaki Y, Ami Y,Shioda T, Nakayama EE, Akari H. 2012b. Allele frequency of antiretroviral host factor trimcyp in wild-caught cynomolgus macaques (Macaca fascicularis). Frontiers in Microbiology, 3: 314.
Sayah DM, Sokolskaja E, Berthoux L, Luban J. 2004. Cyclophilin a retrotransposition into trim5 explains owl monkey resistance to hiv-1.Nature, 430(6999): 569-573.
Sokolskaja E, Luban J. 2006. Cyclophilin, TRIM5, and innate immunity to hiv-1. Current Opinion Microbiology, 9(4): 404-408.
Stremlau M, Owens CM, Perron MJ, Kiessling M, Autissier P, Sodroski J.2004. The cytoplasmic body component trim5alpha restricts hiv-1 infection in old world monkeys. Nature, 427(6977): 848-853.
Stremlau M, Perron M, Lee M, Li Y, Song B, Javanbakht H, Diaz-Griffero F,Anderson DJ, Sundquist WI, Sodroski J. 2006. Specific recognition and accelerated uncoating of retroviral capsids by the trim5alpha restriction factor. Proceedings of the National Academy of Sciences of United States of America, 103(14): 5514-5519.
Tang X, Kuang YQ, Zheng YT. 2009. Research advance of TRIM5 α on structure and restriction mechanism to hiv-1 replication. Chinese Journal of
Virology, 25(2): 148-153. [汤霞, 况轶群, 郑永唐. 2009. TRIM5α分子结构和限制HIV-1复制机制的研究进展. 病毒学报, 25(2): 148-153.]
Virgen CA, Kratovac Z, Bieniasz PD, Hatziioannou T. 2008. Independent genesis of chimeric TRIM5-cyclophilin proteins in two primate species.Proceedings of the National Academy of the Scienes of the United States of America, 105(9): 3563-3568.
Wilson SJ, Webb BL, Ylinen LM, Verschoor E, Heeney JL, Towers GJ.2008. Independent evolution of an antiviral trimcyp in rhesus macaques.Proceedings of the National Academy of the Sciences of the United States of American, 105(9): 3557-3562.
Yap MW, Nisole S, Stoye JP. 2005. A single amino acid change in the spry domain of human Trim5alpha leads to hiv-1 restriction. Current Bioloinggy,15(1): 73-78.
Ylinen LMJ, Price AJ, Rasaiyaah J, Hué S, Rose NJ, Marzetta F, James LC,Towers GJ. 2010. Conformational adaptation of asian macaque trimcyp directs lineage specific antiviral activity. PLoS Pathog, 6(8): e1001062.
