新疆富蕴地震断裂带植被恢复对土壤古菌群落的影响
2013-09-11林青,曾军,张涛,马晶,王重,娄恺,*
林 青,曾 军,张 涛,马 晶,王 重,娄 恺,*
(1.新疆农业科学院微生物应用研究所,乌鲁木齐 830091;2.新疆大学生命科学与技术学院,乌鲁木齐 830046)
地震引发的各种次生地质灾害,造成景观破碎,土壤结构破坏,土壤微生物平衡体系发生改变,使震区生态系统变得脆弱、敏感,严重破坏当地生态环境[1-2]。1931年8月11日,发生于新疆阿勒泰富蕴县境内的8.0级大地震,形成了一条长达176 km、世界罕见的地震断裂带[3],严重破坏了原有植被和表层土壤,形成大面积次生裸地,震后至今,随着植物次生演替,震区生态进入自然恢复过程。恢复生态学认为,植被恢复是恢复退化生态系统的首要问题,因为它几乎是所有自然生态系统恢复的前提[4]。
其它退化生态系统研究表明,植物与土壤微生物关系密切:植物物种组成、群落结构及不同演替阶段影响根际土壤微生物分布、酶活、微生物碳、氮含量等[5-7],而土壤微生物通过竞争、协调、驱动养分循环等作用影响着植物多样性[8]。可见,植被恢复与土壤微生物紧密相关,它们维系着生态系统的稳定和健康。然而目前对震后生态研究多集中于森林、水体、农业等宏观系统受损的调查与恢复[2,9],微观的土壤微生物国内外未见报道。随着分子生物学的发展,古菌的分布与研究范围已从狭隘的极端环境,发展到海洋、湖泊、土壤等非极端环境,同时由古菌介导的有机碳降解转化,甲烷生成氧化,氨氧化等对全球碳、氮循环起重要作用[10]。为此本文利用末端限制性片段长度多态性(Terminal restriction fragment length polymorphism,T-RFLP)技术,对新疆富蕴地震断裂带次生植物根际土壤的古菌群落进行分析,为探究震后植被恢复与土壤微生物的关系提供科学参考。
1 材料与方法
1.1 供试地概况及样品采集
富蕴地震断裂带高烈度区(Ⅹ—Ⅺ度)位于卡拉先格尔一带,此处基岩崩裂破碎,地震断裂纵横交错,山体两侧滚石遍布,山体大面积滑塌,在山腰处形成长1500 m,水平跨度350 m,滑塌面最大高度达63 m的塌陷区[11]。观测塌陷土壤剖面,塌陷区内无表土层及心土层只剩底土层,形成贫瘠的次生裸地。震后至今虽有草本、灌木植物于区内生长,但由于大部分滑塌面几乎与水平垂直,植物难以附着,种群密度稀疏且零星分布,大面积仍为裸地,又因地处山区人迹罕至,植物间与采样环境基本无干扰。
2009年8月(植物生长旺季)在塌陷区(北纬46°44'25″,东经89°54'15″,海拔1901 m)进行采样。经植物调查,裸地共有13种草本及灌木植物生长,群落演替至灌木阶段,在300 m×30 m(滑塌面呈狭长型)可攀爬范围,选取草本、灌木优势植物各4种,东方旱麦草(Eremopyrum orientale),西北绢蒿(Seriphidium nitrosum),,鼠掌老鹳草(Geranium sibiricum),蓬子菜(Galium verum)(草本),皱纹柳(Salix vistita),直穗柳(Salix rectijulis)欧亚绣线菊(Spiraea media),密刺蔷薇(Rosa spinosissima)(灌木)。采集其根际土壤,因土壤呈砂质,极易散落,无法附着于根系,故除去表层1 cm土壤后,在0—20 cm深度采用内径为5 cm的土壤取样器,以植物根系为轴心钻取根围2.5 cm范围内土壤,每株植物取上中下3层土壤混匀作为根际土壤[12],无植物生长的同土层土壤,随机取样3次混匀作为对照。样品分别装入无菌自封袋,放入车载冰箱4℃保存运回实验室。将土样除砂石及植物残体,混匀,过2 mm筛,分成两份,一份用于提取土壤基因组DNA,一份自然风干用于测定土壤化学性质。
1.2 土壤化学性质测定
土样主要化学性质依据土壤分析方法标准(DB65/T602)测定。pH值采用1∶5土水比电位测定法;有机质采用重铬酸钾氧化法测定;全氮采用凯氏法测定;速效氮采用碱解-扩散法测定;速效磷采用碳酸氢钠浸提-钼锑抗比色法测定;有效钾测定采用醋酸氨浸提-火焰光度法。每个土样重复3次。
1.3 土壤基因组DNA提取
称取5.0 g土壤样品,经脱腐处理[13],采用 bead beating-SDS结合法[14-15]提取土壤总 DNA,用0.8%的低熔点琼脂糖凝胶电泳检测,OMEGA E.Z.N.A.TM Gel Extraction Kit回收纯化。总DNA溶于TE溶液并于-20℃储存。
1.4 土壤古菌PCR扩增
选用古菌16SrDNA通用引物(21F:5'-TTCCGGTTGATCCYGCCGGA-3',5'端用TET荧光标记的958R:5'-YCCGGCGTTGAVTCCAATT-3',)进行扩增。PCR 扩增反应体系:premix 15 μL,10 μmol/L 引物各 0.15 μL,DNA 模板 0.5 μL(约 10 ng),0.1%BSA 7.5 μL,25 mmol/L MgCl21 μL,用无菌 ddH2O 补足 30 μL 体积。PCR条件采用Touchdown扩增:95℃预变性4 min,94℃变性30 s,59℃至54℃退火40 s,72℃延伸90 s,每次循环降低0.5℃,10次循环,94℃变性30 s,54℃退火40 s,72℃延伸90 s,25次循环,72℃延伸7 min。每个样品做3个重复。1%琼脂糖电泳检测。为避免PCR技术本身带来的偏差,并保证同等质量和数量的PCR产物进行酶切消化,参考Zhang[16]等人的方法,将3个重复PCR产物合并,用回收试剂盒OMEGA E.Z.N.A.TM Gel Extraction Kit纯化。
1.5 T-RFLP 分析
将纯化的PCR产物均分2份(各13.2 μL)分别用6 U限制性内切酶HhaⅠ、RsaⅠ进行酶切,37℃反应3 h,65℃水浴20 min终止反应。2.5%琼脂糖电泳检测。将酶切产物送至上海基康生物技术有限公司,进行毛细管电泳并使用DNA自动测序仪(ABI 3730 DNA Analyzer)检测。
1.6 数据处理
选取片段长度在30—700 bp的T-RFs(Terminal Restriction Fragments),用公式Ap=ni·N-1×100计算相对峰面积。为避免电泳噪声对数据分析的干扰,本文将大于单个样品总面积0.5%的峰作为有效信号,并将片段大小相差±0.5 bp的T-RFs认为是相同的。分别计算物种丰度S,即图谱中显著峰的总数,多样性指数Shannon-Weiner指数 H,Simpson指数 D,物种均度 E[17]。公式如下:

式中,Pi表示某个峰的峰高占总峰高的比例,Hmax=lnS。
根据Sorensen公式计算古菌群落间的相似性[18]:

式中,c是两个样品中共有的物种数量,a和b分别代表a样品和b样品中物种的数量。
将相对峰面积大于3%总面积的T-RFs作为群落中的优势类群,进行群落成分差异分析,并在MiCA(Microbial Community Analysis)网站(http://mica.ibest.uidaho.edu/)在线 RDP 数据库中进行 T-RFs的初步鉴定。用CANNOCO软件进行典范对应分析(CCA)分析。将优势类群各多样性指数与土壤化学性质参数做对数转换(pH值除外),用SPSS16.0进行相关分析。
2 结果与分析
2.1 塌陷区植物根际土壤及裸地土壤化学性质
方差分析显示,不同植物根际土壤化学性质差异显著(表1),根际土壤养分及pH值(pH=8.29—8.51)多高于对照。东方旱麦草的各项养分含量普遍较高,西北绢蒿的根际土壤有机质、全氮、速效氮含量较高,速效磷含量较低。
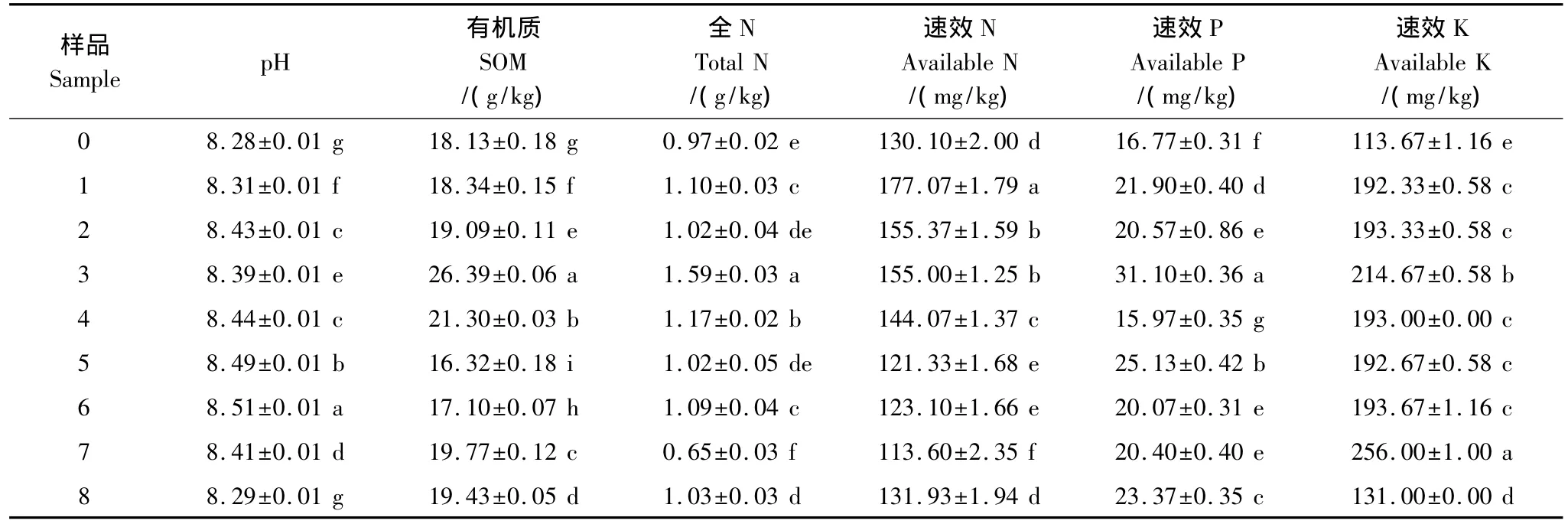
表1 不同植物根际土壤化学性质Table 1 Chemical properties of the rhizospheric soils in different plants
2.2 塌陷区土壤古菌16S rDNA扩增
以纯化后的DNA为模板,PCR扩增古菌16S rDNA基因,琼脂糖凝胶电泳检测,只有植物根际土壤扩增出约900 bp目的片段(图1),对照土壤未扩增出目的条带。
2.3 塌陷区根际土壤古菌群落的T-RFLP分析
2.3.1 根际土壤古菌群落的多样性
用RsaⅠ、HhaⅠ分别酶切PCR产物的结果并不完全一致(表2)。以RsaⅠ酶切,欧亚绣线菊和密刺蔷薇根际古菌丰度和多样性最高,鼠掌老鹳草和欧亚绣线菊的优势度和均匀度较高;以HhaⅠ酶切,东方旱麦草和鼠掌老鹳草古菌多样性指数较高。两种酶切,蓬子菜古菌丰度和多样性均最低。整体来看,RsaⅠ能得到较多的T-RFs。
2.3.2 根际土壤古菌群落的相似性

图1 古菌16S rDNA片段电泳图Fig.1 The fragments of archaeal 16S rDNA
不同植物根际土壤间古菌群落组成具有较大差异(表3)。以RsaⅠ酶切,8种植物根际土壤古菌群落组成相似性较低,在0.04—0.33之间;以HhaⅠ酶切,密刺蔷薇和鼠掌老鹳草,西北绢蒿根际土壤古菌群落相似性较高,分别达0.67和0.62,其它样品间相似性普遍较低。

表2 不同植物根际古菌群落的多样性指数Table 2 Diversity indices of archaeal communities in different rhizosphere soils

表3 不同植物根际古菌群落的相似性Table 3 Similarity of archaeal communities in different rhizosphere soils
2.3.3 根际土壤古菌群落的优势类群组成
参考Zhang[16]等人的方法,采用获T-RFs数目较多的RsaⅠ酶切结果,将相对峰面积大于3%的T-RFs作为优势类群。8种植物根际土壤古菌优势类群数量差异较大(图2),鼠掌老鹳草多达18种,直穗柳次为9种,西北绢蒿少为2种。81 bp的T-RF(可能是泉古菌门(crenarchaeota))在8种植物根际土壤中均出现,所占比例有差异,在皱纹柳根际最高占45.22%,鼠掌老鹳草根际最小,为10.38%;83 bp的T-RF(可能是广古菌门(Euryarchaeota)的盐杆菌属(Halobacterium)或Thaumarchaeota的餐古菌属(Cenarchaeum))存在于5种植物根际土壤中,蓬子菜根际所占比例最大,为68.63%,其余片段的T-RFs多为某些植物所特有。
在MiCA数据库比对2种酶切的T-RFs,确定其可能代表的种属,结果差异较大。以HhaⅠ酶切(图3),主要古菌类群为泉古菌门(45.45%)和广古菌门(27.27%);以RsaⅠ酶切(图3),未匹配的T-RFs所占比例较大(71.11%),其次为泉古菌门(15.56%)、广古菌门(8.89%)及纳古菌门(2.22%)。
2.3.4 根际古菌优势类群与土壤化学性质的典范对应分析

图2 古菌群落的优势类群组成Fig.2 Composition of dominant group of archaeal communities

图3 基于MiCA检索可能的古菌类群Fig.3 Possible archaeal groups searched from MiCA
将HhaⅠ酶切的T-RFs与土壤化学性质进行CCA分析,根据矢量线段的长短,表明土壤有机质、速效磷及总氮对优势类群的分布影响最大(图4a),它们与第一轴的相关系数分别为0.94(P<0.001),0.81(P<0.01),0.76(P<0.05)。在速效磷含量较高的土壤中,分布着 48 bp及 112 bp等 TRFs,较低处为 233 bp、587 bp等TRFs;pH较高处,为29 bp、435 bp等TRFs;585 bp的TRF受6种土壤化学性质影响最小。
RsaⅠ酶切的T-RFs与土壤化学性质的CCA分析表明,土壤有机质、pH值对优势类群的分布影响最大(图4b),土壤有机质与第一轴的相关系数为0.74(P<0.05),pH值与第二轴的相关系数为-0.50(P>0.05)。在土壤有机质、速效氮含量及pH值较高处,分别有83 bp及46 bp、112 bp及320 bp、49 bp及503 bp等TRFs存在;30 bp及204 bp等TRFs受6种土壤化学性质影响最小。
2.3.5 古菌群落多样性指数与土壤化学性质的相关分析
在HhaⅠ酶切结果中(表4),土壤速效磷含量与古菌群落关系最为密切,与物种丰度、多样性指数、优势度和均匀度均呈显著相关性(P<0.05),此外群落均匀度指数E还与土壤总氮含量呈显著相关性(P<0.05)。在RsaⅠ酶切结果中,古菌群落各多样性指数与土壤化学性质无相关性(P>0.05),此处未列出。
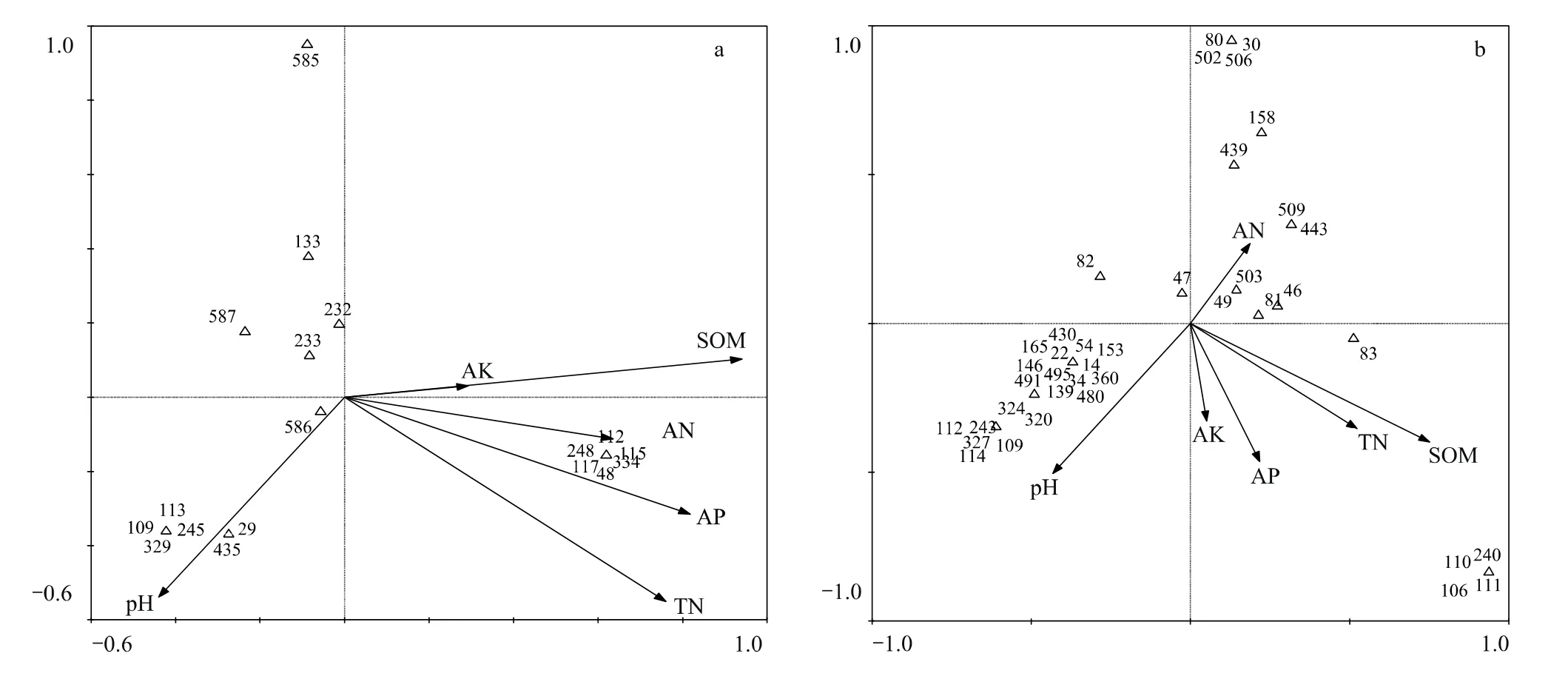
图4 基于T-RFLP结果的CCA分析图Fig.4 CCA plots based on T-RFLP results
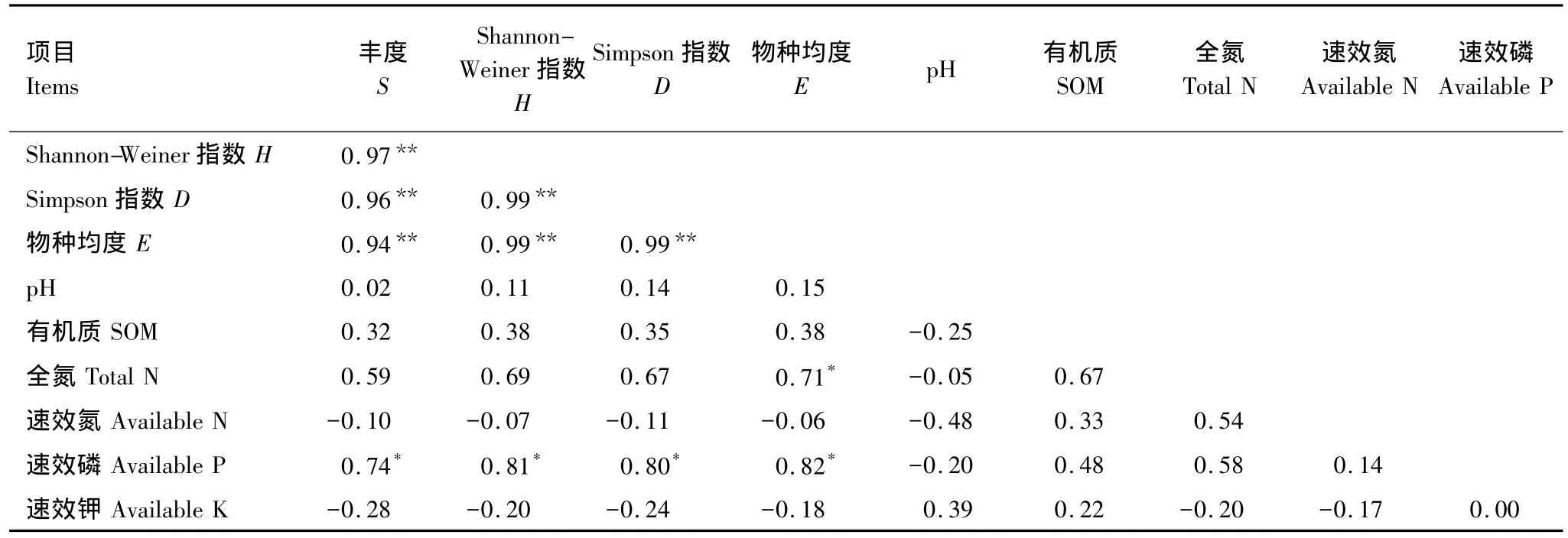
表4 古菌群落多样性指数与土壤化学性质的相关分析Table 4 The correlation coefficients of archaeal diversity indices and soil chemical properties
3 讨论
3.1 T-RFLP技术可行性分析
T-RFLP技术因其相对简便、高通量、能与在线数据库结合分析微生物种类等特点已广泛应用于评估多种生境中微生物群落结构及动态变化的分析[19]。由于在一个特定的引物-酶组合下,一个T-RF可能代表不止一种细菌种群,为减少此类误差常采用多酶切处理,但引物一定时,选用不同内切酶产生的T-RFs数目有显著差别,相应的分析结果也存在一定差异[20],因此,一些学者选取产生最多T-RFs数目的酶切结果作为后续分析的数据,而其它酶切作为辅助参考,进行综合判断[16,21]。本文选取RsaⅠ和HhaⅠ两种常用限制性酶,产生结果存在一定差异,但二者的共同趋势都显示,不同植物根际的古菌群落差异大,古菌优势种群以泉古菌门和广古菌门为主,其分布受土壤有机质影响最大,说明该技术能基本准确的分析研究样地的古菌群落结构及其变化。在RsaⅠ的酶切结果中,与数据库未匹配的T-RFs占有很大比例(71.11%),这些未匹配的T-RFs可能是由于RDP数据库一些16S rRNA基因序列不完整,或信息不完善等本身的缺陷造成,但也不排除该地区有存在大量古菌新种的可能[22]。
3.2 植被恢复对土壤古菌群落分布的影响
本文在植物根际土壤扩增出古菌16S rDNA片段,而同土层裸地土壤并未检出。Jackson等人在森林泥炭土的表层土壤(0—10 cm)未能检测到古菌[23]。这说明在古菌分布少或无的表层土壤,植物扩大了古菌的分布范围,提高其物种丰度。本研究中,次生植物显著改善了原本退化严重的土壤肥力,促进土壤发育[24];加之土壤中根系和通气状况的改善,使微生物区系有较大差异,如在柠条根际土壤中古菌数量较多[25],氨氧化泉古菌在水稻根际比非根际丰富[26]。另一方面,富蕴断裂带地处阿尔泰戈壁沙漠北部,受干旱气候影响[27],阳光直射造成的热压力和干燥能降低土壤养分的利用,裸地上出现的植被对地表温度和干燥有一定的缓冲作用,有利于土壤微生物的生长[28]。
3.3 植被恢复对土壤古菌群落结构的影响
塌陷区不同植物根际间古菌群落结构有很大差异。许多研究表明植物类型是决定土壤微生物群落结构的主要因子:一方面从植物物种角度,因植物根系分泌物、形态、及生理不同,其根围微生境会有所差异[29]。苗果园等人发现,即使在相同土壤条件种植的不同植物,其根际微生物种群和数量也有显著差异[30]。Haichar等人用稳定同位素标记方法证明,培育在相同条件下4种植物的类型及其根系分泌物能显著影响根际细菌群落的组成[31]。另一方面从生理生态学角度,植物演替至灌木阶段,处于草本、灌木丛生状态,在共存物种间植物会产生一定生态分化以适应环境[32],如受体积、光照、空气等影响,演替早、中期草本植物和灌木在光合速率、呼吸速率、水分和养分利用效率、生长速度等方面有明显的差异[33]。
本文根际土壤古菌优势类群为泉古菌门,Nicol等人对草地生态系统的研究,也有类似结果[34]。Simon等人在西红柿根系富集到泉古菌,并认为其可能参与根系生物代谢[35]。泉古菌在地球环境中大量存在,在土壤微生物总量比例最高达12%[36]。泉古菌门中的氨氧化古菌一直是全球氮循环微生物机理研究的前沿领域之一,研究表明氨氧化古菌是海洋生态系统氨氧化过程的重要驱动者,但在土壤中,细菌是氨氧化过程的主要驱动者,古菌的生态学功能还需进一步探索[37]。
3.4 土壤化学性质与古菌群落的关系
本文古菌群落的分布与多项指数与土壤有机质、速效磷、总氮含量呈显著正相关,这很可能是因为植物恢复产生了一定的凋落物,成为土壤有机质的主要来源,为微生物提供物质和能量[38]。研究表明随植物演替发展,在贫瘠土壤中氮素含量因微生物矿化作用越来越迅速,植物充分形成生物量而积累增加[33]。塔克拉玛干沙漠腹地人工绿地土壤中,土壤速效磷、速效氮、全氮对微生物的分布有较大作用[39];百色与胜利油田土壤总氮、速效氮、速效磷对微生物群落代谢活性及多样性呈正相关,并有一定的促进作用[40];奥地利Rotmoosferner冰川前缘土壤古菌群落呈现出清晰的演替现象,泉古菌Group1.1b出现在所有演替阶段,而Group1.1c只存在于演替后期成熟土壤,土壤有机碳、总氮及速效磷的含量随土壤的发育逐渐升高,这些成熟的土壤因素可能是 Group1.1c 泉古菌定殖所必需[41]。
4 展望
植被恢复一直被视为震后生态系统重建的核心,退化生态系统植被恢复的首要任务是选择合适的建群植物种类,以保证系统能迅速地朝良性方向发展[42]。本文研究表明,东方旱麦草在提高根际土壤古菌多样性及改善土壤养分方面均发挥了显著作用,为灾后重建工作提供了科学依据和参考。植物-土壤-微生物是陆地生态系统的重要组成部分,三者间的相互作用是生态系统地上、地下部分结合的重要纽带,本文初步调查了新疆地震断裂带次生植物根际古菌群落结构和土壤化学性质,发现其间存在一定联系,但古菌群落在研究样地的生态功能、是否与其它环境因子相关及如何推动植物演替的发展有待进一步探索。
[1] Liu G Q,Jiao X.Ecological issues caused by Wenchuan earthquake and countermeasures.Soil and Water Conservation in China,2008,(11):11-13.
[2] Pan J X,Zhang Y.Selection and application of plants in ecosystems reconstruction after the earthquake.Journal of Institute of Disaster-Prevention Science and Technology,2009,11(3):9-13.
[3] Shi J B,Ding G Y.Preface//Seismological Bureau of Xinjiang Uygur Autonomous Region eds.The Fuyun Seismic Fault Zone.Beijing:Seismological Press,1985:1-5.
[4] Peng S L.Restoration ecology and vegetation reconstruction.Ecologic Science,1996,15(2):26-31.
[5] Artz R R E,Chapman S J,Siegenthaler A,Mitchell E A D E,Buttler A,Bortoluzzi E,Gilbert D,Ylipetays M,Vasander H,Francez A J.Functional microbial diversity in regenerating cutover peatlands responds to vegetation succession.Journal of Applied Ecology,2008,45(6):1799-1809.
[6] He X Y,Wang K L,Zhang W,Chen Z H,Zhu Y G,Chen H S.Positive correlation between soil bacterial metabolic and plant species diversity and bacterial and fungal diversity in a vegetation succession on Karst.Plant and Soil,2008,307(1/2):123-134.
[7] Zhang S Y,Yu M Q,Zhu L,Qin X L.A study on microbial biomass C,N characteristics in different rehabilitating forests on degraded red soil.Acta Agriculturae Universitatis Jiangxiensis:Natural Sciences Edition,2010,32(1):101-107.
[8] Clay K,Holah J.Fungal endophyte symbiosis and plant diversity in successional fields.Science,1999,285(5434):1742-1744.
[9] Yang H L,Liu D M,Sun H.Ecological impacts of earthquake on local ecosystem and its restoration.Sichuan Environment,2009,28(4):97-101.
[10] He J Z,Shen J P,Zhang L M.Advance in the research of soil non-thermophilic Crenarchaeota.Acta Ecologica Sinica,2009,29(9):5047-5055.
[11] He S H.1716—1980 seismic data of Xinjiang//Seismological Bureau of Xinjiang Uygur Autonomous Region,eds.Compilation of Earthquake Materials of Xinjiang Uygur Autonomous Region.Beijing:Seismological Press,1985:5-132.
[12] Nicol G W,Glover L A,Prosser J I.Spatial analysis of archaeal community structure in grassland soil.Applied and Environmental Microbiology,2003,69(12):7420-7429.
[13] Xi F,Fu L Y,Wang G Z,Zheng T L.A simple method for removing humic acids from marine sediment samples prior to DNA extraction.Chinese High Technology Letters,2006,16(5):539-544.
[14] Zhou J,Bruns M A,Tiedje J M.DNA recovery from soils of diverse composition.Applied and Environmental Microbiology,1996,62(2):316-322.
[15] Zhang R F,Cao H,Cui Z L,Li S P,Fan B.Extraction and purification of soil microbial total DNA.Acta Microbiologica Sinica,2003,43(2):276-282.
[16] Zhang R,Thiyagarajan V,Qian PY.Evaluation of terminal-restriction fragment length polymorphism analysis in contrasting marine environments.FEMS Microbiology Ecology,2008,65(1):169-178.
[17] Li H.Progress of T-RFLP analysis for microbial community.Journal of Anhui Normal University:Natural Science,2006,29(6):582-585.
[18] Eiler A,Bertilsson S.Composition of freshwater bacterial communities associated with cyanobacterial blooms in four Swedish lakes.Environmental Microbiology,2004,6(12):1228-1243.
[19] Schütte U M E,Abdo Z,Bent S J,Shyu C,Williams C J,Pierson J D,Forney L J.Advances in the use of terminal restriction fragment length polymorphism(T-RFLP)analysis of 16S rRNA genes to characterize microbial communities.Applied Microbiology and Biotechnology,2008,80(3):365-380.
[20] Luna G M,Stumm K,Pusceddu A,Danovaro R.Archaeal diversity in deep-sea sediments estimated by means of different terminal-restriction fragment length polymorphisms(T-RFLP)protocols.Current Microbiology,2009,59(3):356-361.
[21] Székely A J,Sipos R,Berta B,Vajna B,Hajdú C,Márialigeti K.DGGE and T-RFLP Analysis of Bacterial Succession during Mushroom Compost Production and Sequence-aided T-RFLP Profile of Mature Compost.Microbial Ecology,2009,57(3):522-533.
[22] Yuan S Q,Xue Y F,Gao P,Wang W D,Ma Y H.Microbial diversity in Shengli petroleum reservoirs analyzed by T-RFLP.Acta Microbiologica Sinica,2007,47(2):290-294.
[23] Jackson C R,Liew K C,Yule C M.Structural and functional changes with depth in microbial communities in a tropical malaysian peat swamp forest.Microbial Ecology,2009,57(3):402-412.
[24] Huang C M,Gong Z T.Progress in quantitative study on soil genesis and development.Soils,2000,32(3):145-150,166-166.
[25] Zhang W,Hu Y G,Huang G H,Gao H W.Soil microbial diversity of artificial peashrub plantation on North Loess Plateau of China.Acta Microbiologica Sinica,2007,47(5):751-756.
[26] Chen X P,Zhu Y G,Xia Y,Shen J P,He J Z.Ammonia-oxidizing archaea:important players in paddy rhizosphere soil.Environmental Microbiology,2008,10(8):1978-1987.
[27] Lin A M,Lin S J.Tree damage and surface displacement:the 1931 M 8.0 Fuyun earthquake.The Journal of Geology,1998,106(6):751-757.
[28] Tscherkoa D,Hammesfahr U,Zeltner G,Kandeler E,Böcker R.Plant succession and rhizosphere microbial communities in a recently deglaciated alpine terrain.Basic and Applied Ecology,2005,6(4):367-383.
[29] Wu L Q,Ma K,Li Q,Ke X B,Lu Y H.Composition of archaeal community in a paddy field as affected by rice cultivar and N fertilizer.Microbial Ecology,2009,58(4):819-826.
[30] Miao G Y,Jia Z H,Yang Z P,Zhang Y Q.Quantity difference of rhizosphere microbe of different crops.Journal of Shanxi Agricultural University:Natural Science Edition,2004,24(2):93-96.
[31] Haichar F Z,Marol C,Berge O,Rangel-Castro J I,Prosser J I,Balesdent J,Heulin T,Achouak W.Plant host habitat and root exudates shape soil bacterial community structure.The ISME Journal,2008,2(12):1221-1230.
[32] Ackerly D.Functional strategies of chaparral shrubs in relation to seasonal water deficit and disturbance.Ecological Monographs,2004,74(1):25-44.
[33] Li Q K,Ma K P.Advances in plant succession ecophysiology.Acta Phytoecologica Sinica,2002,26(Z):9-19.
[34] Nicol G W,Glover L A,Prosser J I.Spatial analysis of archaeal community structure in grassland soil.Applied and Environmental Microbiology,2003,69(12):7420-7429.
[35] Simon H M,Dodsworth J A,Goodman R M.Crenarchaeota colonize terrestrial plant roots.Environmental Microbiology,2000,2(5):495-505.
[36] Roesch L F W,Fulthorpe R R,Riva A,Casella G,Hadwin A K M,Kent A D,Daroub S H,Camargo F A O,Farmerie W G,Triplett E W.Pyrosequencing enumerates and contrasts soil microbial diversity.Multidisciplinary Journal of Microbial Ecology,2007,1(4):283-290.
[37] Ji Z J,Weng J H,Lin X G,Conrad R.Microbial ecology of archaeal ammonia oxidation—A review.Acta Microbiologica Sinica,2010,50(4):431-437.
[38] Cao F Q,Liu Z H,Liu M,Cui J F.Research progress on the forest litterfall and its decomposition process.Guangxi Agricultural Sciences,2010,41(7):693-697.
[39] Zhou Z B,Li P J.Ecological distribution of soil microorganism in artificial greenbelt in hinterland of Takilimakan Desert and their relations with soil factor.Chinese Journal of Applied Ecology,2003,14(8):1246-1250.
[40] Zhang Y N,Liang Y T,Li G H.Studies on relationship between carbon metabolism of soil microbial community and soil physicochemical factors.China Environmental Science,2010,30(12):1639-1644.
[41] Nicol G W,Tscherko D,Embley T M,ProsserJ I.Primary succession of soil Crenarchaeota across a receding glacier foreland.Environmental Microbiology,2005,7(3):337-347.
[42] Zhao P.Advances in plant ecophysiological studies on re-vegetation of degraded ecosystems.Chinese Journal of Applied Ecology,2003,14(11):2031-2036.
参考文献:
[1] 刘广全,焦醒.汶川地震引发的生态问题及对策.中国水土保持,2008,(11):11-13.
[2] 潘静娴,张莹.地震灾后生态系统重建中的植被恢复初探.防灾科技学院学报,2009,11(3):9-13.
[3] 石鉴帮,丁国瑜.前言 //新疆维吾尔自治区地震局.富蕴地震断裂带.北京:地震出版社,1985:1-5.
[4] 彭少麟.恢复生态学与植被重建.生态科学,1996,15(2):26-31.
[7] 张水印,余明泉,朱玲,秦晓蕾.退化红壤区不同重建模式森林土壤微生物碳氮特征研究.江西农业大学学报,2010,32(1):101-107.
[9] 杨红露,刘冬梅,孙辉.地震的生态破坏及其恢复重建研究进展.四川环境,2009,28(4):97-101.
[10] 贺纪正,沈菊培,张丽梅.土壤中温泉古菌研究进展.生态学报,2009,29(9):5047-5055.
[11] 新疆维吾尔自治区地震局.1716—1980年新疆地区地震资料 //新疆维吾尔自治区地震局.新疆维吾尔自治区地震资料汇编.北京:地震出版社,1985:5-132.
[13] 席峰,傅莲英,王桂忠,郑天凌.海洋沉积物DNA提取前的简易脱腐方法研究.高技术通讯,2006,16(5):539-544.
[15] 张瑞福,曹慧,崔中利,李顺鹏,樊奔.土壤微生物总DNA的提取和纯化.微生物学报,2003,43(2):276-282.
[17] 李红.末端限制性酶切片段长度多态性分析技术进展.安徽师范大学学报:自然科学版,2006,29(6):582-585.
[22] 袁三青,薛燕芬,高鹏,汪卫东,马延和.T-RFLP技术分析油藏微生物多样性.微生物学报,2007,47(2):290-294.
[24] 黄成敏,龚子同.土壤发生和发育过程定量研究进展.土壤,2000,32(3):145-150,166-166.
[25] 张薇,胡跃高,黄国和,高洪文.西北黄土高原柠条种植区土壤微生物多样性分析.微生物学报,2007,47(5):751-756.
[30] 苗果园,贾志红,杨珍平,张永清.不同作物根际微生物差异的研究.山西农业大学学报:自然科学版,2004,24(2):93-96.
[33] 李庆康,马克平.植物群落演替过程中植物生理生态学特性及其主要环境因子的变化.植物生态学报,2002,26(增刊):9-19.
[38] 曹富强,刘朝晖,刘敏,崔俊峰.森林凋落物及其分解过程的研究进展.广西农业科学,2010,41(7):693-697.
[39] 周智彬,李培军.塔克拉玛干沙漠腹地人工绿地土壤中微生物的生态分布及其与土壤因子间的关系.应用生态学报,2003,14(8):1246-1250.
[42] 赵平.退化生态系统植被恢复的生理生态学研究进展.应用生态学报,2003,14(11):2031-2036.
