纳米二次离子质谱技术(NanoSIMS)在微生物生态学研究中的应用
2013-09-11胡行伟张丽梅贺纪正
胡行伟,张丽梅,贺纪正,*
(1.中国科学院生态环境研究中心,城市与区域生态国家重点实验室,北京 100085;2.中国科学院研究生院,北京 100049)
氮(N)、碳(C)、硫(S)等生命元素的生物地球化学循环过程主要由微生物所驱动。耦合分析自然环境中微生物遗传多样性与其代谢多样性是当今微生物生态学研究的难点和热点。自然环境中的微生物多样性极为丰富,每吨土壤中的微生物类群可高达400万种,海洋中的微生物类群也超过200万种[1]。实验室富集分离的传统纯培养方法对微生物生理生态功能的认识做出了巨大贡献,但截止到2003年7月,全球范围内仅分离培养出4800种细菌[2],这极大地限制了人们对微生物功能多样性的认识。过去几十年里,人们通过从环境样品中提取微生物DNA直接进行聚合酶链式反应(PCR),并利用PCR产物构建克隆文库并进行测序分析,发现了大量的未知序列。通过将这些未知序列与已知的分离培养微生物序列进行比对,从而推测这些未培养微生物的可能代谢类型,成为人们认识微生物多样性和功能的重要手段。但是,具有相似16S rRNA基因组成的类群不一定具有相同的代谢功能[3-4],丰度较高的微生物并不一定发挥更重要的生态功能[5],而且PCR反应可能带来一定的偏差,这成为基于PCR的分子生物学研究手段的主要缺陷。考虑到微生物的广泛分布及其在元素地球化学循环中的重要作用,需要更先进的技术对自然环境样品中的微生物群落和功能进行探测。
近年逐步兴起的纳米二次离子质谱技术(NanoSIMS)在国际上已被广泛应用于地球科学、材料科学、比较行星学、生物医学和矿物学等领域,并在微生物生态学研究中显示出巨大的潜力,是当前最为先进的表面和界面分析技术,具有广阔的应用前景。通过利用稳定性或放射性同位素在原位或者微宇宙条件下标记目标微生物,并与特异性的荧光原位杂交技术(FISH)、催化报告沉积荧光原位杂交技术(CARD-FISH)、卤素原位杂交技术(HISH)等联合应用,NanoSIMS为从单细胞成像水平上研究复杂环境样品中微生物群落的组成和代谢特征提供了前所未有的可能性,其极高的灵敏度和准确性比其它的单细胞研究手段更具优势。本文将对纳米二次离子质谱的工作原理及其在微生物生态学研究领域的相关应用、存在的问题和发展趋势等进行简要介绍。
1 纳米二次离子质谱技术(NanoSIMS)的原理
二次离子质谱技术(SIMS)是20世纪70年代发展起来的一种表面分析技术,它以一种离子(如Cs+或O-初级离子束)轰击固体表面,再将从表面溅射出来的次级离子引入磁场质量分析器,不同离子根据质荷比不同在静电场区被分离开,经质谱检测器检测记录并成像,得出被分析样品表面的元素或化合物的组分。SIMS可以检测从氢到铀的所有元素、同位素和化合物,并以检测原离子轰击样品表面产生的特征(“指纹”)次级离子谱为基础的,因此既可提供样品表面元素的信息,也可提供化学组分的信息,具有较高的空间分辨率和质量分辨率。SIMS技术经历了几次更新,被广泛应用于材料学、生物医学、地质学和土壤学等领域。SIMS一般分为静态SIMS和动态SIMS两种[6]。静态SIMS,例如飞行时间二次离子质谱(ToF-SIMS),根据测定激发的二次离子飞行到接收器所用的时间来判断离子种类,一般对样品表面的一个到两个原子层(<1 nm)进行分析,只能提供关于样品浅层表面的信息,在应用于生态学研究时很难找到合适的条件同时满足较高质量分辨率和空间分辨率(一般只能达到微米级)的要求[7];而动态SIMS如CAMECA IMS 1270-180型、CAMECA IMS-3f-7f型、及最先进的CAMECA NanoSIMS 50L型,主要根据二次离子在磁场中的偏转半径不同来确定离子种类,可以获得样品表面几微米内元素同位素分布和组成的信息(空间分辨率达到亚微米级或者纳米级)[8],成像水平比静态SIMS明显提高,在微生物生态学中的应用也日渐成熟。
法国CAMECA公司生产的NanoSIMS 50L型是当前最为先进的动态二次离子质谱[9],相比于传统的离子探针具有极高的灵敏度和空间分辨率,其中铯(Cs+)离子源的分辨率达到50 nm,氧(O-)离子源的分辨率达到150 nm,并且具有极高的质量分辨率,如能够有效区分13C-与12C1H-、12C15N-与13C14N-之间的细微质量差异,并且能够同时对7种离子进行成像分析和精确定量[10]。CAMECA NanoSIMS 50L与传统的二次离子质谱仪的工作原理非常相似,都是通过用一次离子束轰击的方式激发样品表面产生二次离子,进而分析样品表面元素同位素组成和丰度[11]。具体工作流程为:首先将离子源(Cs+或者O-)产生的一次离子在真空加速器中加速形成具有几千电子伏特(KeV)的离子束,当这些高能量的离子束聚焦并轰击固体薄片样品表面(微生物细胞)时,溅射出样品中的二次离子,这些具有不同荷质比的二次离子由于在磁场中偏转半径不同而到达多接收器的不同位置,然后通过接收器上的法拉第环或者电子倍增器测量不同离子的强度,进而成像并通过图像定量分析软件计算出样品表面的元素同位素的丰度和组成信息(图 1)。测定的同位素比值信息(例如13C-/12C-)不仅可以用于计算单细胞的元素吸收速率,还可以表征元素的代谢途径[12]。选择合适的离子源对于正确分析样品信息极为关键,铯(Cs+)离子源激发的负二次离子可以用来分析C、CN、P、S、O、H和卤族元素等,一般在微生物生态学研究中被广泛采用,而氧(O-)离子源激发的正二次离子适合于进行金属的表面分析[13]。
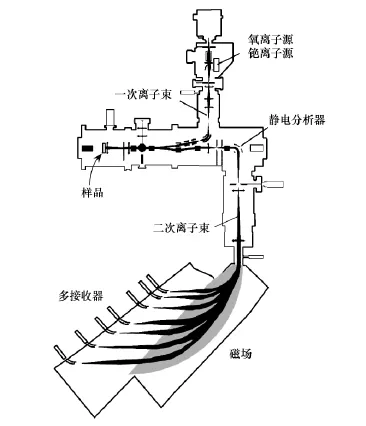
图1 CAMECA NanoSIMS 50L型纳米二次离子质谱仪工作原理示意图[11]Fig.1 Schematic illustration of CAMECA NanoSIMS 50L ion microprobe[11]
2 纳米二次离子质谱技术(NanoSIMS)应用于微生物生态学研究的技术路线
二次离子质谱技术(SIMS)在微生物生态学中的应用始于2001年。Orphan等首次利用FISH与SIMS结合的方法证实了海洋沉积物中存在厌氧甲烷氧化古菌,能够代谢13C—CH4[14]。但当时对同位素组成分析和FISH显影分析需在不同的仪器上完成,而且SIMS的横向分辨率较低,不能有效地对微生物的细胞进行辨识[15-16],最新的CAMECA NanoSIMS 50L型二次离子质谱仪有效克服了这一难题。应用NanoSIMS进行单细胞成像分析的主要步骤为:在实验室微宇宙培养或原位条件下,将采集的环境样品或可培养的微生物暴露在富含稳定性同位素(15N或13C等)或者放射性同位素基质的环境中,经过短期的同位素标记过程之后,将样品进行固定、脱水,形状不规则样品需要用环氧树脂包埋,绝缘的样品(绝大多数微生物生态学研究样品)需要进行导电镀膜(Au,Pt,Au/Pd或者 C)处理[17],制备成表面平整、符合仪器真空条件的薄片样品进行NanoSIMS分析。为了鉴别环境样品中的目标微生物,NanoSIMS需要与透射电子显微镜(TEM)、扫描电子显微镜(SEM)、荧光原位杂交(FISH)、催化报告沉积荧光原位杂交(CARD-FISH)、卤素原位杂交(HISH)、X射线能谱仪(EDS X-ray)等联合使用来识别微生物的种类和功能(图2)。
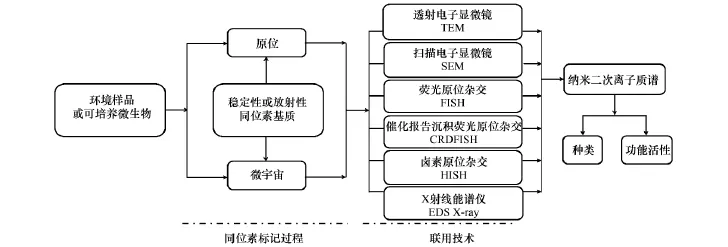
图2 纳米二次离子质谱分析微生物代谢功能活性的技术路线图Fig.2 Schematic diagram of NanoSIMS to detect and image microbial metabolic activities
3 纳米二次离子质谱技术(NanoSIMS)的应用实例
3.1 氮循环微生物
氮的生物地球化学循环主要由一系列微生物所驱动,识别并确定参与不同氮转化途径(包括固氮作用、硝化作用、反硝化作用和氨化作用)的功能微生物是当前氮循环机制研究的核心问题之一[18-19]。NanoSIMS技术在氮循环过程方面的应用首先在固氮微生物的研究上取得进展(表1)。2006年,Lechene等首次利用NanoSIMS从单细胞水平上研究并确定了实验室纯培养的固氮菌株Teredinibacter turnerae的固氮特性[20]。2007年,Lechene等又对与一种名为Lyrodus pedicellatus的船蛆(Shipworm)体内共生的固氮细菌进行了定量研究[21]。由于船蛆本身的食物链中不包含氮元素,所以必须依靠与其共生的微生物提供氮源。Lechene等在实验室培养条件下向含有船蛆Lyrodus pedicellatus的海水中注入稳定性同位素底物15N2,然后经过8d的暴露培养之后,先对样品进行透射电子显微镜(TEM)扫描确定待研究的船蛆组织中共生固氮细菌的位置,通过NanoSIMS分析后发现固氮细菌及周围组织均被15N所标记上,这直观有力地提供了船蛆依赖固氮微生物获取氮源的证据[21]。这些早期利用NanoSIMS技术研究固氮微生物细胞功能的例子显示出单细胞成像技术的巨大应用潜力。
固氮微生物中另一类被广泛研究的是丝状蓝藻,它们能够同时固定N2和CO2,其较大的细胞个体与独特的形态特征为NanoSIMS的应用提供了便利[22]。丝状蓝藻包括营养细胞和异型胞,但是过去由于研究手段的限制,对氮元素和碳元素在这些细胞内及细胞之间的转运和流动并不清楚。Popa等通过结合高分辨率的NanoSIMS与稳定性同位素标记技术对丝状固氮蓝藻A.oscillarioides的研究发现,新固定的氮元素可以被异型胞快速转运,并最终分配到营养细胞中去。此外,NanoSIMS还可以根据细胞组织不同的元素组成从亚细胞水平上进一步观察丝状蓝藻A.oscillarioides体内的元素分布状况[23]。另一个类似的通过NanoSIMS和TEM对固氮蓝藻菌株Trichodesmium的研究发现了更为细致的13C和15N吸收的时空分布特征,其中CO2的固定速率在上午达到最高水平,而N2的固定速率则在下午达到最高水平,并且新固定的C和N主要贮存在藻青素颗粒中[24]。这些研究说明,将同位素标记技术与NanoSIMS和TEM成像技术联合使用,能够在探究微生物细胞的生理组成元素及其相关代谢和转运过程方面发挥巨大作用。在最近的研究中它们用于分析两种不同的丝状蓝藻Nodularia spumigena和Aphanizomenon sp.对于波罗的海(Baltic Sea)碳氮循环的重要性,同时结合微传感器技术测定了两种不同蓝藻类群单细胞水平的氮固定和碳固定速率,从而能够更加准确地估测不同碳氮微生物对全球碳氮循环的贡献率[25-26]。
除了对实验室纯培养的固氮蓝藻进行分析之外,NanoSIMS同样也能够用于分析环境样品中的固氮微生物。最近,Foster等利用15N2同位素标记与NanoSIMS结合的方法首次对寡营养海水中的3种硅藻-蓝藻共生体的单细胞固氮速率进行了测定,解决了过去由于技术手段限制不能够将硅藻-蓝藻共生体与其它固氮菌分离研究的难题[27]。Halm等通过FISH与NanoSIMS联合使用,识别出在湖水厌氧层中发挥固氮作用的微生物主要是数量非常少的绿硫细菌Chlorobium sp.[28]。在更为复杂的河流沉积物样品中,Dekas等运用这2项技术对具有甲烷厌氧氧化功能的古菌ANME-2和细菌DSS类群的聚合体进行了研究,在经过6个月的15N2同位素培养实验之后,根据15N的吸收分布情况,发现这种聚合体中的ANME-2古菌具有固定氮气的功能,并且能够将固定的氮元素转运给DSS细菌[29-30]。
近年来,随着第一株具有氨氧化功能的古菌在实验室获得纯培养,氨氧化古菌(AOA)作为除细菌以外硝化作用的主要参与者,成为当前微生物生态学研究的热点之一[31]。奥地利维也纳大学的Tourna等成功地从土壤中富集到了氨氧化古菌Nitrososphaera viennensis,该菌株只有在添加有低浓度丙酮酸的培养基中才能将转化成为了验证该菌是否利用丙酮酸中的C元素来合成细胞组织,他们在含有N.viennensis的培养基中加入13C标记的丙酮酸,在37°C培养10 d后运用NanoSIMS对样品进行分析,发现大约有10%的C元素来自于13C-丙酮酸,其余的主要来自于对碳酸氢盐的固定,证明N.viennensis具有化能自养和混合营养生长的潜能[32]。该项研究是首次将NanoSIMS技术用于氨氧化微生物,对于探索新发现的氨氧化古菌的生理代谢机制提供了新的思路。
3.2 碳循环微生物
近些年发展起来的卤素原位杂交(HISH)与NanoSIMS联用的技术能够分析环境样品中的微生物组成,并同时定量测定单细胞的代谢功能[33]。Musat等首先将这种联用技术用于研究寡营养湖水中3种厌氧光合细菌 Chromatium okenii,Lamprocystis purpurea 和 Chlorobium clathratiforme对 H13和15的吸收特征[17]。他们通过实验室短期的微宇宙培养发现,同种光合细菌的不同细胞表现出极大的代谢速率差异,不同类群光合细胞之间的差异更大。其中,仅占细胞总数0.03%的C.okenii贡献了光合细菌碳吸收总量的70%和氮吸收总量的40%,这表明湖水中厌氧光合细菌的碳氮吸收过程主要是由一部分数量非常稀少的类群所完成的[17]。
NanoSIMS技术在碳循环微生物方面的另一类应用主要是对于甲基营养菌(包括甲醇营养菌和甲烷营养菌)的研究。Li等利用碘标记的寡核苷酸探针原位杂交与NanoSIMS结合的方法对生物反应器中甲醇营养菌的组成和功能进行了分析[34]。为了研究生物反应器系统中微生物吸收甲醇的机理,他们采用13C-甲醇培养采自生物反应器的样品,厌氧培养25 d后,首先用以碘标记的针对细菌和古菌的特异性探针确定样品里的微生物类群,然后将样品固定用于NanoSIMS分析,结果发现古菌是生物反应器中吸收甲醇的主要功能微生物[34]。此外,Behrens等运用类似的方法发现了口腔生物膜上的Cytophaga-Flavobacterium类群吸收13C-氨基酸的功能[35]。
3.3 硫循环微生物
硫酸盐还原菌(SRB)是硫元素生物地球化学循环过程的关键功能类群,经典的分子生物学研究手段已经对SRB的种群组成和丰度进行了深入研究,但尚不能在单细胞水平解析其生理代谢功能特征[36]。近期,美国研究者Fike等利用最新的NanoSIMS 50L型二次离子质谱仪,并结合CARD-FISH技术,成功地为解决这一问题提供了新的视角,识别出在高盐环境下微生物垫中参与硫循环的主要微生物类群是Desulfobacteraceae科的硫酸盐还原菌[37]。他们首先用外层包裹有35SO4的银箔与微生物垫中可溶性的硫化物反应,由于硫化物与35SO4的氧化还原反应使硫化物中的34S固定在银箔表面,然后通过NanoSIMS成像技术就能够测定样品中δ34S同位素的组成情况,从而计算出不同微生物垫深度上硫化物的浓度。NanoSIMS所生成的二维图像表明,微生物垫中硫化物的浓度随着深度的增加而减少,这种变化趋势与利用CARD-FISH所测定的Desulfobacteraceae科的硫酸盐还原菌丰度随微生物垫深度的变化趋势相吻合,说明Desulfobacteraceae科对微生物垫中硫化物的空间分布具有决定作用[37]。以往对于硫酸盐还原菌的研究多停留在对其总的丰度和结构组成分布上,而通过使用NanoSIMS与CARD-FISH结合的方法能够将微生物的空间分布与硫化物的空间分布联系在一起,更容易识别硫酸盐还原过程中的关键活跃类群。
3.4 其它方面的应用
NanoSIMS技术除了在氮、碳、硫等元素的生物地球化学循环研究中得到广泛应用外,研究者们也在不断拓展着NanoSIMS在微生物生态学方面的应用范围。微生物的空间分布及其在土壤基质中的功能活性对于元素地球化学循环具有极为重要的影响,而且土壤的高度异质性为微生物提供了无数性质各异的微环境[38],将土壤理化环境的异质性与其对生物过程的影响联系起来是当今土壤微生物生态学的研究前沿之一[39],然而过去的技术手段对于分析微生物与土壤基质接触的生物物理界面无能为力。2007年,Herrmann等创新地将NanoSIMS运用于土壤微生物的活性与空间分布的研究,并同时观察了土壤物理微环境对于微生物的影响[40]。他们首先将土壤中广泛分布的 Pseudomonas fluorescens的纯培养菌株(NCTC10038)在含有15的培养基中进行标记24 h后,混入土壤样品中,然后用环氧树脂固定并切片供NanoSIMS观察。其中,28Si-离子成像图用来表示土壤基质的组成,12C-成像图表示树脂的组成,12C14N-成像图表示含氮有机质,12C15N-与12C14N-的比值(即15N/14N)可以表示15N-P.fluorescens的分布(图3)。从NanoSIMS成像图中,可以清晰地看到15N标记的P.fluorescens主要分布在土壤粒子的边缘部分[40]。该项研究显示出NanoSIMS在分析微生物在土壤中的微域分布及其与土壤基质相互作用方面的巨大可能性。类似的研究方法已经开始用于对土壤-微生物-植物相互作用的研究。Clode等利用NanoSIMS在原位状态下研究了15N元素在土壤根际圈的转运情况,并重点关注了植物细胞与根际微生物对N元素的相互竞争[41]。这种原位研究元素流动的高分辨率成像方法显示了NanoSIMS在分析复杂土壤环境中微生物过程的优越性,而以往的此类研究只能主要依靠简单的数学模型分析。NanoSIMS在微生物生态学方面的其它可能应用还包括:控制土壤矿物质表面磷肥固定的微生物机理,促进有机质在土壤中稳定化过程的微生物类群,土壤基质中微生物与特定矿物质的相互作用,土壤中目标微生物活跃类群的空间分布,影响微生物水平基因转移的微观尺度因素等[6]。

图3 15N标记的P.fluorescens在树脂包被的土壤基质中分布的NanoSIMS 成像图[40]Fig.3 NanoSIMS images of the distributions of15N-lableled P.fluorescens in Araldite resin-embedded soil matrix [40](a)28Si-;(b)12C-;(c)12C14N-;(d)15N/14N ratio
4 使用NanoSIMS技术应注意的问题
4.1 样品制备
样品制备是NanoSIMS分析中极为关键的环节,因为NanoSIMS一般在超高真空环境中进行操作,因此制备的环境样品必须充分脱水并且能够承受真空。样品脱水的方法一般包括化学固定液(如加入乙醇、甲醛或戊二醛等)[44]、低温脱水[45]、环氧树脂包埋等[24]。不同的脱水方法适用于不同样品的 NanoSIMS分析,如果在样品制备的过程中待分析的元素有可能发生转移和流动,最好使用低温脱水方法来固定样品的形态,例如在研究固氮微生物的碳氮吸收途径的时候[23-24]。对于形状不规则的样品最好用树脂进行包埋,然后切成薄片状以便进行NanoSIMS分析。甲醛固定液对于微生物细胞的蛋白质、脂质和核酸有非常有效的固定和脱水作用[46],但是在固定的过程中也有可能引入微生物细胞中的12C,从而使NanoSIMS的分析出现一定的偏差。尤其是当NanoSIMS与荧光杂交技术如FISH、CARD-FISH和HISH联用时,最好使用多聚甲醛对样品进行脱水处理[17]。Peteranderl和Lechene比较了低温脱水与化学固定对样品制备的不同影响,发现化学固定导致二次离子12C-和12C14N-的产率降低,并且化学固定液有可能洗脱低分子量的细胞组织[45]。而Ploug等对这2种
脱水方法的研究表明,化学固定法并没有显著地影响待测样品的元素组成[25]。

表1 纳米二次离子质谱在微生物生态学方面的应用举例Table 1 Examples of NanoSIMS applications in microbial ecology
4.2 微生物的识别
在NanoSIMS分析中,不同的二次离子能够表征样品的不同组成,因此如何正确地将离子成像信息与微生物类群联系起来显得非常关键。例如在固氮硅藻-蓝藻共生体的研究中就会遇到类似的难题,因为共生蓝藻细胞经常生长在硅藻的细胞壁与细胞膜之间,为有效区分这两种不同微生物对N的吸收带来较大困难,因此在NanoSIMS分析之前必须首先破坏去除硅藻的细胞壁,然后再进行下一步分析[27]。识别复杂环境样品中的微生物类群也可以通过在NanoSIMS分析之前首先进行FISH或者CARD-FISH分析,以此确定微生物的组成和种类,但是将荧光原位杂交的图像与NanoSIMS分析的图像正确地联系在一起也是非常艰难的工作。为了有效地对微生物进行识别,在NanoSIMS分析之前也最好辅之以TEM或者SEM等电子显微镜技术对样品进行必要的基本特征分析,首先确定好适合NanoSIMS分析的感兴趣区域,这样可以节省大量的时间。例如在分析富含有机质的沉积物样品中的微生物群落时,有机质浓度较高的区域往往也是微生物富集的区域,因此首先用电子显微镜确定有机质的分布区域,可以更加快速地找到目标微生物。
5 总结和展望
以NanoSIMS为代表的单细胞分析技术的兴起为研究微生物生态学提供了崭新的机遇,并且已经在参与氮、碳、硫等元素生物地球化学循环的微生物研究中显示出前所未有的优势和重要的应用前景。NanoSIMS不仅能够提供微生物的生理生态特征信息,而且能够确定并识别复杂环境样品中的代谢活跃的微生物细胞,并将其类群信息与功能联系起来,对于从微观尺度上识别不同微生物群落在元素循环中的作用和在生态系统中的功能具有重要意义[22]。然而,NanoSIMS技术本身依旧存在很多问题:(1)在进行NanoSIMS分析时,如果选择反映同位素富集的感兴趣区域不合适,可能会影响对微生物功能的正确分析;(2)在制备可供NanoSIMS分析的样品时,固定和脱水等过程可能会对微生物细胞的同位素组成造成一定的影响;(3)由于NanoSIMS一般与同位素标记技术联合使用,标记过程中微生物交叉取食和同位素被稀释的风险同样也会影响后续的NanoSIMS分析结果。(4)由于FISH技术应用于复杂土壤样品的难题,NanoSIMS与FISH联用仍然很难区分不同类群土壤微生物的功能活性。当前微生物生态学的研究技术多基于PCR扩增,对环境微生物的认识以易于被PCR扩增出的优势种群为主,对环境中丰度较低的稀有微生物种群认识严重不足,未来基于NanoSIMS的研究方法将有助于揭示自然环境丰度较低但发挥重要功能作用的微生物种群。同时,随着原位杂交技术的进一步发展,结合不断丰富的生物标志物,如脂类、DNA/RNA、蛋白质等,NanoSIMS将可能为研究微生物的类群和功能提供更为完整的信息。
[1] Curtis T P,Sloan W T,Scannell J W.Estimating prokaryotic diversity and its limits.Proceedings of the National Academy of Sciences of the United States of America,2002,99(16):10494-10499.
[2] Keller M,Zengler K.Tapping into microbial diversity.Nature Reviews Microbiology,2004,2(2):141-150.
[3] Acinas S G,Sarma-Rupavtarm R,Klepac-Ceraj V,Polz M F.PCR-induced sequence artifacts and bias:Insights from comparison of two 16S rRNA clone libraries constructed from the same sample.Applied and Environmental Microbiology,2005,71(12):8966-8969.
[4] Jaspers E,Overmann J.Ecological significance of microdiversity:identical 16S rRNA gene sequences can be found in bacteria with highly divergent genomes and ecophysiologies.Applied and Environmental Microbiology,2004,70(8):4831-4839.
[5] Polz M F,Cavanaugh C M.Bias in template-to-product ratios in multitemplate PCR.Applied and Environmental Microbiology,1998,64(10):3724-3730.
[6] Herrmann A M,Ritz K,Nunan N,Clode P L,Pett-Ridge J,Kilburn M R,Murphy D V,O'Donnell A G,Stockdale E A.Nano-scale secondary ion mass spectrometry-a new analytical tool in biogeochemistry and soil ecology:a review article.Soil Biology and Biochemistry,2007,39(8):1835-1850.
[7] Cliff J B,Gaspar D J,Bottomley P J,Myrold D D.Exploration of inorganic C and N assimilation by soil microbes with time-of-flight secondary ion mass spectrometry.Applied and Environmental Microbiology,2002,68(8):4067-4073.
[8] Adams F,van Vaeck L,Barrett R.Advanced analytical techniques:platform for nano materials science.Spectrochimica Acta Part B:Atomic Spectroscopy,2005,60(1):13-26.
[9] Guerquin-Kern J L,Wu T D,Quintana C,Croisy A.Progress in analytical imaging of the cell by dynamic secondary ion mass spectrometry(SIMS microscopy).Biochimica Et Biophysica Acta-General Subjects,2005,1724(3):228-238.
[10] Eybe T,Audinot J N,Bohn T,Guignard C,Migeon H N,Hoffmann L.NanoSIMS 50 elucidation of the natural element composition in structures of cyanobacteria and their exposure to halogen compounds.Journal of Applied Microbiology,2008,105(5):1502-1510.
[11] Kanbach G,Nittler L.Instruments for observations of radioactivities//Diehl R,Hartmann D H,Prantzos N,eds.Astronomy with Radioactivities.Heidelberg:Springer,2011:491-515.
[12] Kuypers M M M,Jørgensen B B.The future of single-cell environmental microbiology.Environmental Microbiology,2007,9(1):6-7.
[13] Boxer S G,Kraft M L,Weber P K.Advances in imaging secondary ion mass spectrometry for biological samples.Annual Review of Biophysics,2009,38:53-74.
[14] Orphan V J,House C H,Hinrichs K U,McKeegan K D,DeLong E F.Methane-consuming archaea revealed by directly coupled isotopic and phylogenetic analysis.Science,2001,293(5529):484-487.
[15] Pernthaler A,Amann R.Simultaneous fluorescence in situ hybridization of mRNA and rRNA in environmental bacteria.Applied and Environmental Microbiology,2004,70(9):5426-5433.
[16] DeRito C M,Pumphrey G M,Madsen E L.Use of field-based stable isotope probing to identify adapted populations and track carbon flow through a phenol-degrading soil microbial community.Applied and Environmental Microbiology,2005,71(12):7858-7865.
[17] Musat N,Halm H,Winterholler B,Hoppe P,Peduzzi S,Hillion F,Horreard F,Amann R,Jørgensen B B,Kuypers M M M.A single-cell view on the ecophysiology of anaerobic phototrophic bacteria.Proceedings of the National Academy of Sciences of the United States of America,2008,105(46):17861-17866.
[18] Francis C A,Beman J M,Kuypers M M M.New processes and players in the nitrogen cycle:the microbial ecology of anaerobic and archaeal ammonia oxidation.The ISME Journal,2007,1(1):19-27.
[19] Gruber N,Galloway J N.An Earth-system perspective of the global nitrogen cycle.Nature,2008,451(7176):293-296.
[20] Lechene C,Hillion F,McMahon G,Benson D,Kleinfeld A M,Kampf J P,Distel D,Luyten Y,Bonventre J,Hentschel D,Park K M,Ito S,Schwartz M,Benichou G,Slodzian G.High-resolution quantitative imaging of mammalian and bacterial cells using stable isotope mass spectrometry.Journal of Biology,2006,5(6):20-20.
[21] Lechene C P,Luyten Y,McMahon G,Distel D L.Quantitative imaging of nitrogen fixation by individual bacteria within animal cells.Science,2007,317(5844):1563-1566.
[22] Musat N,Foster R,Vagner T,Adam B,Kuypers M M M.Detecting metabolic activities in single cells,with emphasis on nanoSIMS.FEMS Microbiology Reviews,2011,36(2):486-511.
[23] Popa R,Weber P K,Pett-Ridge J,Finzi J A,Fallon S J,Hutcheon I D,Nealson K H,Capone D G.Carbon and nitrogen fixation and metabolite exchange in and between individual cells of Anabaena oscillarioides.The ISME Journal,2007,1(4):354-360.
[24] Finzi-Hart J A,Pett-Ridge J,Weber P K,Popa R,Fallon S J,Gunderson T,Hutcheon I D,Nealson K H,Capone D G.Fixation and fate of C and N in the cyanobacterium Trichodesmium using nanometer-scale secondary ion mass spectrometry.Proceedings of the National Academy of Sciences of the United States of America,2009,106(15):6345-6350.
[25] Ploug H,Musat N,Adam B,Moraru C L,Lavik G,Vagner T,Bergman B,Kuypers M M M.Carbon and nitrogen fluxes associated with the cyanobacterium Aphanizomenon sp.in the Baltic Sea.The ISME Journal,2010,4(9):1215-1223.
[26] Ploug H,Adam B,Musat N,Kalvelage T,Lavik G,Wolf-Gladrow D,Kuypers M M M.Carbon,nitrogen and O2fluxes associated with the cyanobacterium Nodularia spumigena in the Baltic Sea.The ISME Journal,2011,5(9):1549-1558.
[27] Foster R A,Kuypers M M M,Vagner T,Paerl R W,Musat N,Zehr J P.Nitrogen fixation and transfer in open ocean diatom-cyanobacterial symbioses.The ISME Journal,2011,5(9):1484-1493.
[28] Halm H,Musat N,Lam P,Langlois R,Musat F,Peduzzi S,Lavik G,Schubert C J,Sinha B,LaRoche J,Kuypers M M M.Co-occurrence of denitrification and nitrogen fixation in a meromictic lake,Lake Cadagno(Switzerland).Environmental Microbiology,2009,11(8):1945-1958.
[29] Dekas A E,Poretsky R S,Orphan V J.Deep-sea archaea fix and share nitrogen in methane-consuming microbial consortia.Science,2009,326(5951):422-426.
[30] Dekas A E,Orphan V J.Chapter twelve-Identification of diazotrophic microorganisms in marine sediment via fluorescence in situ hybridization coupled to nanoscale secondary ion mass spectrometry(FISH-NanoSIMS)//Martin G K,ed.Methods in Enzymology.San Diego:Academic Press,2011:281-305.
[31] Könneke M,Bernhard A E,de la Torre J R,Walker C B,Waterbury J B,Stahl D A.Isolation of an autotrophic ammonia-oxidizing marine archaeon.Nature,2005,437(7058):543-546.
[32] Tourna M,Stieglmeier M,Spang A,Könneke M,Schintlmeister A,Urich T,Engel M,Schloter M,Wagner M,Richter A,Schleper C.Nitrososphaera viennensis,an ammonia oxidizing archaeon from soil.Proceedings of the National Academy of Sciences of the United States of America,2011,108(20):8420-8425.
[33] Amann R,Fuchs B M.Single-cell identification in microbial communities by improved fluorescence in situ hybridization techniques.Nature Reviews Microbiology,2008,6(5):339-348.
[34] Li T L,Wu T D,Mazéas L,Toffin L,Guerquin-Kern J L,Leblon G,Bouchez T.Simultaneous analysis of microbial identity and function using NanoSIMS.Environmental Microbiology,2008,10(3):580-588.
[35] Behrens S,Lösekann T,Pett-Ridge J,Weber P K,Ng W O,Stevenson B S,Hutcheon I D,Relman D A,Spormann A M.Linking microbial phylogeny to metabolic activity at the single-cell level by using enhanced element labeling-catalyzed reporter deposition fluorescence in situ hybridization(EL-FISH)and NanoSIMS.Applied and Environmental Microbiology,2008,74(10):3143-3150.
[36] Liu X Z,Zhang L M,Prosser J I,He J Z.Abundance and community structure of sulfate reducing prokaryotes in a paddy soil of southern China under different fertilization regimes.Soil Biology and Biochemistry,2009,41(4):687-694.
[37] Fike D A,Gammon C L,Ziebis W,Orphan V J.Micron-scale mapping of sulfur cycling across the oxycline of a cyanobacterial mat:a paired nanoSIMS and CARD-FISH approach.The ISME Journal,2008,2(7):749-759.
[38] Nunan N,Ritz K,Rivers M,Feeney D S,Young I M.Investigating microbial micro-habitat structure using X-ray computed tomography.Geoderma,2006,133(3/4):398-407.
[39] O'Donnell A G,Young I M,Rushton S P,Shirley M D,Crawford J W.Visualization,modelling and prediction in soil microbiology.Nature Reviews Microbiology,2007,5(9):689-699.
[40] Herrmann A M,Clode P L,Fletcher I R,Nunan N,Stockdale E A,O'Donnel A G,Murphy D V.A novel method for the study of the biophysical interface in soils using nano-scale secondary ion mass spectrometry.Rapid Communications in Mass Spectrometry,2007,21(1):29-34.
[41] Clode P L,Kilburn M R,Jones D L,Stockdale E A,Cliff J B III,Herrmann A M,Murphy D V.In situ mapping of nutrient uptake in the rhizosphere using nanoscale secondary ion mass spectrometry.Plant Physiology,2009,151(4):1751-1757.
[42] Byrne M E,Ball D A,Guerquin-Kern J L,Rouiller I,Wu T D,Downing K H,Vali H,Komeili A.Desulfovibrio magneticus RS-1 contains an iron-and phosphorus-rich organelle distinct from its bullet-shaped magnetosomes.Proceedings of the National Academy of Sciences of the United States of America,2010,107(27):12263-12268.
[43] Orphan V J,House C H.Geobiological investigations using secondary ion mass spectrometry:microanalysis of extant and paleo-microbial processes.Geobiology,2009,7(3):360-372.
[44] Nunan N,Ritz K,Crabb D,Harris K,Wu K J,Crawford J W,Young I M.Quantification of the in situ distribution of soil bacteria by large-scale imaging of thin sections of undisturbed soil.FEMS Microbiology Ecology,2001,37(1):67-77.
[45] Peteranderl R,Lechene C.Measure of carbon and nitrogen stable isotope ratios in cultured cells.Journal of the American Society for Mass Spectrometry,2004,15(4):478-485.
[46] Berod A,Hartman B K,Pujol J F.Importance of fixation in immunohistochemistry:use of formaldehyde solutions at variable pH for the localization of tyrosine-hydroxylase.Journal of Histochemistry and Cytochemistry,1981,29(7):844-850.
