腹部CT图像序列中的肝脏分割
2013-09-02张春萌黄小川李佳佳
宋 红,张春萌,黄小川,李佳佳
(1.北京理工大学软件学院,100081北京;2.北京理工大学计算机学院,100081北京)
近年来,医学图像在计算机辅助诊断中得到广泛应用,从医学图像中精确的、自动的提取出肝脏区域对肝脏疾病的诊断和肝脏移植等手术的实施有着非常重大的意义.目前,医学图像的分割方法大致可分为:基于像素的分割方法[1-5]、基于轮廓的分割方法[6-7]以及这两种分割方法的结合[8].文献[2]提出了基于纹理特征的区域生长肝脏自动分割方法,自动选择种子点并自动计算阈值停止生长,可以很好的忽略肝脏中血管的干扰,但是在边界处往往有过分割现象.文献[5]采用基于统计学习理论的支持向量机(SVM)来实现肝脏的分割,首先提取图像的纹理特征,然后利用SVM分类,分离出肝脏区域与非肝脏区域,最后通过数学形态学运算来得到最终分割结果.在基于轮廓的分割方法中常用的是主动轮廓模型[9](Active Contours Model,又称Snake).由于Snake模型要求初始轮廓要非常接近目标区域的边界,而且其不能很好的深入到区域凹陷边界内部,所以C.Xu等[10]提出了梯度矢量流-蛇(GVF-Snake)模型.其中,外力由扩散方程得到,在整个图像区域计算梯度场,从而将Snake模型轮廓曲线拖向物体的深度凹陷区域.文献[11]采用基于GVF-Snake模型的肝脏分割方法,用最大力角度图自动找到初始轮廓,一定程度上解决了GVF-Snake模型在凹陷边界经常出错的问题,但是算法计算时间太长.文献[7]提出了基于马尔科夫随机场(Markov Random Field,MRF)模型和GVF-Snake模型的肝脏分割方法,用MRF得到肝脏的初始轮廓,然后用GVF-Snake模型调整初始轮廓.文献[12]提出了一种结合贝叶斯分类器和数学形态学操作以及几何主动轮廓的分割方法,通过迭代计算像素点包括种子点的矩形邻域的均值和方差来判断每个像素是否属于肝脏组织,然后执行形态学操作,最后用几何主动轮廓模型校正得到最终分割结果.
本文采用基于像素与基于轮廓结合的分割方法,首先利用阈值法提取肝脏的初始轮廓,然后用改进的GGVF-Snake[13-14]模型调整初始轮廓得到最终的分割结果,克服了单纯采用基于像素的分割方法存在的易过分割和泄漏问题,同时通过改进的GGVF-Snake模型解决了深入狭长凹陷边界的问题.
1 算法流程
实验所用数据为DICOM格式的腹部CT图像序列集.DICOM格式图像是CT扫描仪扫描后得到的原始图像,包含了许多医学信息,该CT序列的层厚为5 mm,每一层的大小是512*512,每一个像素包含一个12位的CT值,单位为HU(Hounsfield Unit).本文分割算法的总体流程如图1所示,首先采用Window-Leveling算法将原始CT图像转换为灰度图像,并通过改进的曲率各向异性扩散滤波器对图像进行平滑滤波;然后采用阈值法对图像进行粗分割,得到肝脏初始轮廓,同时通过Canny算子得到的边缘图计算外力场;最后采用改进的GGVF-Snake模型调整初始轮廓得到最终的分割结果.
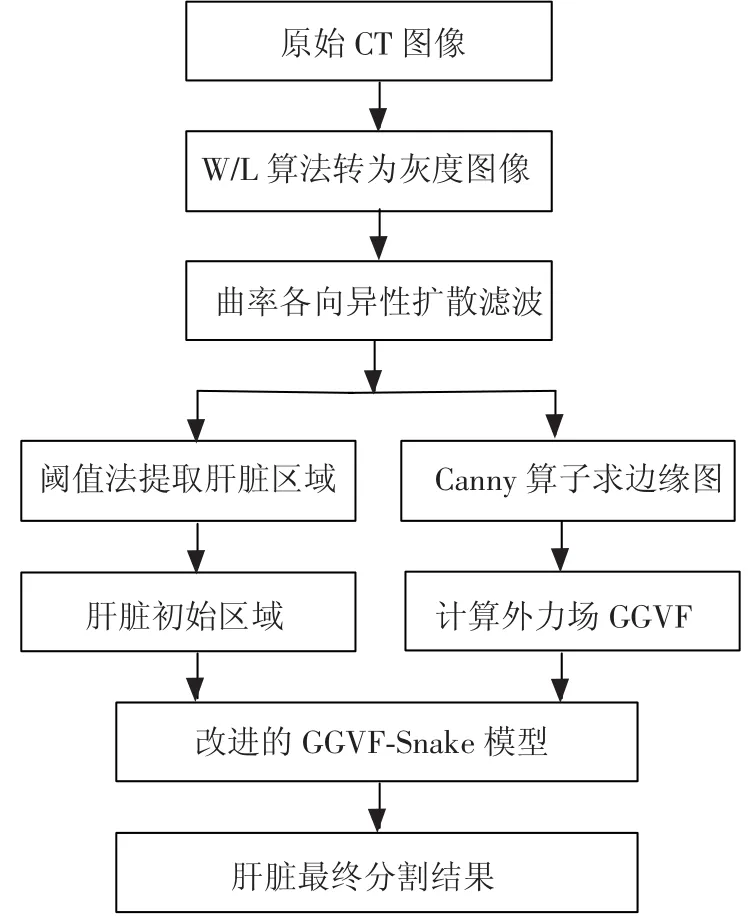
图1 肝脏分割算法流程
2 肝脏初始轮廓的获取
2.1 图像转换
原始腹部CT图像包含了肝脏、胃、脾等多个器官,且各器官的边缘模糊,如果强制将图像灰度化,则图像的分辨度不高,采用图像增强方法,很难突出所要研究的关键部位.在CT图像中,不同器官、组织的密度是在一个固定的值域,提取肝脏区域,只需将肝脏的CT值区间映射到灰度区间即可.本文采用Window-Leveling(W/L)调窗算法[15],实现肝脏CT图像到灰度图像的转换.在(W/L)调窗算法中,将需要处理的CT值区间一般称为窗口,区间长度称为窗口宽度(Window Width,WW),区间的中心称为窗口中心(Window Center,WC).图像转换的具体步骤如下:
1)将图像DICOM转为HU为
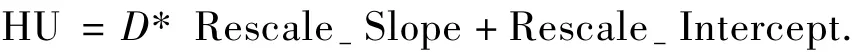
式中:D为图像DICOM值.本文所用数据集的Rescale_Slope值为1,Rescale_Intercept值为-1024.
2)计算映射区间最小值(min)和最大值(max)为
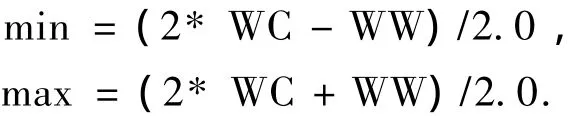
3)将HU映射到灰度区间[0,255]为
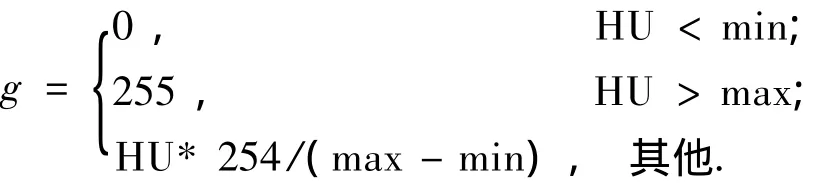
肝脏组织的CT值通常位于50~250之间,取WW=200,WC=150,将大小在50~250之间的HU映射到[1,254]的灰度值区间,<50映射为0,>250映射为255,得到CT图像的灰度图,如图2(b)所示.
2.2 曲率各向异性扩散滤波
P.Perona等[16]提出一种各向异性扩散的滤波器,用扩散的观点看待滤波,并给出了各向异性扩散方程,称为Perona-Malik(P-M)扩散模型,如
式中:div为散度算子;▽u为图像的梯;g(|▽u|)为扩散系数.各向异性扩散是一个能量发散的过程,从数学上等价于一个能量最小化问题的求解.各向异性扩散滤波通过估计噪声和梯度强度,在区域的内部进行平滑,有边缘的地方抑制平滑,很好的保护了图像的边缘特征.
本文采用改进的曲率扩散方程(Modified Curvature Diffusion Equation,MCDE)对图像执行各向异性扩散,如
经典的各向异性扩散在一定的条件下进行一个负扩散并增强边缘差异,MCDE形式的方程进行正扩散,且不具备边缘增强特征.同其他的非线性扩散技术相比,MCDE在质量上更好;与P-M扩散方式相比,MCDE具有更低的敏感度,并可更好的保留图像中的结构细节.图2(c)为经过基于MCDE的曲率各向异性扩散滤波后的图像.
图2 CT图像转换及滤波结果
2.3 肝脏初始轮廓的提取
经过上述处理后的图像,对比度增强,突出肝脏区域,图像直方图如图3所示.
肝脏区域的波峰通常落在灰度区间[100,150]之内,可通过统计方法找到峰值所对应的灰度值μ.但是,在非正常肝脏中,尤其是有大面积的肿瘤或其他病变的情况下,可能会有偏差,所以,为了保证阈值选择的准确性,本文首先选取了一个肝脏区域内的种子点s(x0,y0),然后选择离点s最近的波峰.另外,由于单一的阈值很难覆盖整个肝脏区域,本文使用上限和下限两个阈值对图像进行二值化,其中,下限阈值THL为区间[100,μ]内的极小值,而上限阈值THH为区间[μ,150]的极小值,按式(1)所示将灰度图像二值化,如图4(a)所示.

图像二值化后本文采用基于四连通的区域连通标记法标记连通区域,选择跟种子点s标记相同的像素,初步得到肝脏区域的像素集合,图4(b)为标记连通区域后提取的肝脏区域,从图4(b)可以看出,由于存在噪声,肝脏区域内部有许多的孔洞,利用数学形态学操作处理并填充孔洞得到完整的肝脏区域二值图像,如图4(c)所示.最后,通过边界跟踪得到肝脏区域初始轮廓的坐标序列,初始轮廓曲线如图4(d)所示.
图4 肝脏初始轮廓提取结果
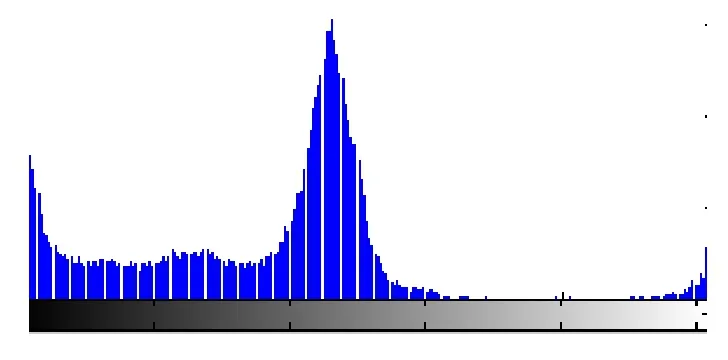
图3 预处理后的肝脏图像直方图
3 肝脏轮廓的调整
虽然传统的Snake模型在一定程度上可以达到分割的目的,但是,其存在两个问题:1)目标区域的初始轮廓必须足够接近目标边界;2)在目标边界有很深的凹陷时很难逼近到真实边界.在腹部CT图像序列中,很多序列中肝脏的边界都非常的不规则,传统的Snake模型分割效果不好.造成这种现象的原因是Snake模型的外力是一种保守力,捕捉范围小,限制了模型的应用.为了扩大外力场的捕捉范围,文献[10]提出了一种新的外力场来代替传统的外力场,这种外力场称为梯度矢量流(Gradient Vector Flow,GVF)外力场,表示为
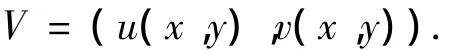
它可通过极小化的能量函数来求得
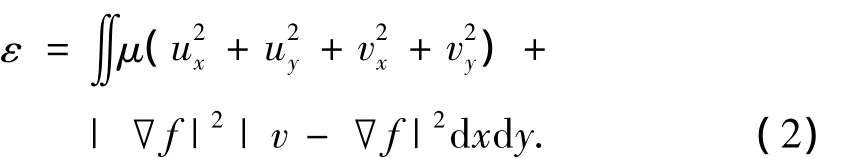
式中:参数μ是第1项和第2项的平衡因子,当μ=0时,式(2)变为经典的图像梯度外力场的情况,μ增大时,第1项起的平滑作用变大,能有效减少噪声,同时GVF场的作用范围也变大.由变分法得到偏微分方程为
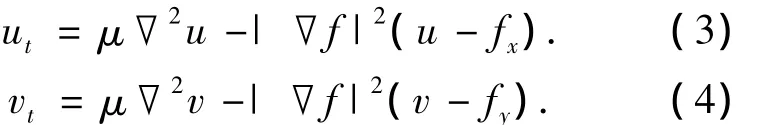
式中:ut、vt分别为对时间参数t的偏导;▽2为拉普拉斯算子;f(x,y)为边缘图,它在图像的边缘处取得极大值,对于灰度图像Ⅰ(x,y),一般边缘图定义为f(x,y)=|▽Gσ*Ⅰ(x,y)|2.其中,Gσ为标准差为σ的高斯核函数.通过求解偏微分方程组可得到式(2)的解.
梯度矢量流外力场作为Snake模型的外力场在一定程度上解决了曲线不能深入到凹陷边界内部的问题,但是,在特别细长的边界处,普通的GVF仍然不能达到很好效果,这是因为在边界处GVF力场存在着过度平滑问题,因此,用一种新的可变的权重函数来代替式(2)中的常数平滑因子μ可以解决这个问题,这就是广义的梯度矢量流(Generalized gradient vector flow,GGVF).用更加泛化的权重函数g(|▽f|)和h(|▽f|)分别取代式(3)、(4)中的μ和|▽f|2后得到GGVF所对应的偏微分方程为
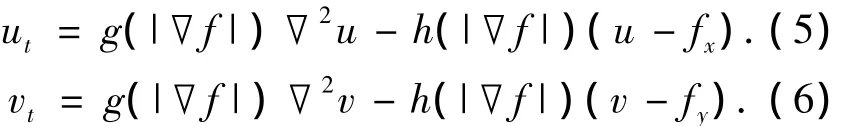
当g(|▽f|)=μ,h(|▽f|)=|▽f|2时,式(5)、(6)就分别退化为式(3)、(4).在GGVF中,权重函数g(|▽f|)和h(|▽f|)分别为
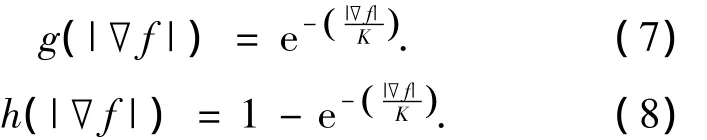
当权重函数h(|▽f|)变大时,g(|▽f|)变小时,在梯度比较大的地方,平滑度就会比较小,反之,平滑度将变大.式(7)、(8)即为广义的扩散方程[13].
另外,在腹部CT图像中,噪声较多,组织器官也较多,所以传统的通过计算图像梯度得到式(2)中的边缘图f,即f(x,y)=|▽Gσ*Ⅰ(x,y)|2,往往会使产生的梯度矢量流场杂乱,最终影响分割结果的准确性.如果通过增大标准差σ来减少噪声,则会使本来就模糊甚至是重叠的图像边缘更加不清晰,也会导致流场错乱,进而导致分割失败.所以,本文采用了边缘检测效果更好的Canny算子获得到图像的边缘图[11,14,17].Canny算法主要分4步来得到边缘图.
1)用高斯函数与原图像作卷积.
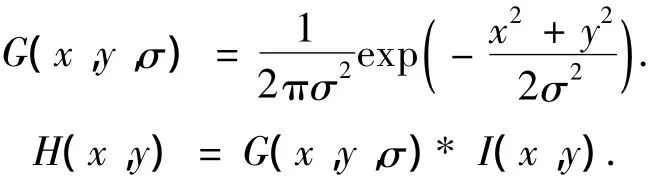
2)利用一阶偏导的有限差分计算梯度的幅值和方向.
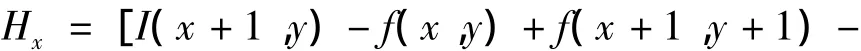
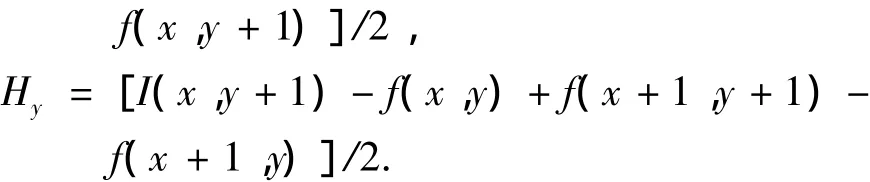
3)非极大值抑制.仅仅得到全局的梯度并不足以确定边缘,为了确定边缘,必须保留局部梯度值最大的点,并抑制非极大值.每一个像素点与沿梯度方向相邻的两个像素比较,如果不比沿梯度方向的两个相邻相素梯度值大,则设为0.
4)利用双阈值法检测和连接边缘.通过Canny算子处理得到的边缘图,不需要的边缘细节被过滤,真实边缘被精确的保留,利用此边缘图计算得到的广义梯度矢量流场有着很好的稳定性和清晰度.在此力场中,轮廓曲线可以很好的被校正为真实轮廓.图5为Canny算子得到的边缘图与传统的梯度法得到的边缘图.

图5 Canny算子和梯度法获取的边缘图
4 结果与分析
实验所用的腹部CT序列图像共2组420张,算法由Matlab以及ITK共同实现.目前,已经有许多的评估方法被应用于医学图像的分割中.但是,由于在肝脏的分割研究中,没有统一的、标准的可用数据集,使得在肝脏的分割中并没有统一的评测标准.当前比较常用的评估分割结果的方法是差异实验法,即将计算机分割结果与正确的分割结果相比较.在医学图像的分割中,正确的分割结果一般通过手动的分割来得到.本文将通过Dice相似性系数[18](Dice Similarity Coefficient,DSC)、误报率(False Positive Ratio,FPR)和漏报率(False Negative Ratio,FNR)3个指标来评估分割结果,它们的定义为
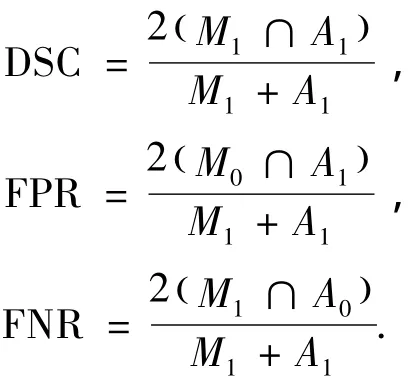
式中:M、A分别为手动分割结果与计算机分割结果;M0、A0分别为相应的分割结果中非肝脏的像素集;M1、A1分别为相应的分割结果中肝脏区域像素集.DSC的取值范围在0~1之间,表示计算机分割结果与真实结果之间的重合程度,DSC=0说明M1和A1两个集合完全没有重叠,分割效果最差;DSC=1则说明两个集合完全一致,分割效果最好.FPR为本不属于肝脏的像素却被标记为肝脏像素的比例;FNR为本属于肝脏的像素却没有被标记为肝脏像素的比例.
表1是10个CT图像序列样例分割结果的评估数据.在用改进的GGVF-Snake模型对肝脏轮廓的调整中,每张序列图像的迭代用时为5 s左右,单次迭代时间为45 ms左右.从表1可以看出,算法分割结果与手动分割结果的DSC在97%以上,证明了算法的准确度,同时FNR和FPR也降至0.05以下.而基于图割理论的文献[18]中DSC为95%,FPR、FNR分别为5%和3%,基于统计信息的文献[19]中DSC为94%、FPR为3.9%、FNR为6.3%.在肝脏区域没有跟其他的组织重叠的序列中,算法的DSC可达到98%以上(如图7第1行);在肝脏区域形状比较复杂且跟周围组织有重叠,甚至某段边界不存在的序列中(如图7第2行),DSC会稍有下降,一般在97±1%左右,这是因为在有些边缘差异极小的情况下,采用灰度信息的分割方法会有过分割的现象.另外,从图6可看出,采用改进的GGVF作为Snake模型的外力后,在肝脏凹陷边界处的力场有了明显的改进,这使得轮廓曲线可以快速有效的深入到真实的边界内部.

图6 肝脏边界凹陷处力场图
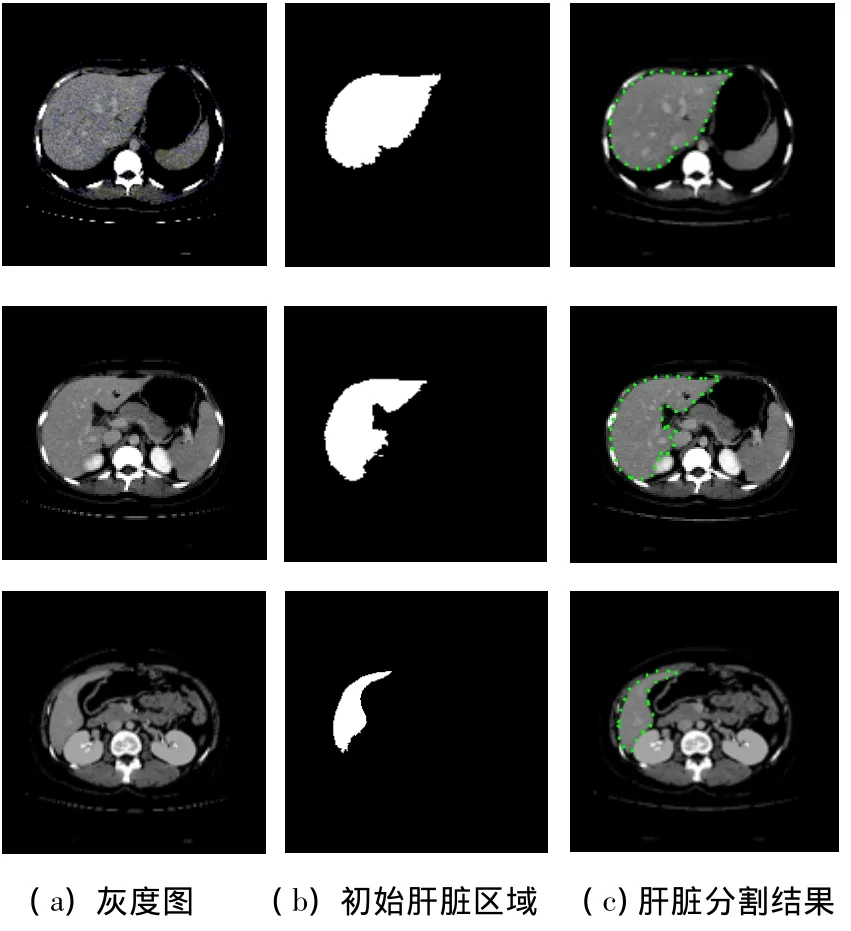
图7 肝脏最终分割结果
5 结论
1)采用基于CT值的W/L算法将原始图像转换为灰度图像,使得阈值法提取肝脏区域变得容易,同时采用双阈值法得出的肝脏初始轮廓也具有很高的精确度;
2)通过改进GVF外力场,提高了外力场的稳定性与清晰度,降低了Snake模型对初始轮廓精度的要求,解决了GVF-Snake模型不能深入到极细长的凹陷边界内部问题,得到较好的分割结果.
[1]CHRIST M C J,PARVATHI R M S.Fuzzy C-means algorithm for medical image segmentation[C]//Proceedings of the 3rdInternational Conference on Electronics Computer echnology(ICECT).Kanyakumari:IEEE,2011,(4):33-36.
[2]GAMBINO O,VITABILE S,RE G L,et al.Automatic volumetric liver segmentation using texture based region growing[C]//Proceedings of the International Conference on Complex,Intelligent and Software Intensive Systems(CISIS).Krakow:IEEE,2010:146-152.
[3]CHITTAJALLU D R,SHAH S K,KAKADIARIS I A.A shape-driven MRF model for the segmentation of organs in medical images[C]//Proceedings of the Conference on Computer Vision and Pattern Recognition(CVPR).San Francisco,CA:IEEE,2010:3233-3240.
[4]ZHAO Yu-qian,ZAN Yun-long,WANG Xiao-fang,et al.Fuzzy-means clustering-based multilayer perceptron neural network for liver ct images automatic segmentation[C]//Proceedings of 2010 Chinese Control and Decision Conference(CCDC).Xuzhou,Jiangsu:IEEE,2010:3423-3427.
[5]LUO Su-huai,HU Qing-mao,HE Xiang-jian,et al.Automatic liver parenchyma segmentation from abdominal CT images using support vector machines[C]//Proceedings of the International Conference on Complex Medical Engineering(ICME).Tempe,AZ:IEEE,2009:1-5.
[6]WANG Wen-feng,MA Li,YANG Lin.Liver contour extraction using modified snake with morphological multiscale gradients[C]//Proceedings of the International Conference on International Conference on Computer Science and Software Engineering.Wuhan,Hubei:IEEE,2008,(6):117-120.
[7]CAMPADELLI CASIRAGHI P E,PRATISSOLI S.Fully automatic segmentation of abdominal organs from CT images using fast marching methods[C]//Proceedings of the 21stIEEE International Symposium on Computer-Based Medical Systems.Washington,DC:IEEE Computer Society,2008:554-559.
[8]ALOMARI R KOMPALLI S S,CHAUDHARY V.Segmentation of the liver from abdominal CT using markov random field model and GVF snakes[C]//Proceedings of the 2008 International Conference on Complex,Intelligent and Software Intensive Systems.Washington,DC:IEEE Computer Society,2008:293-298.
[9]KASS M,WITKIN A,TERZOPOULOS D.Snakes:active contour models[J].International Journal of Computer Vision,1988,1(4):321-331.
[10]XU Chen-yang,PRINCE J L.Snakes,shapes,and gradient vector flow[J].IEEE Transactions on Image Processing,1998,7(3):359-369.
[11]HUANG Shao-hui,WANG Bo-liang,HUANG Xiaoyang.Using GVF snake to segment liver from CT images[C]//Proceedings of the 3rdInternational Summer School on Medical Devices and Biosensors.Cambridge,MA:IEEE,2006:145-148.
[12]FREIMAN M,ELIASSAF O,TAIEB Y,et al.An iterative Bayesian approach for nearly automatic liver segmentation:algorithm and validation[J].International Journal of Computer Assisted Radiology and Surgery,2008,3(5):439-446.
[13]XU Chen-yang,PRINCE J L.Generalized gradient vector flow external forces for active contours[J].Signal Processing,1998,71(2):131-139.
[14]GUI Tian-yi,HUANG Lin-Lin,SHIMIZU A.Liver segmentation for CT images using an improved GGVFSnake[C]//Proceeding of the 2007 Annual Conference on SICE.Takamatsu:IEEE,2007,676-681
[15]Mayo-SMITH W W,GUPTA H,.RIDLEN M S,et al.Detecting hepatic lesions:the added utility of CT liver window settings[J].Radiology,1999,210(3):601-604.
[16]PERONA P,MALIK J.Scale-space and edge detection using anisotropic diffusion[J].IEEE Transactions on Pattern Analysis and Machine Intelligence,1990,12(7):629-639.
[17]CHENG Jin-yong,XUE Ruo-juan,LU Wen-peng,et al.Segmentation of medical images with Canny operator and GVF snake model[C]//Proceedings of the 7thWorld Congress on Intelligent Control and Automation.Chongqing:IEEE,2008,1777-1780.
[18]MASSOPTIER L,CASCIARO S.Fully automatic liver segmentation through graph-cut technique[C]//Proceedings of the29thAnnunal International Conference on Engineering in Medicine and Biology Society.Lyon:IEEE,2007:5243-5246.
[19]MASSOPTIER L,CASCIARO S.A new fully automatic and robust algorithm for fast segmentation of liver tissue and tumors from CT scans[J].European Radiology,2008,18(8):1658-1665.
