传统分离培养结合PCR-DGGE技术分析广式腊肠中优势菌
2013-08-07余晓峰郑海松连英琪刘国庆
谢 科,余晓峰,郑海松,宗 凯,连英琪,刘国庆,*
(1.合肥工业大学生物与食品工程学院,安徽 合肥 230009;2.安徽出入境检疫检验局,安徽 合肥 230022)
广式腊肠是中国传统特色食品之一,具有外形美观、色泽明亮、香味醇厚、鲜味可口、皮薄肉嫩的特点,备受国人喜爱。目前,市售广式腊肠仍处于作坊式的生产阶段,主要靠原料肉自身的微生物与环境微生物的竞争作用完成其发酵过程,属于自然发酵[1]。而其在肉制品产业发达的国家,已经完成了从传统的自然发酵向微生物定向接种发酵的工业化生产的转变。微生物定向接种发酵具有发酵启动快、发酵时间短、低值等优点,极大地改善了肉制品的感观品质,提高了食品的安全性[2]。因此分离筛选广式腊肠中的主要微生物,构建具有优良生产性状的微生物发酵剂,对广式腊肠的生产和发展具有重要意义。目前对传统发酵食品中微生物的研究主要还是应用传统的分离﹑纯化﹑鉴定方法,需要进行一系列繁杂的形态特征和生理生化试验,这种方法最大的缺点是即使使用最复杂的试验组合也不能对分离物进行精确鉴定,不能反映分离物间的系统发育关系,不能获得微生物多样性的真正概貌[3-4]。1993年,Muzyer等[5]首次将变性梯度凝胶电泳法(denaturing gradient gel electrophoresis,DGGE)技术应用于微生物生态学研究,并证实了这种技术在研究自然界微生物群落的遗传多样性和种群差异方面具有明显的优越性。DGGE能有效分析复杂微生物群落及其多样性,且无需培养微生物,既可以用于传统发酵产品中微生物的种群结构分析,也可以用来对分离出的纯菌株进行分组和筛选,减少分子或生化鉴定的数量,具有快速可靠的特点[6]。Fontana等[7]利用PCR-DGGE技术对两种手工阿根廷发酵干香肠发酵过程进行监控和调查优势细菌群落,发现这种技术能够区分乳酸菌和革兰氏阳性凝固酶阴性球菌,是一种有效的确定手工发酵干香肠优势微生物菌群的方法。目前对广式腊肠微生物的研究主要集中在微生物分布及作用机理了解等方面,而通过传统分离方法从广式腊肠中分离出某一个菌株或几个菌株,系统全面地分析广式腊肠微生物群落结构的研究尚未有报道。本实验采用传统培养技术与PCR-DGGE指纹技术相结合的方法对广式腊肠微生物进行全面系统研究,分析其微生物群落结构组成,从而筛选出其中的优势菌,以期为广式腊肠发酵剂的开发研究提供实验依据。
1 材料与方法
1.1 材料与试剂
皇上皇广式腊肠购自广州越秀区东川路皇上皇腊味店。
PCA、MRS琼脂 青岛海博生物技术有限公司;细菌基因组DNA提取试剂盒 北京天根生化科技有限公司;Taq DNA聚合酶、2000bp DNA Marker、引物合成 大连宝生物工程公司;聚丙烯酰胺、尿素、去离子甲酰胺 西班牙Sigma公司;GelRed核酸凝胶染色剂 美国Biotium公司;DNA测序 上海生工生物工程公司。
1.2 仪器与设备
LDZX-40AI型立式自动电热压力蒸汽灭菌器 上海申安医疗器械厂;DH5000AB型电热恒温培养箱 天津泰斯特仪器有限公司;高速冷冻离心机 德国Beckman公司;DGGE电泳分析系统 北京君意东方电泳设备有限公司;PCR仪 美国AB公司。
1.3 方法
1.3.1 细菌的分离和鉴定
在无菌条件下称取25g广式腊肠样品,剪碎后加入225mL灭菌生理盐水(含1g/L蛋白胨、9g/L NaCl)摇床振荡培养30min。取1mL上清液依次进行10倍梯度稀释,选择合适的稀释度涂布于不同的培养基中。MSA、MRS培养基分别于30℃和37℃培养48h后挑选特征性菌落,在分离培养基上多次划线分离纯化,最后对所得菌株根据《伯杰氏细菌鉴定手册》第九版[8]及《常见细菌系统鉴定手册》[9]对所得的优势菌群进行形态和生理生化测定。
1.3.2 细菌DNA提取
无菌条件下称取广式腊肠10g,剪碎加入50mL灭菌生理盐水,摇床振荡培养30min。4℃条件下500r/min离心10min,取上清液于4℃条件下12000r/min离心15min,弃上清液,将沉淀置于1.5mL离心管,应用细菌基因组DNA提取试剂盒提取细菌总DNA。分离纯化得到的菌株先将其接种到50mL生理盐水中,振荡培养30min。按上述方法分别提取其DNA,将所提取DNA溶于TE缓冲液,-20℃条件下贮藏。
1.3.3 PCR扩增
采用细菌通用引物F357和R518对细菌16S rDNA的V3可变区进行PCR扩增。上游引物为带GC夹的F357:GC-F357(5′-CGCCCGCCGCGCGCGGCGGGCGGGGCG GGGGCACGGGGGGCCTACGGGAGGCAGCAG-3’);下游引物为R518(5’-ATTACCGCGGCTGCTGG-3’)[10]。PCR反应体系:10×Ex Taq Buffer 2.5μL,dNTP(2.5mmol/L)2μL,MgCl2(25mmol/L)2μL,Ex Taq DNA聚合酶(5U/μL,TaKaRa)0.25μL,模板DNA 2μL,上下游引物各1μL,补充ddH2O 14.25μL至终体积25μL。DNA扩增采用降落PCR,反应程序见参考文献[6]。取5μL PCR产物,经1.0%琼脂糖凝胶电泳检测含量及特异性后置于-20℃冰箱保存备用。
1.3.4 DGGE分析
PCR产物的DGGE分析在DGGE电泳分析系统上进行。参照Tatsadjieu等[11]的方法进行,电泳条件:质量分数8%的聚丙烯酰胺凝胶(丙烯酰胺:甲叉双丙烯酰胺= 37.5:1),变性梯度从30%到50%(100%变性剂含有7mol/L尿素和40%去离子甲酰胺),PCR产物上样量20μL,电泳温度恒定60℃,在1×TAE缓冲液中先200V预电泳10min,然后在80V的固定电压下电泳8h,采用GelRed核酸凝胶染色剂在1×TAE缓冲液中染色15min后,用凝胶成像系统进行拍照保存,图像分析用Quantity one分析软件进行分析。
1.3.5 DNA的回收测序
在紫外灯下切下DGGE胶泳道中的主要亮带,条带按序分别放入标号的1.5 m L E P 管中,捣碎后加入3 0 μ L 灭菌d d H2O,-2 0 ℃条件下浸泡过夜,离心,取上清液作为模板进行P C R 扩增。引物为F357(5′-ACTCCTAC GGGAGGCAGCAG-3′)和R518(5′-ATTACCGCGGCTGCTGG-3′)。扩增程序:94℃预变性4min,30个循环(94℃变性30s,55℃退火30s,72℃延伸30s),最终72℃延伸10min。PCR产物经1.0%琼脂糖凝胶电泳检验产量及特异性后测序。登陆NCBI,将所得序列与数据库中已知序列进行比较,用Clustal X和MEGA进行相似性分析。
2 结果与分析
2.1 可培养优势菌的分离和鉴定
用MSA、MRS培养基从广式腊肠中分离出葡萄球菌19株,乳酸菌12株。对19株球菌进行形态学观察,参照《常见细菌系统鉴定手册》和《伯杰氏系统细菌学手册》对菌株进行生理生化试验、糖醇发酵试验、凝固酶反应等,最后筛选得到4株葡萄球菌,分别标号为P1~P4。对分离得到12株乳酸菌进行耐盐试验、耐亚硝盐试验、接触酶活性试验、氨基酸脱羧酶活性试验、硝酸盐还原酶活性试验、产氨试验、产硫化氢试验、产气试验、产粘液试验等生理生化试验鉴定,最终有2株乳酸菌属于不同种属,标号为L1、L2。
2.2 细菌16S rDNA的V3可变区的PCR扩增结果
将所提取的广式腊肠混合菌群总DNA和各纯化菌株DNA用16S rDNA的V3区引物(带GC夹)进行PCR扩增,扩增产物经1.0%琼脂糖电泳检测,获得约200bp左右的特异性扩增条带,见图1。扩增产物大小符合目的片段要求,且条带清晰,适合进行下步DGGE电泳。

图 1 细菌16S rDNA的V3区的PCR扩增电泳图Fig.1 PCR amplification pattern of bacterial 16S rDNA from V3 region
2.3 广式腊肠混合菌群和分离纯菌株的PCR-DGGE指纹图谱分析
如图2所示,广式腊肠混合菌群样(泳道1)在DGGE电泳图谱上显示出7条可鉴别的条带,分别为A、B、C、d、e、f、g。DGGE 图谱上不同条带代表不同的微生物种类,条带的亮度代表微生物的数量,条带越亮则微生物的数量越多[5],即A、B、C条带所代表的菌株为广式腊肠中的主要优势菌,d、e、f、g条带所代表的菌株为广式腊肠中的次要优势菌。传统方法分离纯化所得的菌株(泳道2~7)DGGE电泳图谱显示,2、5号泳道条带与混合菌群中的条带C在同一位置,3号泳道条带与d条带在同一位置,4号泳道条带H与混合菌群样中条带无对应,6号泳道条带与g条带位置一致,7号泳道条带与B位置一致。2、5号泳道条带出现在同一位置,表明两者可能为同一菌株。4、6、7号泳道出现多条杂带,表明PCR产物不纯,可能为PCR扩增中出现的引物二聚体或小片段产物。
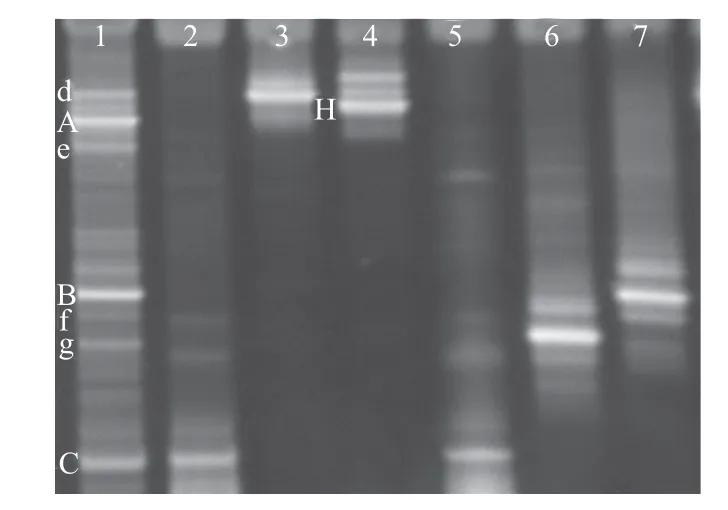
图 2 广式腊肠中混合菌群和分离纯菌株PCR-DGGE指纹图谱Fig.2 PCR-DGGE analysis of 16S rDNA fragments from microbial community (1) and isolated bacteria (2-7) in Cantonese sausages
2.4 细菌16S rDNA的测序及序列分析

表 1 DGGE图谱优势条带序列比对结果Table 1 Tentative identification of predominant bacteria from DGGE bands by sequencing
对图2中标记的较明显的条带进行割胶回收、测序,将所得序列与数据库中已知序列进行比较,用Clustal X和MEGA进行相似性分析,见图3,测序结果如表1所示。通过序列比对和相似性分析,条带A、B、C、d、e、f、g、H代表的菌种分别为:腐生葡萄球菌、乳杆菌属、木糖葡萄球菌、孔氏葡萄球菌、表皮葡萄球菌、芽孢杆菌属、格氏乳球菌、阿尔莱特葡萄球菌。传统分离方法所得菌株分别为木糖葡萄球菌(P1、P4)、孔氏葡萄球菌(P2)、阿尔莱特葡萄球菌(P3)、格氏乳球菌(L1)、乳杆菌属(L2)。

图 3 DGGE条带序列与GenBank数据库中相关序列的相似性分析Fig.3 Similarity analysis of sequences derived from DGGE bands and relative sequences from GenBank
3 讨 论
3.1 本研究将常规的分离技术与PCR-DGGE指纹图谱技术相结合,应用于广式腊肠中优势细菌的筛选,结果显示用PCR-DGGE方法直接从广式腊肠中获得的细菌群落结构与用传统培养技术获得的有一定的差异。条带A、e、f均未在分离菌中条带出现,这说明PCR-DGGE方法能够克服传统分离培养方法的不足,更能直观地反映出广式腊肠中细菌的组成。分离菌中有两个条带出现在同一位置,经测序为同一菌株,这表明传统的分离培养方法不够准确,存在一定的缺陷。分离菌中条带H未在混合菌群条带中出现,Heuer等[12]报道了在PCR过程中群落结构中相对丰富的菌株由于竞争作用会影响某些菌株的扩增,也可能是因为该群落细菌数量太少,用常规的DNA提取方法可能提不到或提到微量的DNA,这会影响其PCR扩增,从而使得条带H在混合菌群泳道条带中表现不出来。
3.2 本实验将PCR-DGGE指纹图谱技术应用于广式腊肠中微生物群落结构研究,通过PCR-DGGE指纹图谱技术分析确定的主要优势菌为腐生葡萄球菌、乳杆菌属、木糖葡萄球菌,次要优势菌为孔氏葡萄球菌、表皮葡萄球菌、芽孢杆菌属、格氏乳球菌。传统分离培养试验从广式腊肠中得到3株葡萄球菌、2株乳酸菌,分别为木糖葡萄球菌、孔氏葡萄球菌、阿尔莱特葡萄球菌、格氏乳球菌、乳杆菌属。实验分析结果显示广式腊肠中微生物群落有3类,乳酸菌、葡萄球菌和芽孢杆菌,这与乔晓玲[13]对广式腊肠微生物的检测结果相同。在发酵肉制品中,葡萄球菌能分泌蛋白酶,在一定程度上对蛋白质有水解作用[14],有助于提高发酵肉制品中可溶性蛋白质和游离氨基酸的含量,对发酵肉制品风味的形成有重要意义,乳酸细菌则主要是降低了环境中的pH值,抑制了部分腐败和病原微生物,提高了产品的安全,延长了货架期[15]。将分离所得的葡萄球菌和乳酸菌相结合,组成发酵剂,检验其安全性,验证其发酵效果,最终可得到安全高效的广式腊肠发酵剂,为广式腊肠的工业化生产提供技术支持。
[1] 吕兵, 张国农. 分离自传统腊肠中的乳酸菌的特性研究[J]. 食品与发酵工业, 2004, 30(8): 64-67.
[2] MARTA M, JOSEP M. Bacteria starter cultures for meat fermentation [J]. Food Chemistry, 1997(59): 457-554.
[3] AMANN R I, LUDWIG W, SCHLEIFER K H. Phylogenetic identification and in situ detection of microbial cells without cultivation [J]. Micro- biological Reviews, 1995, 59(1): 143-169.
[4] HUGENHOLTZ P, GOEBEL B M, PACE N R. Impact of cultureindependent studies on the emerging phylogenetic view of bacterial diversity[J]. Journal of Bacteriology, 1998, 180(18): 4765-4774.
[5] MUYZER G, de WAAL E C, UITTERLINDEN A G. Profiling of complex microbial populations by denaturing gradient gel elect- rophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA[J]. Applied of Environment Microbiology, 1993, 59(3): 695-700.
[6] 周剑忠, 董明盛, 江汉湖. PCR-DGGE指纹技术与分离技术结合筛选藏灵菇奶发酵过程的优势菌[J]. 中国农业科学, 2006, 39(8): 1632-1638.
[7] FONTANA C, VIGNOLO G, COCCONCELLI P S. PCR-DGGE analysis for the identification of microbial populations from Argentinean dry fermented sausages[J]. Journal of Microbiological Methods, 2005, 63(3): 254-263.
[8] JOHN G H, NOBEL R K, PETER H A, et al. Bergey’s Manual of determinative bacteriology[M]. 9th ed. Baltimore: Williains&Wilkins Press, 1994: 97-116.
[9] 东秀珠, 蔡妙英, 王宝玲, 等. 常见细菌系统鉴定手册[M]. 北京: 科学出版社, 2001: 216-635.
[10] KRIS A, KLAAS D, KATHY M, et al. Diversity of lactic acid bacteria from modified atmosphere packaged sliced cooked meat products at sell-by date assessed by PCR-denaturing gradient gel electrophoresis[J]. Food Microbiology, 2010, 27(1): 12-18.
[11] TATSADJIEU N L, MAIORE J, HADJIA M B, et al. Study of the microbial diversity of Oreochromis niloticus of three lakes of Cameroon by PCR-DGGE: application to the determination of the geographical origin[J]. Food Control, 2010, 21(5): 673-678.
[12] HEUER H K, HARTUNG K, WIELANG G, et al. Polynucleotide probes that target a hypervariable region of 16S rRNA genes to identify bacterial isolates corresponding to bands of community fi ngerprints[J]. Applied and Environmental Micro-biology, 1999, 65: 1045-1049.
[13] 乔晓玲. 七种名牌广式腊肠的品质及贮藏期的分析研究[J]. 肉类工业, 1995(12): 21-23.
[14] 陆明. 按西德香肠质量标准测定中国腊肠的质量指标[J]. 肉类工业, 1988(8): 37-39.
[15] 李宗军. 中国传统酸肉中葡萄球菌的分离鉴定与应用研究[J]. 生物技术通报, 2006(3): 77-80.
