面向GWAS未覆盖基因组区的数据集成
2013-07-31裘嵘刘春宇吴敏
裘嵘 ,刘春宇,吴敏
(1. 中南大学 信息科学与工程学院,湖南 长沙,410083;2. 先进控制与智能自动化湖南省工程实验室,湖南 长沙,410083;3. 伊利诺伊大学 芝加哥分校人类遗传学研究所,美国 芝加哥,60607)
全基因组关联研究(genome-wide association studies,GWAS)是在全基因组范围内对常见遗传变异进行多基因整体关联研究分析[1-2]。目前已报道的常见基因变异仅能解释部分常见遗传病,而当前的基因型分析(genotyping)技术无法检测到的罕见遗传变异或表观遗传变异极有可能位于 GWAS未覆盖基因组区域。为此,很有必要对该区域进行研究。GWAS的特点是:(1)数据库数量多,异构复杂,分布广;(2)数据量极大;(3)转化算法复杂;(4)数据更新量大,无自动机制,接口可能变化。大部分生物数据以文本文件的形式存在[3-6],目前已有大量针对生物数据库的数据集成的研究[7],国内韩建文等[8-10]也对GWAS现状进行了分析和综述,但还未见针对整个GWAS未覆盖基因组区域的研究。为了有效整合GWAS中众多生物信息学数据库中的数据资源,便于对整个GWAS未覆盖基因组区域进行研究,有必要对整个GWAS数据进行数据集成。GWAS未覆盖基因组区域的数据集成研究的总体思路为:原始数据源中的数据经过语意映射对应到全局语意上,形成缓存局部数据库;用户查询时,用户接口将针对全局视图的查询通过查询重组转化为对缓存局部数据库的查询,从而保持了数据在不同系统上的完整性和一致性;同时,向用户隐藏不同数据源的差异,给用户提供1个统一和透明的数据访问接口。其中的关键步骤为语意映射,如 Adaptive clustering[11]和 Automatch[12]等。基于学习的技术优点在于能够挖掘模式和样本中的潜在对应关系,具备统计意义上的健壮性,已成为当前语意映射问题中普遍采用的解决办法。为此,本文作者针对当前基于机器学习语意映射中的不足,结合半监督学习方法,提出一种基于自训练的自动语意映射技术。该技术利用未标注样本的分布信息对于分类边界的提示作用,增强语意映射的准确性,减小对人类专家的依赖性,从而实现自动的语意映射和对整个 GWAS未覆盖基因组区域的数据集成。
1 初始数据源及特性分析
数据集成的研究对象为目前 GWAS中的未覆盖基因组区。首先,选取GWAS中有代表性的3种数据资源,在这些数据资源上进行处理,以得到分析整个GWAS未覆盖基因组区的方法。这3种数据资源为:
(1)SNPs in HapMap。HapMap是人类基因组中常见遗传多态位点的目录。
(2)SNPs in dbSNP。dbSNP数据库是一个公共开放的简单基因多态点数据库。
(3)UCSC Genome Browser,是由 University of California Santa Cruz (UCSC)创立和维护的多个物种的基因组数据库。
数据下载地址等信息见表1。
分析数据源的特点选取合适的数据集成方法。各初始数据源具有以下特点。
(1)多样性。面向GWAS中的未覆盖基因组区的数据研究需要选取各数据库中描述SNP的编号、染色体名、位置等关键字段来进行计算,而在 HapMap,dbSNP和1000 Genome sequencing Data等数据库中这些字段的顺序、位置各不相同,有的还需要经过计算或处理其他相关字段才能得出,呈现出高度的多样化。
(2)更新性。由于相关领域的研究处于持续发展的状态,局部数据库中的数据常常会不断地变化。许多数据源几个月内就要扩展或更改数据库,并以同样的频率修改数据库的使用界面。
(3)自治性。各局部数据库是松散、自治的,其数据来源由不同的研究机构创建和维护,有自己的数据模型,采用不同的数据模式,还可以对已有的模型、模式和结构等自由地进行更改,提供不同的查询服务等。
(4)异构性。数据元素在数据格式上是异构的,如文本文件、XML文件、二进制文件等;同时,在语意上可能也是异构的。不同局部数据库中相同或相关数据在含义、解释和用途方面都有可能不同。
2 数据集成的实现原理
当前语意映射的研究集中在基于机器学习的方法上。这种方法的典型过程为:首先由人类专家将已有的少数几个数据源映射到全局语意上,然后,根据这种映射形成若干训练样本,采用机器学习算法在训练样本上训练语意映射分类器。当需要加入新的数据源时,只需将新数据源中的数据转化为测试样本并提交给语意映射分类器,语意映射分类器即可预测新数据源与全局语意的映射关系。

表1 本研究使用的数据来源Table 1 Data sources used in this study
目前,基于机器学习的语意映射主要采用监督学习算法。而要使监督学习算法训练得到的分类器达到理想的分类正确率,训练样本的数量必须达到一定的数量,而这往往不易满足。
近年来,半监督学习技术开始受到机器学习研究人员的关注。半监督学习基于一个基本假设即聚类假设。该假设的直观表达为:在高密度区域内相近的点很可能具有相同的标注。可以简单地将其看作数据具有局部相似性。半监督学习即利用这种局部相似性增强分类器的分类能力。这种技术适用于训练样本较少而未标注样本却很多的应用环境。
半监督学习中主要的研究方法包括以下几类:自训练[13-14]、生成模型[15]、低密度分割[16]、基于图的方法[17]等。其中,自训练方法易于理解,实现简单,应用较广泛。为此,本文作者开发了基于自训练的自动模式映射技术。
2.1 基于自训练的全局-局部自动模式映射
模式匹配的目标是寻找2个或多个模式之间在语意上的对应关系。本文从自动加入新的相关数据源角度出发,提出一种基于自训练的自动模式映射方法。这种方法的目的在于找出未来新加入数据源时实现新数据源数据模式与全局模式的匹配,即找出字段之间的一一对应关系。问题的形式化形式如下。
假设有 2个数据库模式:S=<S1,S2,… ,Sn>,T=<T1,T2,… ,Tm>。其中:S1,S2, …,Sn表示源表模式的字段;T1,T2, …,Tm表示用基于自训练的自动模式映射方法寻找到的,与S1,S2, …,Sn一一映射语意相同的字段。
映射问题可以被视为分类问题,给定全局模式,试图给每个源表模式的字段Si分配 1个合适Tj,建立映射关系。设训练样本为为源模式的1个实例的属性,cj(j=1, 2, 3…,m)为该属性对应的字段名。
从过程上看,本方法包括3个阶段即训练阶段、自训练增强阶段、匹配阶段。在训练阶段,系统请求用户手工标定几个数据源与全局语意的映射关系;然后,系统从已标定的数据源中提取数据,形成训练样本;最后,系统采用基于adaboost的集成学习算法在训练样本上进行训练,得到分类器。在自训练增强阶段,系统从新加入的数据源中提取数据,形成测试样本;系统使用自训练算法,用测试样本中分类器预测确定度高的样本加入到训练集中,再次训练分类器,整个过程重复多次,得到增强后的分类器。在匹配阶段,系统将增强后的分类器应用到测试样本上,形成匹配结果。
2.2 训练阶段
训练阶段包括3步。
(1)系统请求用户手工标定几个数据源和全局语意的映射关系。
(2)系统从已标定的数据源中提取数据,形成训练样本。因为全局模式字段rsid对应dbsnp中字段B5,于是,从数据源dbsnp中提取1 000条记录中B5字段的值,对每个b5构造样本为<b5,rsid>。再取全局模式字段chrName,从数据源dbsnp中也提取1 000条记录中B2字段的值,也构造类似的样本。依此类推,共构造4 000条训练样本,其第1个属性为字段取值,类标注为全局模式字段名。
(3)系统在训练样本上训练分类器(AdaBoost)。采用 AdaBoost算法[18]对训练样本进行训练,其基准学习器采用朴素贝叶斯方法。AdaBoost是一种集成分类器,能够将多个弱分类器集成起来对样本的类标注进行分类判断。
2.3 自训练增强阶段
(1)系统从新加入的数据源中提取数据,形成测试样本;
新加入的数据源为Hapmap。其中共有17个字段:rs#, chrom, pos, strand, build, center, protLSID,assayLSID, panelLSID, QC_code, refallele, refallele_freq,refallele_count, otherallele, otherallele_freq, otherallele_count, totalcount。从hapmap数据中提取5 000条记录,对其每个字段都构造1条未标注样本,将这些样本组成测试集,则测试集中包含样本为17×5 000个样本,
(2)系统将训练得到的分类器在测试样本上进行自训练。 在测试样本集上采用自训练算法对分类器进行增强。其算法如下所示。
输入:已训练好的分类器L,训练样本集T,测试样本集U。
1. 重复
2. 使用f对U进行分类,获得U上的预测确定度;
3. 将U中预测确定度高于阈值ε的样本,组成一个集合S;
4. 将S从U中去除;
5. 对S中的样本加上L预测的标注,加入到训练样本集T中;
6. 使用adaboost算法在T上再次训练,得到更新后的分类器L′。
其中:预测确定度为使用分类器预测样本标注时预测概率最高的类标注的预测概率。
2.4 匹配阶段
使用自训练得到的学习器L对测试集中的所有样本进行测试。其中L测试得到的结果为这个结果构成了1个矩阵。对每一列计算取值,得分最多的类标注即为该列对应的全局模式字段。
3 实验结果与分析
针对语意映射进行了实验比较。实验比较的目的在于验证系统所采用的基于自训练的语意映射方法的有效性。实验平台为Windows XP操作系统,机器配置为Thinkpad X201。实验中基于weka开发了相关代码。实验测试的算法包括:基于单学习器(朴素贝叶斯算法)的传统监督学习算法、基于集成学习器(AdaBoost算法)的传统监督学习算法、基于集成学习器(AdaBoost算法)的图半监督学习算法和基于集成学习器(AdaBoost算法)的自训练学习算法。采用的评估指标为精度(P)和召回率(R)。精度为Ncorrect为正确的样本在结果中被正确判定的数量;Nwrong为错误的样本在结果中被误判定为正确的数量;Ndesign为正确样本的总数。
3.1 实验数据
在实验中全局模式为:编号 rsid、染色体名chrName、起始位置begin、终止位置end。
局部模式分别来自 hapmap中的 snp数据以及dbsnp中的snp数据,要将这些异构的snp模式映射到全局模式上。
Hapmap中1条典型的数据为:rs10399749, chr1,45162, +, ncbi_b36, perlegen, urn:lsid:perlegen.hapmap.org:Protocol:Genotyping_1.0.0:2, urn:lsid:perlegen.hapmap.org:Assay:25761.5318498:1, urn:lsid:dcc.hapmap.org:Panel:CEPH-30-trios:1, QC+, C, 1.000, 106, T, 0, 0,106。在其文档中,对各字段的名称分别标记为:rs#,chrom, pos, strand, build, center, protLSID, assayLSID,panelLSID, QC_code, refallele, refallele_freq, refallele_count, otherallele, otherallele_freq, otherallele_count,totalcount。
而dbsnp中的1条典型的snp数据信息为:585,chr1, 433, 433, rs56289060, 0, +, -, -, -, /C, genomic,insertion, unknown, 0, 0, unknown, between, 1, 将每个字段命名为B1,B2,…,B19。
测试数据来自 http://hgdownload.cse.ucsc.edu/goldenPath/hg19/database/的 snp数据库,其中1条典型的snp数据记录格式为:590, chr1, 754191, 754192,G, 1, 0, 8。将其每个字段命名为C1, C2, C3, C4, C5, C6,C7, C8。下面将C1, C2, C3, C4, C5, C6, C7和C8各自对应到全局模式中的1个字段。
3.2 实验流程
(1)确定hapmap中的snp数据和dbsnp中的snp数据字段同全局模式的关系。通过手工标注,确定hapmap的snp数据同dbsnp的snp数据与全局模式的字段对应关系见表2。
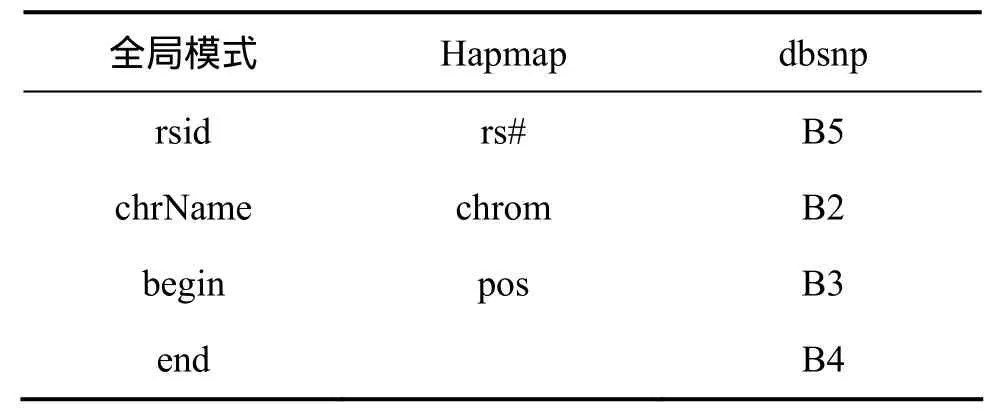
表2 Hapmap和dbsnp数据库中的snp数据与全局模式的字段对应关系Table 2 SNPs data in Hapmap and dbSNP databases and their corresponding relationship with fields of global model
(2)生成训练样本集。训练样本集中共随机抽取5 000个样本。样本生成的规则为将hapmap和dbsnp数据中对应全局模式字段的数据值作为特征值,将全局模式字段名作为类标注,构成1条样本。如hapmap中rs#对应全局模式的rsid,则任取hapmap中的1条记录,将其rs#字段的值作为特征值,再将rsid作为类标注,构成1条样本。共生成5 000条样本。
(3)用待测试的算法在训练集上进行训练。调用weka平台中的朴素贝叶斯算法和AdaBoost算法,在5 000条样本的训练集上进行训练,得到对应的分类器。
(4)在测试数据上测试各分类器的精度(Precision)和召回率(Recall)。
从测试数据中随机提取500条测试样本,这些测试样本以各字段的值作为特征值,类标注空缺。在这500条样本上测试朴素贝叶斯算法和AdaBoost算法生成的分类器,生成其预测的类标注。测试基于集成学习器(AdaBoost算法)的图半监督学习算法时,采用高斯核的方法确定图中的边权值,即:x为样本,利用 Zhu[19]提出的一种启发式方法确定)。该方法利用Kruskal最小生成树算法首先要对边从小到大排序,再在生成所有点的最小生成树过程中,找到第1条连接2个分别含有不同类标注的分支的边,然后,设这条边的长度为d0。d0被认为是类间距离,取σ=d0/3。这样,使得当2点距离为d0时,其相似度约为0。测试采用集成学习器(AdaBoost算法)的自训练学习算法时,从测试数据中随机提取5 000条测试样本,然后,使用自训练算法更新分类器,迭代20次,确定度阈值为0.75。将测试数据中同一字段对应的样本按不同类标注计数,计数最多的类标注即为测试数据对应字段所应当映射到的全局模式字段名。
3.3 结果分析
4种学习器经过学习后的精度和召回率,均为20次运算的平均值,实验结果如表3所示。

表3 4种学习器的精度和召回率测试结果对比Table 3 Comparison of precision and recall of four kinds of machine learning
由表 3可以看出:后 3种集成学习器(AdaBoost算法)的学习算法与单学习器的传统监督学习算法相比,在不同程度上都提高了精度和召回率,其中,集成学习器(AdaBoost算法)的自训练学习算法实现简单,时间代价较小,加入自训练学习算法的集成学习器无论是精度还是召回率都较高。
单学习器的传统监督学习算法精度达到 73.5%,召回率达到 84.1%,精度和召回率均比集成学习算法的低。集成学习器(AdaBoost算法)的传统监督学习算法的精度达到87.2%,比单学习器的精度提高13.7%;其召回率达到91.7%,比单学习器的召回率提高7.6%。
2种集成学习器(AdaBoost算法)的半监督学习算法的精度和召回率与前2种算法相比均有明显提高。这种基于自训练的集成学习器其精度达到 94.2%,比集成学习器的传统监督学习算法的精度又进一步提高7%;其召回率达到97.5%,比传统监督学习算法的召回率又进一步提高5.8%。这是由于在自训练过程中,未标注样本的分布信息提供了分类边界的位置信息,从而有效地增强了分类器的泛化精度。在集成学习器(AdaBoost算法)的图半监督学习算法中,参数选择对于算法性能有重要作用,当前研究中并无一般的成熟方法确定最优参数,这使其性能受到限制,虽然达到了 92.2%的精度和 94.6%的召回率,但与集成学习器的自训练学习算法的测试结果精度和召回率相比仍分别有2%和2.9%的差距。
4 结论
(1)通过对相关权威生物信息数据库进行分析和处理,形成支持GWAS未覆盖基因组区研究的统一接口,大大减少了数据量的存储、处理和访问,避免了相关研究中对海量数据的访问和处理。
(2)通过实验测试的3种算法即单学习器(朴素贝叶斯算法)的传统监督学习算法、集成学习器(AdaBoost算法)的传统监督学习算法和集成学习器(AdaBoost算法)的自训练学习算法的比较,基于自训练的集成学习器在精度和召回率上比前3种算法都有提高。
(3)采用自训练的半监督机器学习实现语意映射,分类器可以在未标注样本的分布信息中提取隐含的分类边界信息,从而使泛化能力得到增强,更加准确地预测语意映射关系,大大减少了人工参与量,从而实现对整个 GWAS未覆盖基因组区数据的快捷有效的数据集成。
[1]International HapMap Consortium. A second generation human haplotype map of over 3.1 million SNPs[J]. Nature, 2007,449(7164): 851-861.
[2]Wall J D, Pritchard J K. Using haplotype blocks to map human complex trait loci[J]. Trends in Genetics, 2003, 19(3): 135-140.
[3]Barrett T, Troup D B, Wilhite S E, et al. NCBI GEO: Archive for functional genomics data sets: 10 years on[J]. Nucleic Acids Res,2011, 39(1): 1005-1010.
[4]Croft D, O'Kelly G, Wu G, et al. Reactome: A database of reactions,pathways and biological processes[J]. Nucleic Acids Res, 2011, 39(1): 691-697.
[5]Keseler I M, Collado-Vides J, Santos-Zavaleta A, et al. EcoCyc:A comprehensive database of Escherichia coli biology[J].Nucleic Acids Res, 2011, 39(1): 583-590.
[6]Mardis E R. The $1,000 genome, the $100,000 analysis?[J].Genome Med, 2010, 2(11): 84.
[7]Manconi A, Rodriguez-Tom`e P. A survey on integrating data in bioinformatics[C]//Learning Structure and Schemas from Documents. Biba M, Xhafa F, eds. Computational Intelligence,2011, 375: 413-432.
[8]韩建文, 张学军. 全基因组关联研究现状[J]. 遗传, 2011,33(1): 25-35.HAN Jianwen, ZHANG Xuejun. Current status of genome-wide association study[J]. Hereditas, 2011, 33(1): 25-35.
[9]权晟, 张学军. 全基因组关联研究的深度分析策略[J]. 遗传,2011, 33(2): 100-108.QUAN Cheng, ZHANG Xuejun. Research strategies for the next step of genome-wide association study[J]. Hereditas, 2011, 33(2):100-108.
[10]凃欣, 石立松, 汪樊, 等. 全基因组关联分析的进展与反思[J].生理科学进展, 2010, 41(2): 87-94.TU Xin, SHI Lisong, WANG Fan, et al. Genomewide association study:advances, challenges and deliberation[J].Progress In Physiological Sciences, 2010, 41(2): 87-94.
[11]Cohen W, Richman J. Learning to match and cluster large high dimensional data sets for data integration[C]//Proceedings of ACM SIGKDD. Edmonton, Canada, 2002: 475-480.
[12]Berlin J, Motro A. Database schema matching using machine learning with feature selection[C]//Proceedings of CAISE.Toronto, Canada, 2002: 452-466.
[13]Yarowsky D. Unsupervised word sense disambiguation rivaling supervised methods[C]//Proceedings of the 33rd Annual Meeting of the Association for Computational Linguistics. Cambridge,Massachusetts, 1995: 189-196.
[14]Riloff E, Wiebe J, Wilson T. Learning subjective nouns using ex-traction pattern bootstrapping[C]//Proceedings of the Seventh Conference on Natural Language Learning at HLT-NAACL. NJ,USA, 2003: 25-32.
[15]KachitesMccallum A, Mitchell T. Text classification from labeled and unlabeled documents using EM[J]. Machine Learning, 2000, 39(2/3): 103-134.
[16]Joachims T. Transductive inference for text classification using support vector machines[M]. San Francisco, USA: Morgan Kaufmann, 1999: 200-209.
[17]Belkin M, Niyogi P. Semi-supervised learning on riemannian manifolds[J]. Machine Learning, 2004, 56(1/2/3): 209-239.
[18]Yoav F, Robert E, Schapire. Experiments with a new boosting algorithm[C]//Machine Learning:Proceedings of the 13th International Conference. San Francisco, 1996: 148-156.
[19]Zhu X. Semi-supervised learning with graphs[D]. Pittsburgh:Carnegie Mellon University, 2005: 5-6.
