6种鱼mtDNA D-loop及其邻近区段的序列比较分析
2013-07-07郝君杨蔷鲍迪梁爱军张兴华董仕
郝君,杨蔷,鲍迪,梁爱军,张兴华,董仕
(天津师范大学生命科学学院天津市动植物抗性重点实验室,天津300387)
6种鱼mtDNA D-loop及其邻近区段的序列比较分析
郝君,杨蔷,鲍迪,梁爱军,张兴华,董仕
(天津师范大学生命科学学院天津市动植物抗性重点实验室,天津300387)
采用PCR技术对乌克兰鳞鲤Cyprinus carpio、鲫Carassis auratus、鲢Hypophthalmichthys molitrix、鳙Aristichthys nobilis、草鱼Ctenopharyngodon idellus和乌苏里拟鲿Pseudobagrus ussuriensis共28个个体(分属于天津市动植物抗性重点实验室应用PCR-RFLP方法已检测出的不同单倍型)的mtDNA D-loop及其邻近区段进行扩增并测序,获得了大小为1 500~1 800 bp的扩增产物。用Clustal 1.83软件与GenBank中的鲤、金鱼、鲢、草鱼、长吻鮠5种鱼的mtDNA全序列进行排序比较,确定扩增产物包含完整的tRNAPro、D-loop、tRNAPhe序列以及12S rRNA长度约为400 bp的部分序列。将6种鱼共28个样本的序列提交GenBank后获得序列号为KC292921~KC292948。运用Mega 4.0软件计算出6种鱼的碱基组成和碱基差异,基于Kimura双参数模型计算种间的遗传距离,并与GenBank中5种鱼的mtDNA序列一起构建NJ系统树。序列结构分析表明,在6种鱼序列的4个区段中,D-loop区段在种内、种间的差异性均高于另外3个区段。6种鱼NJ系统树的结果与传统分类方法一致,可为鱼类分类以及种类鉴定提供科学依据。另外,6种鱼各种单倍型均在所测得序列中找到相应的酶切位点,印证了RFLP实验结果,在酶切位点以外也发现了同种鱼的不同单倍型间存在碱基差异。
乌克兰鳞鲤;鲫;鲢;鳙;草鱼;乌苏里拟鲿;D-loop;NJ系统树
鱼类线粒体DNA(Mitochondrial DNA,mtDNA)是一种环状共价闭合的双链超螺旋分子,为核外遗传物质。具有严格的母系遗传、结构简单、分子量小、无组织特异性和进化速率快等特点,被广泛应用于物种起源、遗传分化、种内种间系统发生关系及原种鉴定等方面[1-9]。
D-loop区是mtDNA上最重要的非编码区,受进化压力小,变异程度高,进化速率是mtDNA其他区段的2~5倍。D-loop区位于 tRNAPro和 tRNAPhe之间,包含与DNA复制和转录相关的序列,是分析相近物种种间和种内遗传多样性的有效分子遗传标记,也是近年来 mtDNA 的研究热点[6-7,10-12]。在测序技术未被广泛应用于mtDNA研究之前,对鱼类的种群结构、物种多样性方面的研究大多基于RFLP分析[13-14]。随着PCR技术以及自动化测序技术的发展,已有许多研究人员将D-loop区作为分子标记应用于鱼类遗传分化的研究[1-7]。
鲤Cyprinus carpio与鲫Carassis auratus是中国最重要的淡水养殖鱼类,在世界范围内广泛养殖,历史悠久,并形成许多亚种和品系,鲤品种之一的乌克兰鳞鲤的养殖已产生了巨大的经济效益[15];鲢Hypophthalmichthys molitrix、鳙Aristichthys nobilis和草鱼Ctenopharyngodon idellus是中国特产鱼类,与青鱼 Mylopharyngodon piceus一起被誉为中国“四大家鱼”,是世界水产养殖业发展的重要养殖种类[16-19];乌苏里拟鲿 Pseudobagrus ussuriensis主要分布于中国黑龙江水系,其营养丰富,肉质鲜美,具有很高的经济价值[20-21]。本研究中,作者选用天津市动植物抗性重点实验室进行过mtDNA D-loop区PCR-RFLP分析的6种鱼的部分个体为试验材料,对其D-loop及其邻近区段进行PCR扩增和序列测定,对测序结果进行种内、种间遗传结构差异分析,构建系统树,探讨D-loop及其邻近区段作为分子标记对系统分化研究的意义。同时对6种鱼PCR扩增区段的序列结构与PCR-RFLP检测结果进行比较,旨在为鱼类分类以及种类鉴定提供科学依据。
1 材料与方法
1.1 材料
试验用乌克兰鳞鲤2尾(每种单倍型各1尾)[15],鲫3尾(采自子牙河天津段)[22],鲢12尾(每种单倍型各1尾)[23],鳙7尾(每种单倍型各1尾)[24],草鱼2尾(每种单倍型各1尾)[25],乌苏里拟鲿2尾(采自天津市天祥水产养殖场)。
1.2 方法
1.2.1 基因组DNA的提取 从每尾鲜活试验鱼体上剪下大小约1.0 cm×0.5 cm的尾鳍鳍条,用苯酚-氯仿抽提法提取,于4℃下保存备用[26]。
1.2.2 mtDNA的D-loop及其邻近区段的扩增及凝胶电泳 以提取的 DNA为模板,使用位于mtDNA的D-1oop区段两侧的引物对每尾试验鱼的D-loop及其邻近区段进行扩增。所用引物为L15923:5′TTA AAG CAT CGG TCT TGT AA 3′[27]和H1067:5′ATA GTG GGG TAT CTA ATC CCA GTT 3′[28],两个引物的结合位点分别位于tRNAThr和12S rRNA编码区上。PCR方法与扩增片段的确认参照梁爱军等[15]的方法。将PCR产物委托深圳华大基因科技有限公司进行测序。测序得到的序列依据峰图进行确认。
1.3 数据处理
以从GenBank中获得的鲤序列(X61010.1)、金鱼序列(AB111951.1)、鲢序列(EU315941.1)、草 鱼 序 列 (EU391390.1) 和 长 吻 鮠 序 列(GU596454.1)为参考序列,用Clustal 1.83软件对测序结果进行同源序列比对分析[29],确定PCR扩增产物所包含的各个区段。将比对好的6种鱼28个个体的序列提交到GenBank库中获得序列号。用Mega 4.0软件统计6种鱼D-loop及其邻近区段的碱基组成和碱基差异[30],基于Kimura-双参数计算6种鱼群体间的遗传距离[31],并依此与Gen-Bank中5种鱼的序列一起构建NJ系统树。
2 结果
2.1 mtDNA D-loop及其邻近区段的PCR扩增
6种鱼mtDNA D-loop及其邻近区段的PCR扩增产物经琼脂糖凝胶电泳后,得到大小不同的扩增片段(图1)。PCR产物电泳结果显示条带清晰单一,未发现非特异性条带。其中1号草鱼个体的片段长度约为1 800 bp,12、13号乌苏里拟鲿两个个体的片段长度约为1 500 bp,其他个体的片段长度约为1 600 bp。

图1 6种鱼mtDNA D-loop及其邻近区段的PCR电泳图Fig.1 The PCR electrophoretogram of mtDNA D-loop and adjacent regions in six fish species
2.2 PCR产物序列分析
将测序结果与GenBank中的序列进行比对后发现,6种鱼使用引物L15923和H1067扩增出的片段包括完整的tRNAPro、D-loop、tRNAPhe序列以及12S rRNA长度约为400 bp的部分序列。将6种鱼共28个个体的序列提交到GenBank中,获得的序列号为KC292921~KC292948。确定了每种鱼D-loop的位置以及片段长度。其中,鲤D-loop区段的长度为926~929 bp,鲫为925 bp,草鱼为928 bp(GenBank序列号为KC292921的1号草鱼个体的69号碱基插入了一段长度为231 bp的重复序列,其中包括草鱼D-loop区段前部的203个碱基和tRNAPro尾部的28个碱基),鲢为933~938 bp,鳙为935~939 bp,乌苏里拟鲿为893 bp。
2.3 6种鱼序列的碱基组成与差异
2.3.1 碱基组成 用Mega 4.0计算出6种鱼的D-loop区段及扩增所得全部序列的碱基频率(表1)。每种鱼的A+T含量都明显高于G+C含量,符合mtDNA的特点,表明6种鱼的碱基组成具有明显偏向性[32-33]。
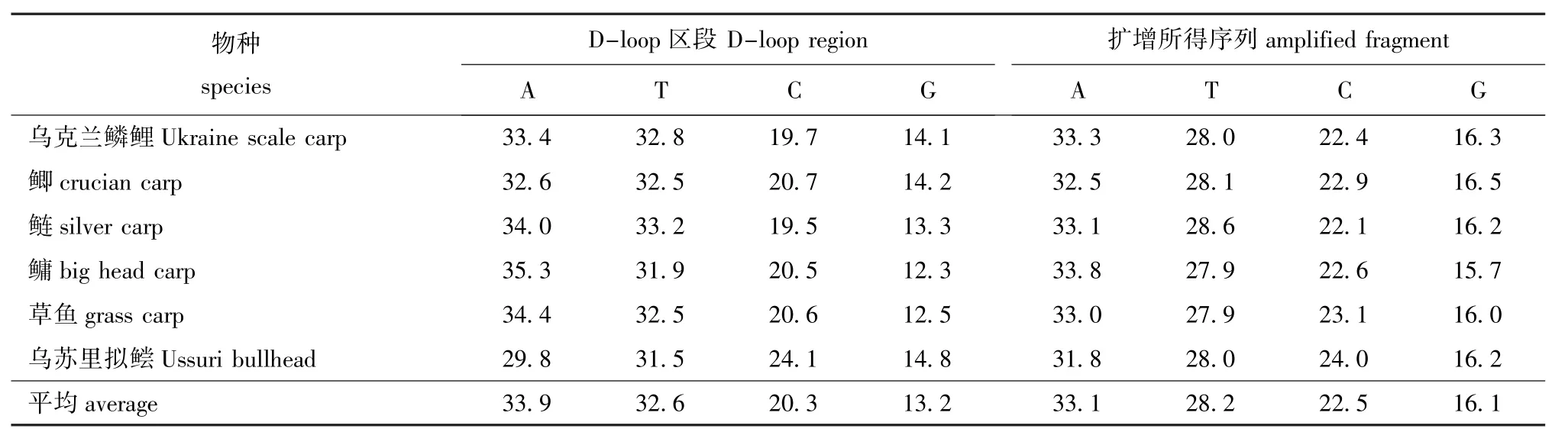
表1 6种鱼D-loop区段及扩增所得序列的碱基频率Tab.1 Nucleotide frequencies in D-loop region and amplified fragment in six fish species %
2.3.2 种内和种间的碱基差异 将6种鱼进行排序比较后发现,6种鱼序列结构的差异性表现在种间和种内两方面,种间的差异性远大于种内。而且6种鱼的tRNAPro、tRNAPhe和12S rRNA三个区段的两种差异性均远小于D-loop区段(表2)。不同种间在D-loop区段的起始端就表现出明显的碱基差异,与其他5种鱼亲缘关系较远的乌苏里拟鲿的差别性更加明显。
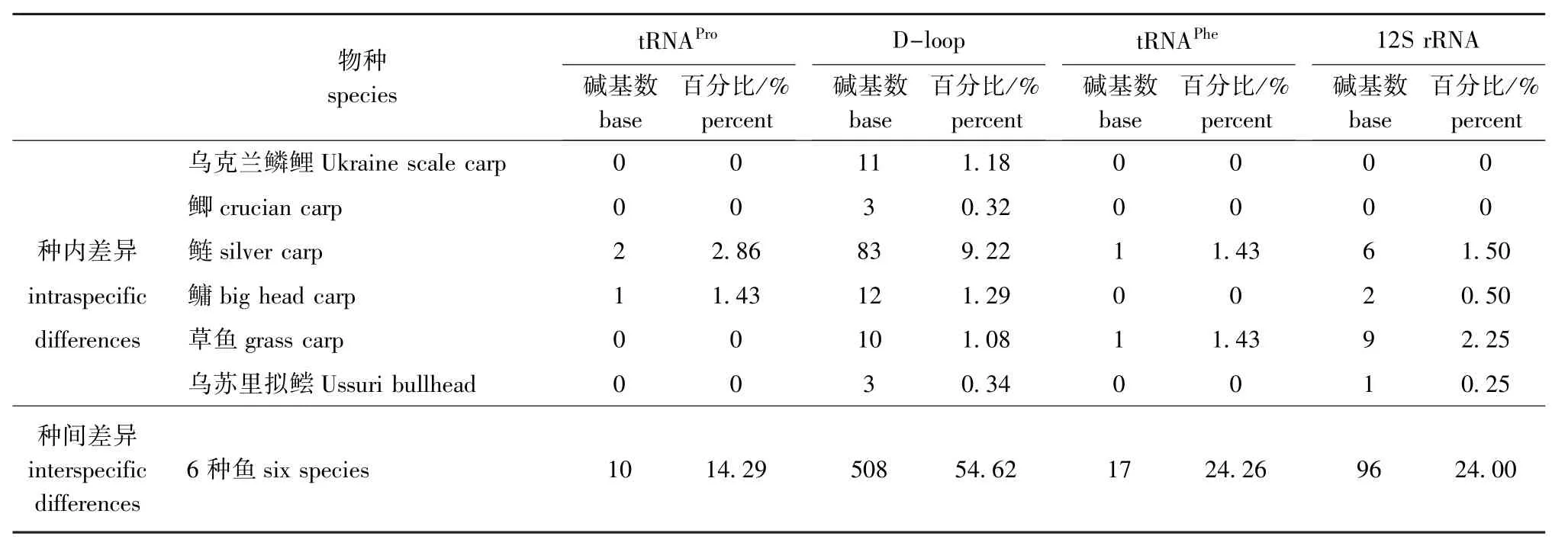
表2 6种鱼的扩增片段中种内和种间的碱基差异Tab.2 The base difference in amplified fragment between intraspecies and interspecies
2.3.3 PCR-RFLP酶切结果与序列分析的比较
使用相同引物对6种鱼的mtDNAD-loop及其邻近区段进行了RFLP分析[15,22-25](乌苏里拟鲿的结果未发表),在序列分析结果中均找到了RFLP的相应酶切位点,印证了RFLP实验结果。同时在酶切位点以外也发现了同种鱼的不同单倍型间存在碱基差异(GenBank号:KC292921~KC292948)。
2.4 6种鱼的遗传距离与系统发育树
2.4.1 遗传距离 基于 Kimura双参数法(Kimura's 2-Parameter)计算6种鱼种间D-loop区段的遗传距离(表3)和扩增所得全部序列的遗传距离(表4)。其中,鳙与乌苏里拟鲿的遗传距离最大,为0.534668;鲢与鳙的遗传距离最小,为0.106186。

表3 6种鱼D-loop区段种间的平均Kimura's 2-Parameter遗传距离Tab.3 The genetic distance of D-loop in six species based on Kimura's 2-Parameter model
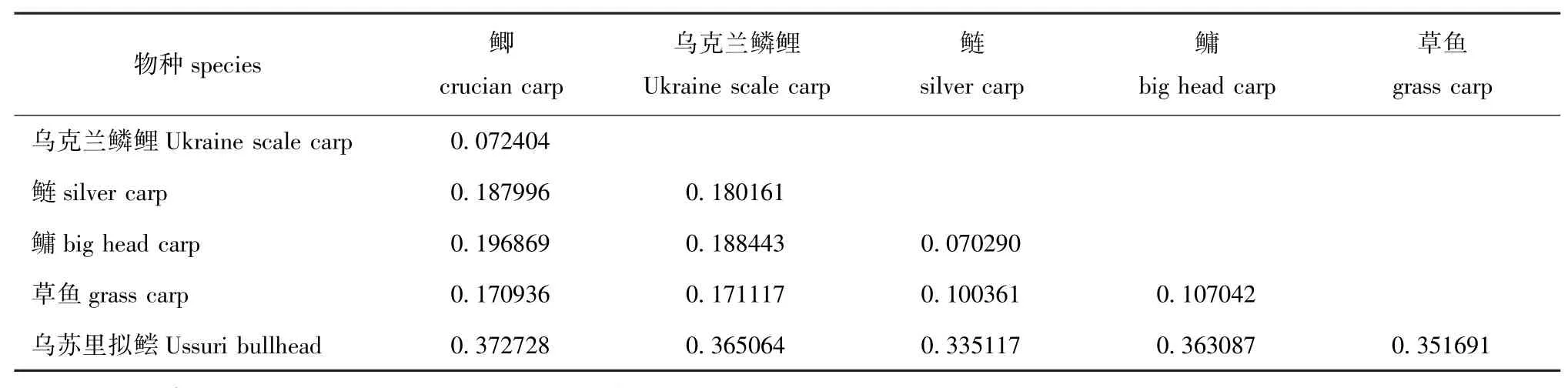
表4 6种鱼扩增所得序列中种间的平均Kimura's 2-Parameter遗传距离Tab.4 The genetic distance of amplified fragment in six species based on Kimura's-Parameter model
2.4.2 系统发育树 运用Mega 4.0软件,基于Kimura双参数模型,进行1 000次抽样重复检测,将对6种鱼测序所得片段与GenBank中获得的鲤序列(X61010.1)、金鱼序列(AB111951.1)、鲢序列(EU315941.1)、草鱼序列(EU391390.1)和长吻鮠序列(GU596454.1)构建NJ系统树,如图2所示。结果显示,除鲢分支的支持率为85%外,其余均在90%以上,并可明显分出6个分支。
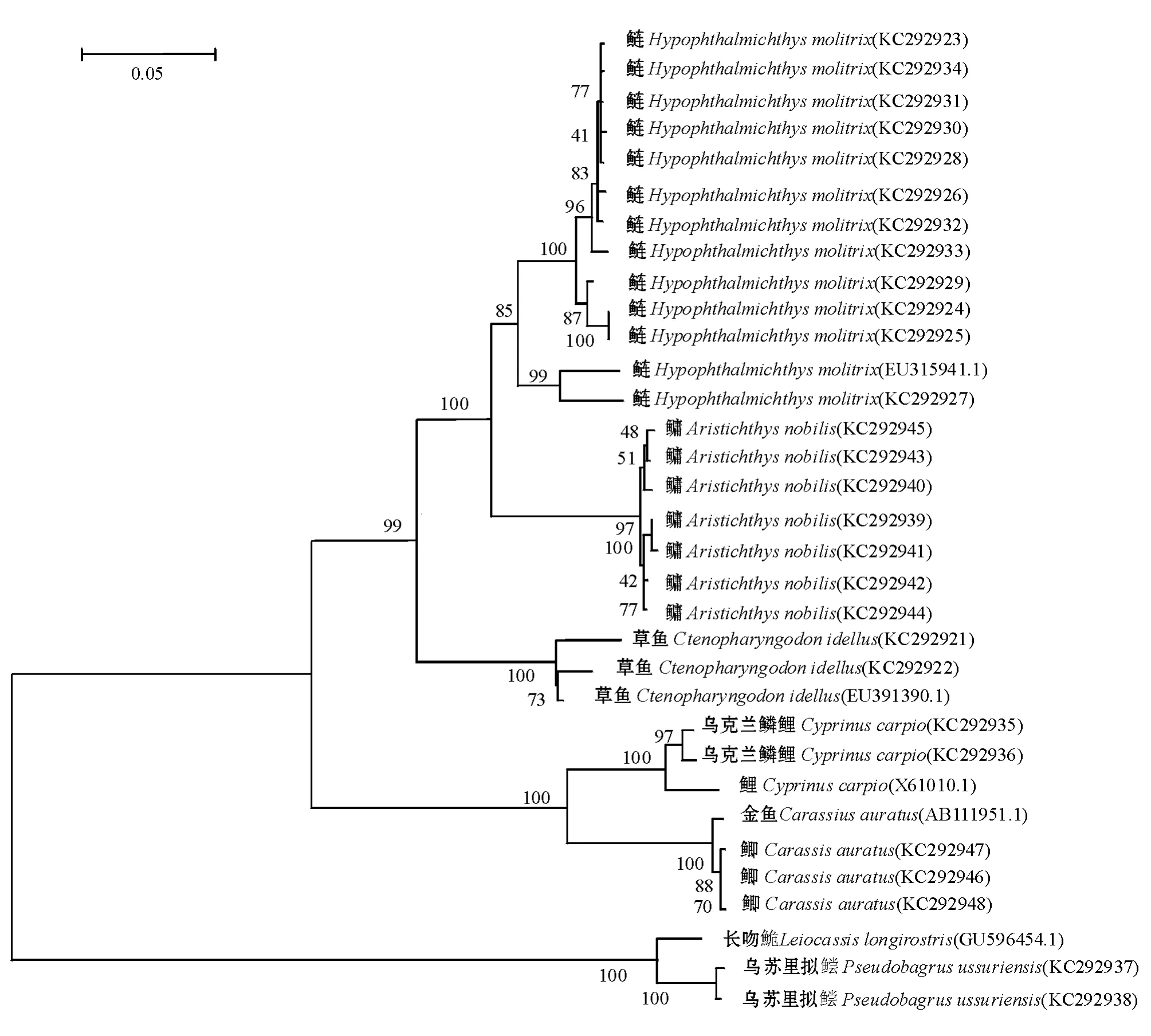
图2 基于Kimura's 2-Parameter模型构建的PCR扩增片段的NJ系统树Fig.2 NJ phylogenetic tree of amplified fragment based on the Kimura's 2-Parameter model
3 讨论
近年来,对鱼类mtDNA的研究逐步向序列分析的方向发展,序列遗传多样性分析及结构研究已成为D-loop区的研究重点。本试验中运用软件对6种鱼的mtDNAtRNAPro、D-loop、tRNAPhe和部分12S rRNA序列进行分析,得到6种鱼碱基含量、碱基差异、遗传距离和NJ系统树等相关数据。
与GenBank中5种鱼的同源序列比对后,确定了6种鱼的PCR产物中所包含的各个区段以及mtDNA D-loop区的位置和片段长度。发现6种鱼D-loop区的长度在种内和种间都存在差异。吴海防等[25]报道的草鱼单倍型Ⅱ比一般草鱼长出一段DNA为231 bp的重复序列,基于mtDNA D-loop区段用PCR-RFLP法检测出的草鱼两种单倍型的差异源于此重复序列。
本研究中选取了除鲫之外的5种鱼mtDNA D-loop及其邻近区段经PCR-RFLP法检测的单倍型,经过序列分析,印证了每种鱼的不同单倍型在限制酶酶切位点上的差异性,并且除酶切位点以外也存在碱基差异。经过酶切位点比对,发现单酶切之后会出现一些较小的酶切片段,这些片段在使用琼脂糖凝胶电泳分辨时受到限制,因而可以认为,在很多用PCR-RFLP检测分析的报道中酶切片段长度之和会小于 PCR 扩增的片段长度[13-14,23-25]。与PCR-RFLP法相比,序列分析的方法精确度更高。
6种鱼序列差异性的研究证明了mtDNA的D-loop区段的变异程度比其他序列高。在6种鱼的NJ系统树中,亲缘关系较近的鲢和鳙,鲤和鲫都可以明显的进行区分,并且具有较高的支持率。本研究结果表明,D-loop区段或者D-loop区段结合其邻近区段适合系统发育研究,并且可以为鱼类种类识别以及遗传多样性分析等方面提供科学依据。
[1]肖武汉,张亚平.鱼类线粒体DNA的遗传与进化[J].水生生物学报,2000,24(4):384-391.
[2]杨明生,熊邦喜.动物mtDNA的研究及在鱼类生态学中的应用[J].孝感学院学报,2005,25(3):23-27.
[3]汪泰初,刘朝良,肖林珍.线粒体基因组(mtDNA)的研究进展[J].安徽农业科学,2006,34(10):2068,2071.
[4]潘宝平,卜文俊.线粒体基因组的遗传与进化研究进展[J].生物学通报,2005,40(8):1-3.
[5]邹习俊,韩雪,韩虎峰.鱼类mtDNA及其非编码区的研究概况[J].贵州畜牧兽医,2009,33(3):23-25.
[6]徐钢春,魏广莲,李建林,等.基于线粒体DNA D-loop序列分析养殖刀鲚与湖鲚的遗传多样性[J].大连海洋大学学报, 2012,27(5):448-452.
[7]朱雪莲,王志勇,韩志强.借助mtDNA控制区序列分析金鱼与不同地域鲫的亲缘关系[J].大连水产学院学报,2010,25(2): 153-157.
[8]黄立群,仪慧兰,崔松林,等.历山中国大鲵线粒体片段序列的测定及其遗传差异研究[J].大连海洋大学学报,2012,27(6): 513-517.
[9]程起群,马春艳,缪炯,等.基于线粒体12Sr RNA序列研究凤鲚两个野生群体的遗传多样性[J].大连水产学院学报,2007, 22(5):387-391.
[10]Rosel P E,Dizon A E,Haygood M G.Variability of the mitochondrial control region in population of the harbour porpoise,Phocoena phocoena,on interoceanic and regional scales[J].Can J Fish Aquat Sci,1995,52:1421-1429.
[11]Rosel P E,Haygood M G,Perrin W F.Phylogenetic relationship among the true porpoise(Cetacean:Phocoenidae)[J].Molecular Phylogenetics and Evolution,1995,4(4):463-474.
[12]Zhu D,Jamieson B G,Hugall A,et al.Sequence evolution and phylogenetic signal in control region and cytochrome b sequences of rainbow fishes(Melanotaeniidae)[J].Mol Biol Evol,1994,11: 672-683.
[13]Sugaya T,Ikeda M,Taniguchi N.Typing of mtDNA in the kuruma prawn based on the PCR-RFLP analysis of mtDNA control region [J].Fish Genetics and Breeding Science,2001,31:11-15.
[14]Ikeda M,Taniguchi N.Genetic variation and divergence in populations of ayu Plecoglossus altivelis,including endangered subspecies,inferred from PCR-RFLP analysis of the mitochondrial DNA D-loop region[J].Fisheries Science,2002,68:18-26.
[15]梁爱军,王淞,鲍迪,等.乌克兰鳞鲤同工酶及mtDNA D-loop区段的RFLP分析[J].安徽农业科学,2011,39(27):17109-17113.
[16]李思发,吴力钊,王强,等.长江、珠江、黑龙江鲢、鳙、草鱼种质资源研究[M].上海:上海科学技术出版社,1990:28-39.
[17]宋平,李小迎,熊全沫.鲢鳙线粒体DNA的九种限制性内切酶酶切图谱的比较[J].水产学报,1994,18(3):221-230.
[18]张锡元,张德春,杨代淑,等.长江鲢遗传多样性的随机扩增多态DNA分析[J].水产学报,1999,23(增刊):7-14.
[19]沈玉帮,张俊彬,李家乐.草鱼种质资源研究进展[J].中国农学通报,2011,27(7):369-373.
[20]潘伟志,陈军,王鹏,等.乌苏里拟鲿人工繁育技术研究[J].水产学杂志,2006,19(1):31-35.
[21]陈军,潘伟志,赵春刚,等.乌苏里拟鲿苗种培养技术[J].黑龙江水产,2006(3):13-17.
[22]刘琳.应用DNA及同工酶遗传标记检测分析二、三倍体鲫鱼的遗传结构[D].天津:天津师范大学,2008.
[23]王淞,曹晓霞,谷口顺彦,等.4个群体鲢mtDNA D-loop的PCR-RFLP分析[J].淡水渔业,2010,40(4):3-9.
[24]单淇,董仕,吴海防,等.3个群体鳙鱼mtDNA D-loop区段的限制性片段长度多态性分析[J].中国水产科学,2006,13(2): 174-180.
[25]吴海防,董仕,单淇,等.3个群体草鱼mtDNA D-Loop的PCR-RFLP分析[J].水产科学,2006,25(4):184-188.
[26]Perez-Enriquze R,Taniguchi N.Genetic structure of red sea bream(Pagrus major)population of Japan and the Southwest Pacific,using microsatellite DNA markers[J].Fisheries Science, 1999,65(1):23-30.
[27]Iguchi K,Tanimura Y,Nishida M.Sequence divergence in the mtDNA control region of amphidromous and landlocked forms of ayu[J].Fisheries Science,1997,63:901-905.
[28]Takahashi Y,Kobayashi T,Ueda T.Genetical variation of the D-loop region in mitochondrial DNA using PCR method in metropolitan bitterling[J].Fish Genetics and Breeding Science,1994,20: 39-45.
[29]Thompson J D,Gibson T J,Plewniak F,et al.The Clustal X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J].Nucleic Acids Research,1997, 25(24):4876-4882.
[30]Kumar S,Tamura K,Nei M.MEGA3:integrated software for molecular evolutionary genetics analysis and sequence alignment[J]. Brief Bioinform,2004,5(2):150-163.
[31]李彬彬,黄培春,钟复光.生物学软件在线粒体DNA序列多态性分析中的应用[J].生物信息学,2010,8(2):153-155.
[32]张方,米志勇,毛钟荣,等.草鱼(Ctenopharyngodon idellus)线粒体DNA控制区及其两侧tRNA基因的克隆与结构分析[J].中国生物化学与分子生物学报,1999,15(3):417-422.
[33]Broughton R E,Roe B A.The complete sequence of the zebra fish(Danio rerio)mitochondrial genome and evolutionary patterns in vertebrate mitochondrial DNA[J].Genome Res,2001,11(11): 1958-1967.
The sequence comparison of mtDNA D-loop and adjacent regions in six fish species
HAO Jun,YANG Qiang,BAO Di,LIANG Ai-jun,ZHANG Xing-hua,DONG Shi
(College of Life Science,Tianjin Normal University,Tianjin Key Laboratory of Animal and Plant Resistance,Tianjin 300387,China)
The mtDNA D-loop and its adjacent regions were amplified and sequenced in 28 individuals from different haplotypes detected using PCR-RFLP in Ukraine scale carp(Cyprinus carpio),crucian carp(Carassius auratus),silver carp(Hypophthalmichthys molitrix),big head carp(Aristichthys nobilis),grass carp(Ctenopharyngodon idellus)and Ussuri bullhead(Pseudobgrus ussuriensis)by PCR.The amplified fragment length of mtDNA D-loop and its adjacent regions in the fish were varied from 1 500 bp to 1 800 bp,and are compared with the mtDNA complete sequences of five fish species from GenBank by Clustal 1.83 software.It was found that the amplified fragment was comprised of complete tRNAPro,D-loop,tRNAPhesequences and about 400 bp part of the 12S rRNA sequence.Sequences of 28 individuals were submitted to GenBank for serial numbers(KC292921-KC292948), and the base composition and difference of the six species,and the genetic distance were calculated based on Kimura's 2-Parameter model and NJ phylogenetic tree with five sequences from GenBank was constructed by Mega 4.0 software.The sequence structure analysis showed that the intraspecific and interspecific differences in D-loop region were higher in the four region sequences of six species than those in other species.NJ phylogenetic trees of six species were consistent with the traditional classification methods,showing useful for fish species classification and identification.In addition,restriction sites of all kinds of haplotypes were found in sequences,which is consistent with PCR-RFLP experimental results.More variation out of the restriction sites were observed compared with RFLP analysis.
Ukraine scale carp;crucian carp;silver carp;big head carp;grass carp;Ussuri bullhead;D-loop; NJ phylogenetic tree
S965.113;Q953
A
2012-12-13
天津市动植物抗性重点实验室开放研究基金资助项目
郝君(1987-),女,硕士研究生。E-mail:haohaojunjun2645@163.com
董仕(1959-),男,博士,研究员。E-mail:skyds@mail.tjnu.edu.cn
2095-1388(2013)02-0160-06
