小黑杨bHLH转录因子基因和启动子的克隆与分析
2013-04-03梁楠松李蕾蕾詹亚光曾凡锁
梁楠松 ,周 姗 ,李蕾蕾 ,詹亚光 ,b,曾凡锁 ,b,李 博
(东北林业大学a.生命科学学院;b.林木遗传育种与生物技术国家重点实验室,黑龙江 哈尔滨 150040)
多细胞生物的正常生长依赖于维持细胞增殖和分化的适当平衡,破坏这个平衡会导致器官的生长和发育异常。bHLH(basic helix-loop-helix)转录因子是真核生物的生长发育过程中的一类重要转录因子,构成了真核生物蛋白质中的一个大家族,其成员在生物的生长发育过程中起着极为重要的调控作用[1]。植物bHLH转录因子,在植物生长发育、胁迫应答和植物次生代谢中起到重要作用[2-7]。例如,bHLH可以参与植物的生长发育和形态建成,转录调控复合体(MYB-bHLH-WD40)可以调控表皮毛和根毛的发育[8-9];bHLH转录因子还参与了胁迫应答作用,Kiribuchi等报道外源JA诱导水稻bHLH转录因子RERJ1基因在营养生长期的叶片、叶鞘和根中表达,并且RERJ1 基因在伤害和干旱胁迫下的叶片中上调表达[10];bHLH转录因子对植物次生物质代谢和花色素生物合成等方面也起着重要的调节作用,研究表明,含有bHLH结构域的转录因子R基因的产物Lc参与调控植物多个组织花青素的合成,对花青素合成的时间、分布和数量具有决定性作用[11]。
杨树在全世界广为栽植,也是我国主要用材树种之一。小黑杨(Populus×xiaoheiT.S.Hwang et Liang)在我国大部分地区均有种植,具有生长快、适应能力强的特点,有着较强的抗寒、抗旱、耐瘠薄、耐盐碱等生物学特性。其材质细密,色白,心材不明显,是良好的造纸用材,并在民用木材方面有着重要作用。小黑杨在黑龙江等干旱、寒冷地区长势较好,是我国北方荒沙、干旱地区优良的速生绿化树种[12-13]。
本研究中获得的2个bHLH转录因子PxbHLH01和PxbHLH02与拟南芥转录调控因子bHLH UPBEAT1(UPB1)具有较高的同源性,而后者在拟南芥的活性氧代谢以及调节植物根部细胞增殖与分化过程中起到了重要作用,UPB1通过直接调控一组氧化物酶来调控细胞增殖和分化之间的平衡[14]。笔者认为PxbHLH01和PxbHLH02也可能在这些方面具有重要的调控作用。为给小黑杨PxbHLH01和PxbHLH02基因功能的研究提供参考,对小黑杨PxbHLH01和PxbHLH02转录因子基因的编码区和启动子区进行了克隆,同时进行了详细的生物信息学分析。
1 材料与方法
1.1 材 料
1.1.1 取 样
小 黑 杨(Populus×xiaoheiT.S.Hwang et Liang)取材于东北林业大学实验林场,取小黑杨新鲜叶片,液氮冻存后使用。
1.1.2 试剂与载体
Primer star Taq酶、凝胶回收试剂盒、T4 DNA连接酶、JM109感受态细胞、pMD18-T载体,购自于宝生物工程(大连)有限公司,扩增引物由生工生物工程(上海)股份有限公司合成。
1.2 方 法
1.2.1 PxbHLH01和PxbHLH02基因编码区全长的克隆
以小黑杨新鲜叶片作为材料,应用CTAB法提取总RNA。应用反转录试剂盒将RNA反转录为cDNA。参照毛果杨基因组序列[15],对PxbHLH01和PxbHLH02基因分别设计特异引物(见表1),以小黑杨叶片cDNA为模板,进行PCR扩增。PCR扩增程序:94 ℃预变性3 min;94 ℃变性45 s;55 ℃退火45 s;72 ℃延伸90 s;共30个循环;72 ℃延伸10 min。应用凝胶回收试剂盒对特异性扩增产物回收纯化,纯化产物与pMD18-T载体连接,转化克隆进入JM109感受态细胞,送生工生物工程(上海)股份有限公司测序。

表1 克隆PxbHLH01和PxbHLH02基因编码区对应引物Table 1 The primer used for cloning the coding regions in PxbHLH01 and PxbHLH02 genes
1.2.2 PxbHLH01和PxbHLH02启动子的克隆
以小黑杨新鲜叶片作为材料,应用CTAB法提取总DNA。参照毛果杨基因组序列[15],对PxbHLH01和PxbHLH02基因上游启动子区,按照扩增片段为800~2000 bp大小,分别设计特异引物(见表2),以小黑杨叶片的DNA为模板,进行PCR扩增。PCR扩增程序:94 ℃预变性3 min;94 ℃变性 45 s;55 ℃退火 45 s;72 ℃延伸 150 s;共30个循环;72 ℃延伸10 min。PCR产物直接送生工生物工程(上海)股份有限公司测序。

表2 克隆PxbHLH01和PxbHLH02基因启动子对应引物Table 2 The primer used for cloning the promoters in PxbHLH01 and PxbHLH02 genes
1.2.3 PxbHLH01和PxbHLH02基因编码区和启动子序列的生物信息学分析
利用在线分析工具GENSCAN[16]对小黑杨PxbHLH01和PxbHLH02基因的核苷酸序列进行分析。利用在线分析软件Protparam[17]对PxbHLH01和PxbHLH02蛋白的理化性质进行分析。利用在线分析软件ProtScale[17]的Kyte and Doolittle算法对PxbHLH01和PxbHLH02蛋白进行亲水/疏水性分析。利用在线分析工具SignalP[18]的神经网络算法对PxbHLH01和PxbHLH02蛋白进行信号肽的预测和分析。利用在线工具TMPred[19]进行跨膜结构预测和分析。利用在线分析工具COILS[20]进行卷曲螺旋预测和分析。利用在线工具wolf psort[21]进行亚细胞定位分析。采用GOR4[22-23]分析蛋白的二级结构。利用NCBI数据库Blast对PxbHLH01和PxbHLH02基因序列以及推测的编码蛋白进行同源序列比对,搜索不同物种中的同源基因及蛋白。根据李军等[24]推荐的软件组合,先采用ClustalX将氨基酸序列进行多序列比对的分析,然后利用MEGA 5.0软件,算法为Neighbor-Joining,自检举1 000次,构建进化树[25-26]。
利用PLACE(http://www.dna.affrc.go.jp/PLACE/signalscan.html)分析PxbHLH01和PxbHLH02基因启动子序列[27]。
2 结果与分析
2.1 PxbHLH01和PxbHLH02基因编码区全长和启动子序列的鉴定
以小黑杨叶片cDNA为模板进行PCR扩增,扩增结果如图1所示。连接pMD18-T载体,转化克隆进入JM109感受态细胞后,通过与NCBI比对,确定获得了PxbHLH01和PxbHLH02基因序列,分别为411 bp和417 bp。经过比对,与拟南芥UPBEAT1、bHLH151基因核苷酸序列(AT2G47270.1)的一致性分别为38.85%和40.71%;与毛果杨未知功能基因核苷酸序列(XM_002320932.1)的一致性分别为97.57%和82.73%;与毛果杨未知功能基因核苷酸序列(XM_002301486.1)的一致性分别为82.38%和98.32%。
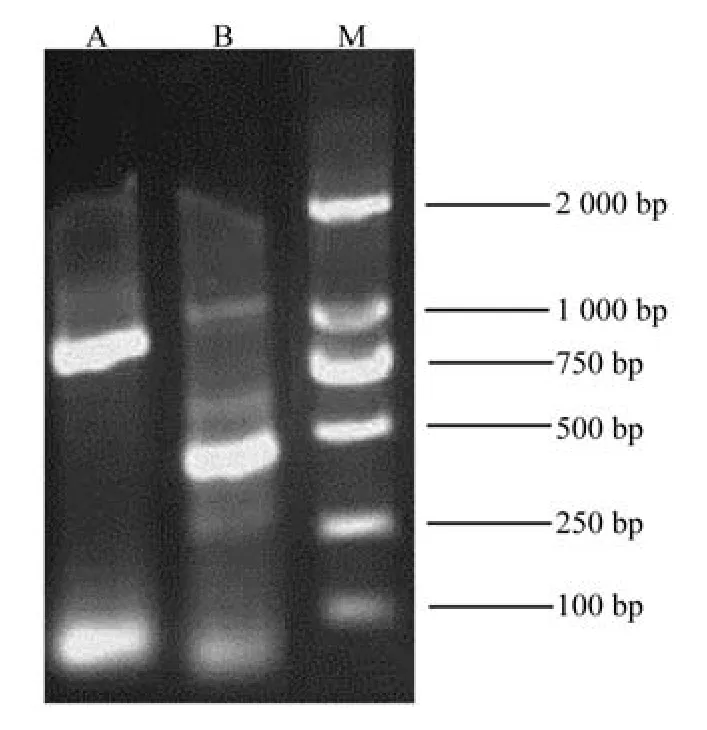
图1 PxbHLH01(A)和PxbHLH02(B)基因PCR扩增片段Fig.1 PCR ampli fi ed fragments of PxbHLH01 (A) and PxbHLH02 (B)
以小黑杨叶片DNA为模板进行PCR扩增,扩增结果如图2所示。对PCR扩增产物直接测序,通过与NCBI比对,确定获得了PxbHLH01和PxbHLH02基因启动子序列,长度分别为1 360 bp和1 913 bp。经过比对,与毛果杨未知功能基因(XM_002320932.1)启动子的一致性分别为95.17%和68.91%;与毛果杨未知功能基因(XM_002301486.1)启动子的一致性分别为69.46%和96.25%。

图2 PxbHLH01(A)和PxbHLH02(B)基因启动子PCR扩增片段Fig.2 PCR ampli fi ed fragments of PxbHLH01 (A) and PxbHLH02 (B) promoters
2.2 PxbHLH01和PxbHLH02蛋白质一级结构的分析
2.2.1 氨基酸序列理化性质分析
利用在线分析软件Protparam对PxbHLH01和PxbHLH02基因的氨基酸序列的理化性质进行分析。PxbHLH01蛋白等电点(pI)为10.39;不稳定系数为38.22(不稳定系数小于40时,预测蛋白质稳定,反之则不稳定),为稳定类蛋白;总平均疏水性为-0.767,说明该蛋白为亲水性蛋白。PxbHLH02蛋白等电点为10.59;不稳定系数为53.02,为不稳定类蛋白;总平均疏水性为-0.666,为亲水性蛋白。
2.2.2 疏水区域/亲水区域预测
蛋白质亲疏水性氨基酸的组成是蛋白质折叠的主要驱动力,通过亲水性预测可以反映蛋白质的折叠情况。利用在线分析软件ProtScale的Kyte and Doolittle算法对PxbHLH01和PxbHLH02蛋白进行亲水/疏水性分析(>0.5区域为疏水区,<-0.5区域为亲水区,介于+0.5~-0.5 之间主要为两性区域)。结果表明,PxbHLH01蛋白共有8个疏水区和10个亲水区(见图3A);PxbHLH02蛋白共有4个疏水区和9个亲水区(见图3B)。
2.2.3 信号肽的预测和分析
信号肽是N端的一段氨基酸序列,指导分泌性蛋白到内质网膜上合成,在蛋白质合成结束之前被切除,信号肽位于蛋白质的N端,一般由16~26个氨基酸残基组成,其中包括疏水核心区、信号肽的C端和N端[28]。利用在线分析工具SignalP的神经网络算法对PxbHLH01和PxbHLH02蛋白进行预测。结果表明,PxbHLH01(见图4A)和PxbHLH02(见图4B)蛋白可能均不存在信号肽。

图3 PxbHLH01(A)和PxbHLH02(B)蛋白亲水/疏水性分析Fig.3 Hydrophobicity / phydrophilicity prediction of PxbHLH01 (A) and PxbHLH02 (B) proteins
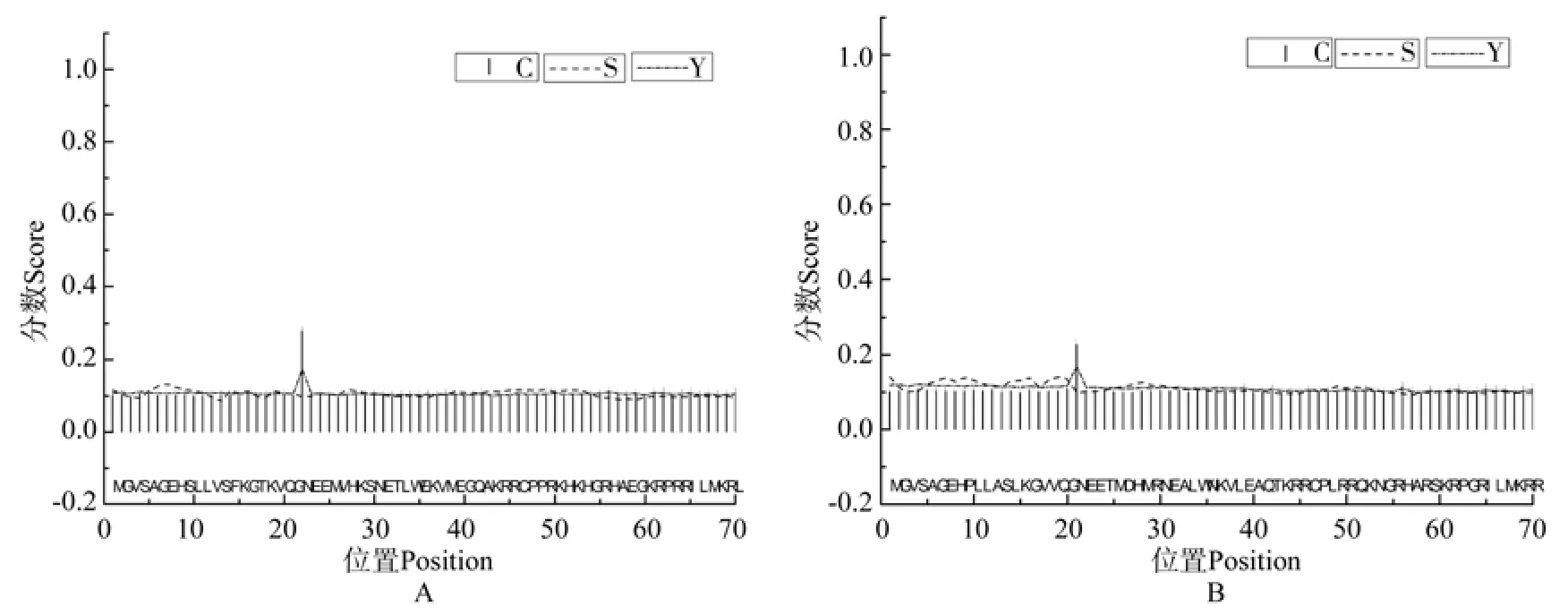
图4 PxbHLH01(A)和PxbHLH02(B)蛋白信号肽的预测和分析(神经网络算法)Fig.4 Prediction and analysis of signal peptides in PxbHLH01 (A) and PxbHLH02 (B) proteins using the neural network algorithm
2.2.4 跨膜结构的预测和分析
跨膜结构是蛋白质与膜内在蛋白的静电相互作用和氢键键合相互作用与膜结合的一段氨基酸片段,一般由20个左右的疏水性氨基酸残基组成,主要形成α-螺旋[29]。利用在线工具TMPred对PxbHLH01和PxbHLH02蛋白进行跨膜结构分析。结果表明,PxbHLH01(见图5A)和PxbHLH02(见图5B)蛋白可能不存在跨膜结构。

图5 PxbHLH01(A)和PxbHLH02(B)蛋白跨膜结构域预测和分析Fig.5 Prediction and analysis of transmembrane domain prediction in PxbHLH01 (A) and PxbHLH02 (B) proteins
2.2.5 亚细胞定位预测
利用在线工具WoLF PSORT分别对PxbHLH01和PxbHLH02蛋白进行亚细胞定位分析。PxbHLH01蛋白在细胞质基质得分为8.0;PxbHLH02蛋白在细胞质基质中得分为7.0。
2.3 PxbHLH01和PxbHLH02蛋白质二级结构的预测和分析
应用GOR4对PxbHLH01和PxbHLH02蛋白的二级结构进行分析。PxbHLH01和PxbHLH02蛋白均是由α-螺旋(Alpha helix)、延伸链(Extended strand)、无规则卷曲(Random coil)所组成的,并且分布于整个蛋白。PxbHLH01蛋白由39.71%的α-螺旋、57.47%的无规则卷曲和8.82%延伸链组成(见图6A);PxbHLH02蛋白由54.35%的α-螺旋、42.03%的无规则卷曲和3.62%延伸链组成(见图6B)。
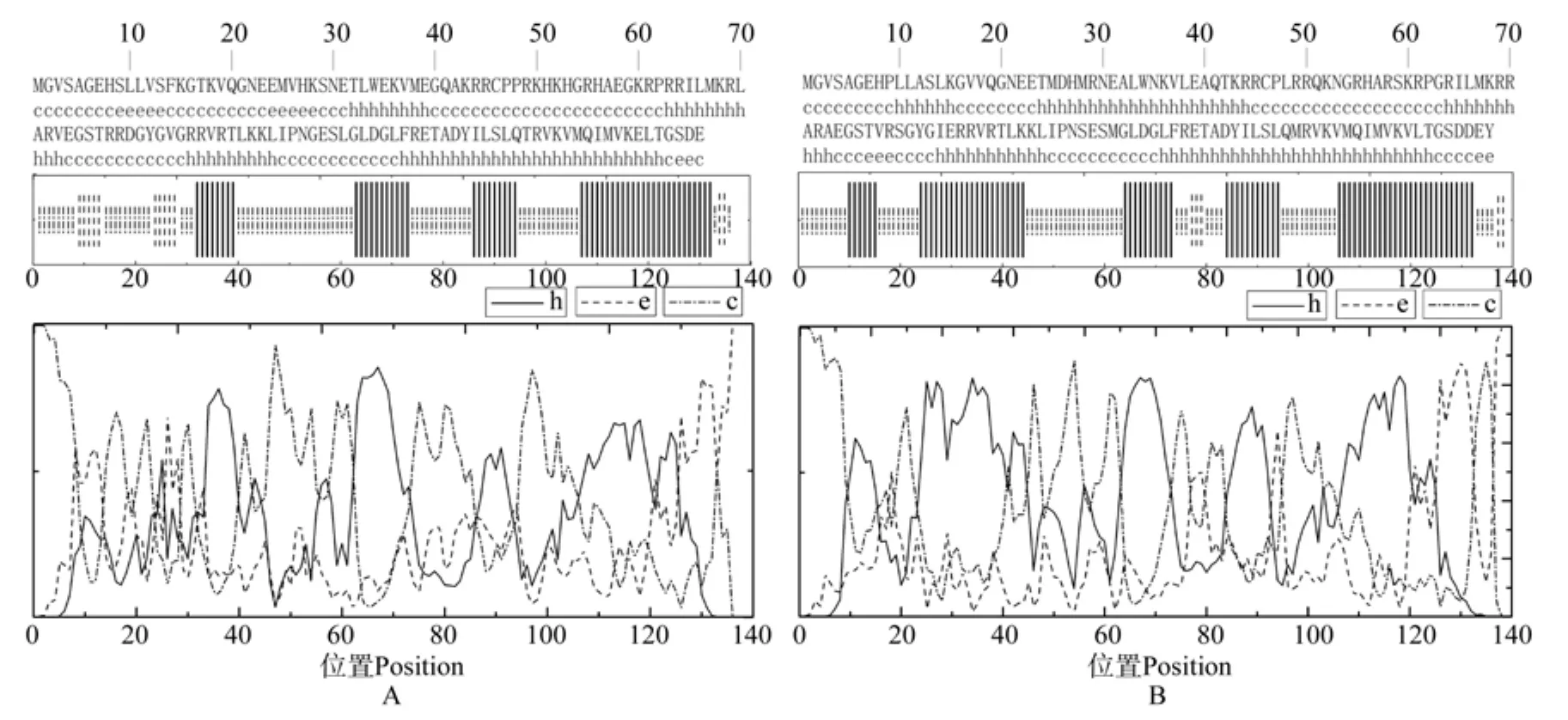
图6 PxbHLH01(A)和PxbHLH02(B)蛋白二级结构预测Fig.6 Secondary structure prediction of PxbHLH01 (A) and PxbHLH02 (B) proteins
2.4 PxbHLH01和PxbHLH02蛋白质三维结构的预测
利用在线分析工具Swiss-Model,对PxbHLH01和PxbHLH02蛋白进行三级结构同源建模,用Swiss-PdbViewer软件对建模结果进行处理[30-32]。PxbHLH01蛋白同源建模结果如图7 A所示,PxbHLH02蛋白同源建模结果如图7 B所示。同时,利用软件的α-碳与酰胺平面交角图功能,可以找到1个残基与1个特定平面的构象角(二面角),通过分析二面角,可以判断一个模型的质量。对PxbHLH01(见图8 A)和PxbHLH02(见图8 B)蛋白的二面角分析结果表明,蛋白质残基位于核心区域,说明空间结构稳定,所以同源建模的方法对PxbHLH01和PxbHLH02蛋白的氨基酸序列的建模结果可靠。
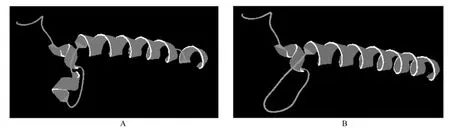
图7 PxbHLH01(A)和PxbHLH02(B)蛋白三级结构预测Fig.7 Three-dimensional structure prediction of PxbHLH01 (A) and PxbHLH02 (B) proteins
2.5 PxbHLH01和PxbHLH02蛋白氨基酸序列比对及系统进化树构建
利用NCBI数据库Blast对PxbHLH01和PxbHLH02基因序列以及推测的编码蛋白进行同源序列比对,Blastp比对结果表明,PxbHLH01和PxbHLH02蛋白与毛果杨Populus trichocarpa(XP_002320968.1和 XP_002301522.1)、 拟南 芥Arabidopsis thaliana(NP_566098.1)、大 豆Glycine max(XP_003556194.1)、 玉 米Zea mays(DAA58466.1)、 水 稻Oryza sativaIndica Group(EAY87819.1)、 葡 萄Vitis vinifera(XP_003632412.1)、 甘 蓝Brassica oleraceavar.capitata(ADK56282.1)、 苜 蓿Medicago truncatula(XP_003617449.1)、 云 杉Picea sitchensis(ADE76325.1)、高粱Sorghum bicolor(XP_002462412.1) 和 蓖 麻Ricinus communis(XP_002515253.1)同源性较高,一致性分别为97%和76%、77%和97%、50%和45%、53%和54%、41%和41%、42%和35%、50%和57%、50%和24%、49%和48%、42%和44%、43%和43%、50%和57%。
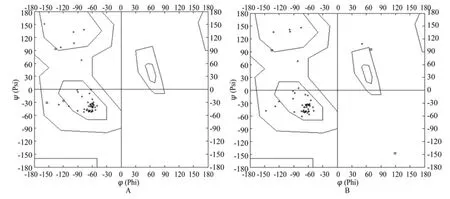
图8 PxbHLH01(A)和PxbHLH02(B)蛋白质残基二面角预测Fig.8 Prediction of residue dihedral angles in PxbHLH01 (A) and PxbHLH02 (B) proteins
系统进化树是物种的进化史,通过构建系统进化树可以根据这些物种的祖先描述它们的进化关系[33]。先采用ClustalX对NCBI比对的氨基酸序列进行多序列比对的分析,然后利用MEGA 5.0软件,应用Neighbor-Joining算法构建系统进化树(见图9)。结果表明,12个物种的14条蛋白序列大致分为4个大类:其中小黑杨、毛果杨、蓖麻、葡萄、大豆、拟南芥和甘蓝聚成一类,云杉、苜蓿和玉米聚成一类,水稻和高粱分别单独聚成一类。PxbHLH01和PxbHLH02蛋白与毛果杨P.trichocarpa(XP_002301522.1和XP_002320968.1)有着较近的亲缘关系。小黑杨、毛果杨等与拟南芥A.thaliana(NP_566098.1)的遗传距离也较近,说明它们在进化上亲缘关系较近;与水稻和高粱遗传距离较远,说明其亲缘关系较远。
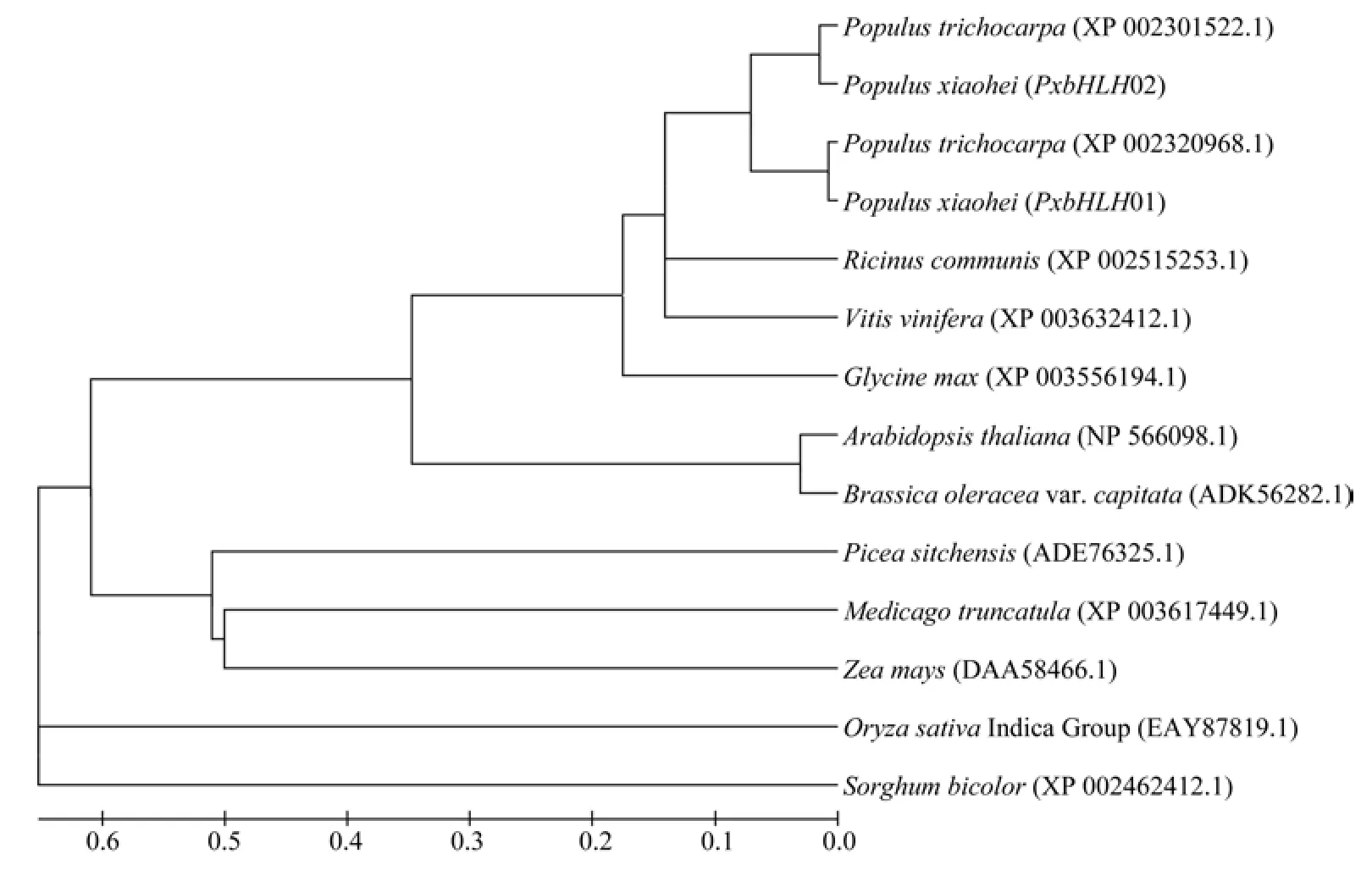
图9 根据PxbHLH01和PxbHLH02蛋白NCBI Blastp比对结果构建的系统进化树Fig.9 Phylogenetic tree of PxbHLH01 and PxbHLH02 proteins based on the Blastp results from NCBI
2.6 PxbHLH01和PxbHLH02基因启动子中含有的顺式作用元件的鉴定及分析
利 用 PLACE(http://www.dna.affrc.go.jp/PLACE/)分析PxbHLH01和PxbHLH02基因启动子序列。PxbHLH01和PxbHLH02基因可能含有的重要转录调控元件如表3所示。结果表明在PxbHLH01和PxbHLH02基因启动子中有转录必备的RNA聚合酶结合TATA box、CAAT box以及一些压力响应元件(如脱水响应元件)和激素响应元件(如生长素、脱落酸、赤霉素、水杨酸等的调控元件),此外,还有一些光调控元件等。
3 结论与讨论
在拟南芥中,bHLH转录因子UPBEAT1(UPB1)通过直接调控一组氧化物酶来调控根部细胞增殖和分化之间的平衡。根据毛果杨序列设计引物克隆所获得的PxbHLH01和PxbHLH02基因,经多序列比对,结果表明与拟南芥编码bHLH结构域的功能蛋白UPBEAT1(UPB1)有较高同源性。
为了鉴定PxbHLH01和PxbHLH02蛋白的功能,通过在线工具对PxbHLH01和PxbHLH02蛋白进行了预测和生物信息学分析。经预测分析,结果表明PxbHLH01和PxbHLH02蛋白均为亲水性蛋白质。这2个蛋白可能不存在信号肽和跨膜结构。这正与亚细胞定位预测结果相互印证,2个蛋白亚细胞定位预测是在细胞质。这说明,PxbHLH01和PxbHLH02蛋白可能在细胞质中起作用,而非分泌蛋白。对PxbHLH01和PxbHLH02蛋白的二级结构的分析预测结果表明,2个蛋白都是由α-螺旋、延伸链、无规则卷曲所组成的。通过对蛋白进行三级结构的同源建模,绘制出2个蛋白可能的结构模型(见图7、8),表明了这2个基因编码蛋白的结构为bHLH(basic helix-1oop-helix)结构。将PxbHLH01和PxbHLH02的氨基酸序列应用NCBI网站的Blastp进行比对,比对结果表明,PxbHLH01和PxbHLH02蛋白与毛果杨、拟南芥、大豆、玉米、水稻、葡萄、甘蓝、苜蓿、云杉、高粱和蓖麻等物种的对应蛋白的同源性较高。同时应用比对结果构建了系统进化树(见图9)。进化树表明,小黑杨PxbHLH01和PxbHLH02蛋白与毛果杨、蓖麻、葡萄、大豆、拟南芥和甘蓝的bHLH转录因子UPBEAT1聚成一类,说明它们在进化上亲缘关系较近。
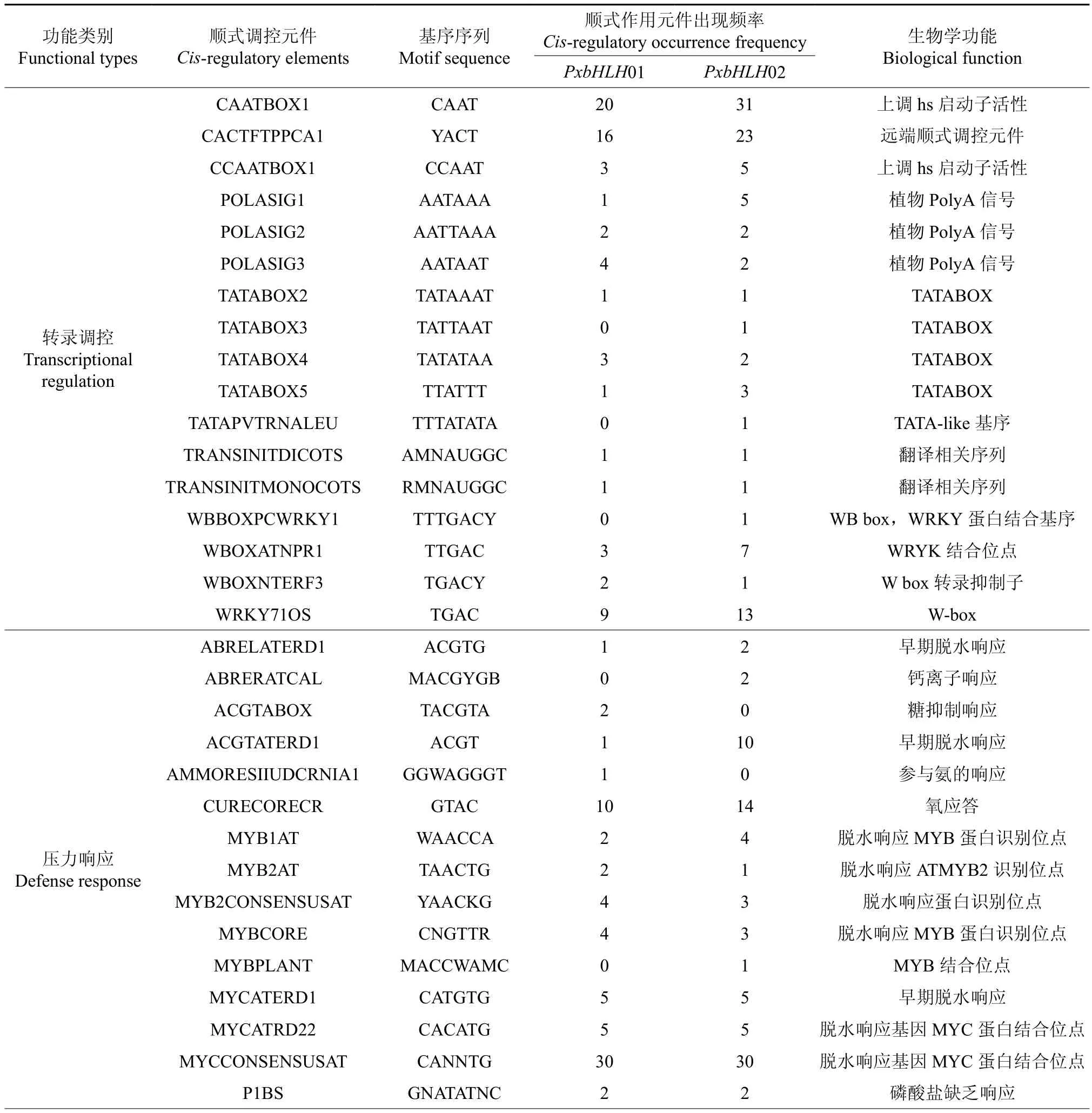
表3 PxbHLH01和PxbHLH02基因启动子顺式调控元件分析Table 3 Analysis of promoter cis-regulatory elements in PxbHLH01 and PxbHLH02
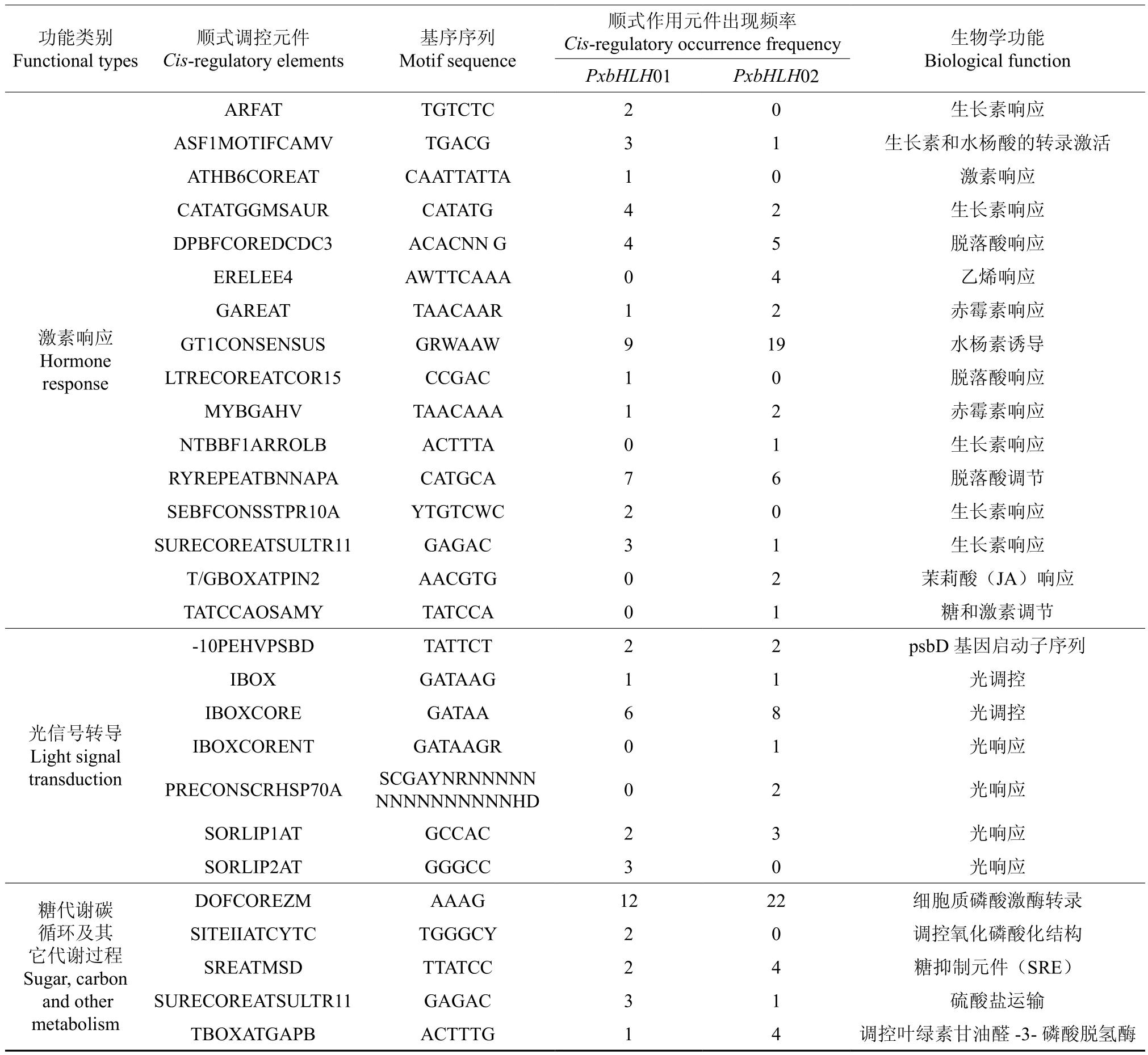
续表3Table 3 Continuation
本研究中同时对PxbHLH01和PxbHLH02基因上游启动子进行了克隆与分析,通过顺式作用元件分析表明,所获得的启动子序列具有启动转录的必要元件。同时,存在一些逆境调控、激素调控、光调控和代谢相关的一些元件。启动子中有大量的抗胁迫功能元件,而这些逆境调控元件中,脱水响应基因占有较大比例,表明PxbHLH01和PxbHLH02基因有可能参与了植物对胁迫的响应过程,并且,在植物的抗旱过程中起到重要作用,参与植物的脱水响应调控。大量的激素调控元件表明,PxbHLH01和PxbHLH02基因可能与植物激素调控有关。值得注意的是,在分析启动子元件时发现了多个氧应答元件(CURECORECR),表明PxbHLH01和PxbHLH02基因的功能可能与之前的研究相一致,通过调控植物体内的氧化物酶来调控植物生长。
参考文献:
[1] 王勇江,陈克平,姚 勤.bHLH转录因子家族研究进展[J].遗传, 2008, 30(7): 821-830.
[2] 张全琪,朱家红,倪燕妹,等.植物bHLH转录因子的结构特点及其生物学功能[J].热带亚热带植物学报,2011,19(1):84-90.
[3] 何开平,吴 楚.bHLH转录因子对植物形态发生的影响[J].安徽农业科学, 2010, 38(35): 19957-19959.
[4] 刘晓月,王文生,傅彬英.植物bHLH转录因子家族的功能研究进展[J].生物技术进展, 2011, 1(6): 391-397.
[5] 张 莉,苏曼琳.植物抗旱基因HDCS1的克隆和表达载体的构建[J].中南林业科技大学学报, 2012, 32(6): 115-117.
[6] KHANNA R, SHEN Y, MARION C M.The basic helix-loophelix transcription factor PIF5 acts on ethylene biosynthesis and phytochrome signaling by distinct mechanisms[J].Plant Cell,2007, 19(12): 3915-3929.
[7] Krizek B A, Fletcher J C.Molecular mechanisms of flower development: an armchair guide[J].Nature Reviews Genetics,2005, 6: 688-698.
[8] Zhao M, Morohashi K, Hatlestad G.The TTG1-bHLH-MYB complex controls trichome cell fate and patterning through direct targeting of regulatory loci[J].Development, 2008, 135:1991-1999.
[9] 普 莉, 索金凤, 薛勇彪.植物表皮毛发育的分子遗传控制[J].遗传学报, 2003, 30(11): 1078-1084.
[10] Kiribuchi K, Jikumaru Y, Kaku H.Involvement of the basic helix-loop-helix transcription factor RERJ1 in wounding and drought stress responses in rice plants[J].Biosci Biotechn Biochem, 2005, 69(5): 1042-1044.
[11] Albert N W, Lewis D H, Zhang H B.Light-induced vegetative anthocyanin pigmentation inPetunia[J].Journal of Experimental Botany, 2009, 60(7): 2191-2202.
[12] 王 战,方振富.中国植物志:第20卷[M].北京: 科学出版社,1984: 66-67.
[13] 黄 婷,徐刚标.毛果杨WND1B基因启动子的克隆与缺失初步分析[J].中南林业科技大学学报, 2012, 32(4): 164-169.
[14] Tsukagoshi1 H, Busch1 W, Benfey P N.Transcriptional Regulation of ROS Controls Transition from Proliferation to Differentiation in the Root[J].CELL, 2010, 143(4): 606-616.
[15] Tuskan GA, Difazio S, Jansson S,et al.The genome of black cottonwood,Populus trichocarpa(Torr & Gray)[J].Science,2006,313 (5793): 1596-1604.
[16] Burge C B, Karlin S.Finding the genes in genomic DNA [J].Curr Opin Struct Biol,1998, 8: 346-354.
[17] John M W.The Proteomics Protocols Handbook[M].Totowa:Humana Press, 2005: 571-607.
[18] Thomas N P, Soren B, Gunnar H.SignalP 4.0: discriminating signal peptides from transmembrane regions[J].Nature Methods,2011,(8):785-786.
[19] Hofmann K, Stoffel W.TMbase-A database of membrane spanning proteins segments[M].Biol Chem Hoppe-Seyler, 1993,(374): 166.
[20] Lupas A, Van D M, Stock J.Predicting Coled Coils from Protein Sequences[J].Science 1991, (252): 1162-1164.
[21] Horton P, Park KJ, Obayashi O.WoLF PSORT: Protein Localization Predictor[J].Nucleic Acids Research, 2007: 1-3.
[22] Sen TZ, Jernigan RL, Garnier J,et al.GOR V server for protein secondary structure prediction[J].Bioinformatics, 2005,(21):2787-2788.
[23] 谢禄山,谭晓风, 张 琳,等.油桐种子FAD2基因全长cDNA序列分析[J].经济林研究, 2012, 30(2): 1-9.
[24] 李 军,张莉娜, 温珍昌.生物软件选择与使用指南[M].北京:化学工业出版社, 2008: 80.
[25] Tamura K, Dudley J, Nei M.MEGA4: Molecular evolutionary genetics analysis (MEGA) software version 4.0[M].Molecular Biology and Evolution, 2007: 1596-1599.
[26] 罗 静,刘正富,武 峥,等.桃水孔蛋白基因克隆及序列分析[J].经济林研究, 2012, 30(1): 77-80.
[27] Higo K, Ugawa U, Iwamoto M.Plantcis-acting regulatory DNA elements (PLACE) database[J].Nucleic Acids Research, 1999,(27): 297-300.
[28] 翟中和,王喜忠,丁明孝.细胞生物学[M].北京: 高等教育出版社,2000: 191.
[29] 王镜岩,朱圣庚,徐长法.生物化学[M].北京: 高等教育出版社, 2002: 229.
[30] Arnold K, Bordoli L, Kopp J,et al.The SWISS-MODEL Workspace: A web-based environment for protein structure homology modeling[J].Bioinformatics, 2006, 22:195-201.
[31] Schwede T, Kopp J, Guex N,et al.SWISS-MODEL: an automated protein homology-modeling server[J].Nucleic Acids Research, 2003, 31: 3381-3385.
[32] Guex, N, Peitsch M C.SWISS-MODEL and the Swiss-PdbViewer: An environment for comparative protein modeling[J].Electrophoresis, 1997, 18: 2714-2723.
[33] Harrison C J, Langdale J A.A step by step guide to phylogeny reconstruction[J].The Plant Journal, 2006,(45): 561-572.
