油桐β-酮脂酰-CoA合成酶(KCS)基因的克隆与序列分析
2013-04-03周俊琴谭晓风刘美兰龙洪旭
周俊琴,谭晓风,刘美兰,龙洪旭
(1.经济林培育与保护教育部重点实验室,湖南 长沙 410004;2.经济林育种与栽培国家林业局重点实验室,湖南 长沙 410004)
油桐是大戟科Euphorbiaceae油桐属Vernicia植物,是我国特有而重要的经济林木,它与油茶、乌桕和核桃并称为我国四大木本油料植物[1]。油桐种子的含油率高达50%~70%,它具有优异的防水、防腐和绝缘等性能,是重要的工业油料[2-3]。
β-酮脂酯-CoA合酶(KCS)是植物体内超长链脂肪酸及其衍生物第2步合成中最重要的脂肪酰-CoA延长酶,它以油酯-CoA(或C20∶1脂酸-CoA)和丙二酰-CoA为原料,合成C20∶1或C22∶1的β-酮脂酰-CoA[4]。超长链脂肪酸是生物体中碳原子数超过18碳的脂肪酸,这一类脂肪酸在生物体中具有广泛而重要的生理功能,它们参与种子中甘油酯、生物膜膜脂和鞘脂的合成,同时为角质层蜡质的生物合成提供前体物质。角质层是植物自我防护的最后一道屏障,对植物生长发育和适应外界环境方面有着重要的作用[5]。目前,人们对脂肪酸延长酶基因的研究主要集中在β-酮脂酯-CoA合酶(KCS)基因的研究上。KCS基因家族主要存在于植物界中,且不同物种所具有的KCS基因家族成员的种类和数量都不完全相同。在拟南芥中,共有21个成员(KCS1~KCS21)[6],其中,KCS1[7]、CER6(KCS6)[8-10]、FDH(KCS10)[11-12]和 FAE1[13-14]的功能已有报道。之后陆续分离得到西蒙德木KCS[15]、雷斯克勒KCS3[16]和苔藓类植物地衣FAE2[7]。在油菜中,主要对种子油脂中参与VLCFAs合成相关的FAE1(KCS18)基因做了大量研究[17]。β-酮脂酯-CoA合酶是超长链脂肪酸合成第1步缩合反应的限速酶,是在内质网中催化合成的。为给使用生物技术定向调控油料作物油脂组成奠定基础,对β-酮脂酯-CoA合酶基因进行了克隆与序列分析研究。
1 材料与方法
1.1 试验材料
1.1.1 植物材料
2012年8月底在湖南省永顺县青坪镇中南林业科技大学油桐试验基地(国家油桐种质资源保存库)采集油桐种子,材料贮存于-80 ℃冰箱中。
1.1.2 试验试剂
试验所用试剂有:购自Invitrogen公司的PureLinkTM RNA Mini Kit;购自全式金公司的2×EasyTaq PCR SuperMix、pEASY-Blunt Simple Cloning Kit、大肠杆菌Trans-T1感受态细胞;购自Takara的PrimeSTAR HS DNA Polymerase高保真DNA聚合酶;购自天根公司的引物合成及测序;引物合成及测序结果由华大基因完成,其它试剂均为分析纯。
1.2 试验方法
1.2.1 总RNA的提取和cDNA的合成
采用试剂盒加CTAB裂解和氯仿/异戊醇抽提的综合方法提取油桐种子内的总RNA,之后用1.5%的琼脂糖凝胶电泳检测RNA提取效果。反转录反应按照TaKaRa 公司cDNA 合成试剂盒说明书完成得到1条链cDNA。
1.2.2 油桐KCS基因的克隆
从油桐种子转录组测序结果中获得KCS的全长序列,利用Primer Premier 5 软件设计的上游引物为:Vf_KCS-WF “GCAGAGCGTGAATCTCAA TAACT”;下游引物为:Vf_KCS-WR“CCAACT GATCTATCAAAGTACACTCCTC”。然后以油桐种子反转录的单链cDNA 为模板,进行 PCR 扩增。反应体系及程序见Takara的PrimeSTAR HS DNA Polymerase高保真DNA聚合酶说明书。回收PCR产物并连接到pEASY-Blunt Simple平末端载体中,转化Trans-T1大肠杆菌感受态细胞,在含有氨苄青霉素的LB培养基上筛选阳性,筛选的阳性克隆送华大基因公司测序。
1.2.3 生物信息学分析
通过生物信息学软件Vector NTI 10.3.0、GENDOC、MEGA5.10、UCSF Chimera对 油桐β-酮脂酯-CoA合酶基因序列和测序结果进行分析和预测,并用在线软件Clustalw2、ProtParam、SignalP4.1、TargetP 1.1 Server、ProtScale、TMpred Server、Mobyle portal、SOPMA 和EsyPred 3 Dwebserver 1.0全面剖析KCS基因的结构与功能。
2 结果与分析
2.1 油桐种仁RNA的提取和KCS基因的克隆
对提取的油桐种仁RNA并电泳检测其完整性,结果如图1所示。由图1可知,RNA没有降解,且28S条带较亮。随后根据 TaKaRa 公司cDNA 合成试剂盒说明书完成反转录反应,从而得到第1条链cDNA,以此为模板进行PCR 扩增。
根据测序结果比对分析,Vf_KCS全长为 1 440 bp,见图2。其中 5′UTR 长为 41 bp,3′UTR 长为 61 bp,CDS长为 1 338 bp,推测其编码446个氨基酸。将Vf_KCS的CDS序列进行BLASTX 分析后共搜索到100多条相似度很高的KCS同源序列,其中与葡萄、草莓、陆地棉的相似度达81%,与油茶、浙江红山茶、黄瓜、鹰嘴豆的相似度为80%,与大豆、苜蓿、番茄的相似度为76%。因此可以判断Vf_KCS的CDS为β-酮脂酯-CoA合酶基因。

图1 油桐种仁RNA检测Fig.1 RNA detection of Vernicia fordii seed
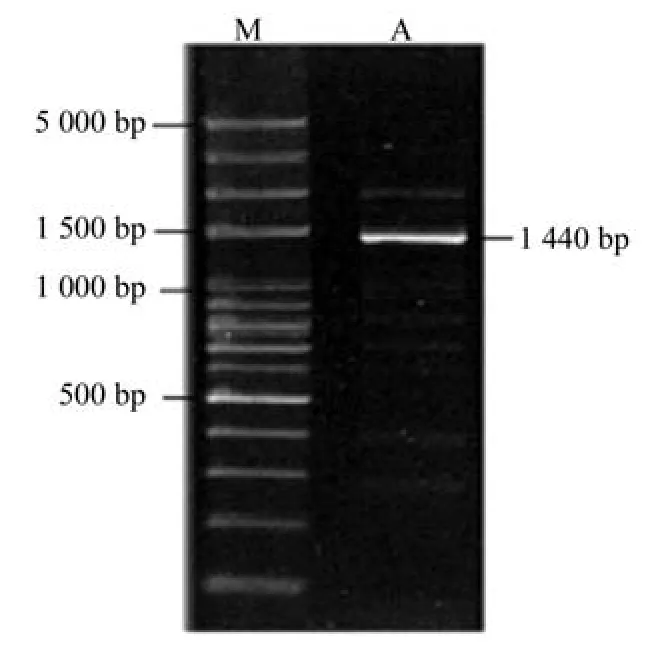
图2 Vf_KCS的PCR扩增检测Fig.2 PCR ampli fi cation of Vf_KCS
2.2 油桐KCS蛋白结构分析
2.2.1 油桐KCS基因编码蛋白的氨基酸序列分析
油桐KCS基因全长1 338 bp,编码1个完整的开放阅读框(ORF)。该基因编码1个含446个氨基酸的核苷酸序列,是缩合酶超家族中的一员,由图3可以看出该基因编码的蛋白质具有3个酶活性位点、6个产物结合位点、5个丙二酰-CoA结合位点和多个二聚体形成位点,以及类查尔酮合酶(CHS-like)高度同源区域。通过得到的油桐β-酮脂酯-CoA合酶基因的氨基酸序列信息以及其它物种的同种基因的序列信息,可知Vf_KCS的氨基酸序列有3个高度保守区,序列分别为“PTPSLSAM”、“FGNTSSSS”、“GSGFKCNSAVW”;1个β-酮脂酯-CoA合酶基因的催化区域,其中半胱氨酸cys为其催化位点,序列为“GMGCSA”,具体位置见图7。分析结果证实本研究克隆到了1条完整的油桐KCS基因序列。
2.2.2 油桐KCS基因编码蛋白的理化性质和疏水性/亲水性的分析
利用在线ProtParam对KCS基因编码的蛋白质进行了理化性质分析,结果表明:Vf_KCS所形成的蛋白相对分子质量为50.15 kDa;等电点为9.21;理论推导半衰期为30 h;编码蛋白的原子组成分别为C2264H3552N622O627S20;不稳定系数是33.88,由此说明这2个蛋白是稳定性蛋白。

图3 Vf_KCS蛋白保守结构域分析结果Fig.3 Analysis result of conserved domain of Vf_KCS protein
采用在线ProtScale分析预测Vf_KCS编码蛋白的疏水性,参照其数字文本格式分析发现Vf_KCS有265个亲水性氨基酸残基和173疏水性氨基酸残基,分别占总残基数的60.5 %和39.5 %,编码的氨基酸序列中亲水性氨基酸、疏水性都均匀分布在整条肽链中,脂肪系数为94.44,总平均亲水性为-0.035,综合分析可推测Vf_KCS基因编码的蛋白是亲水性蛋白。
2.2.3 油桐KCS基因编码蛋白的跨膜拓扑结构预测
根据在线Mobyle portal对Vf_KCS进行跨膜区拓扑结构预测结果如图4显示:Vf_KCS有2个跨膜域和3个疑似跨膜域,2个跨膜域的位置分别在整个序列的第17~37,300~320的氨基酸残基,3个疑似跨膜域位置分别在整个序列的第138~158,168~188,220~240的氨基酸残基。每个跨膜结构域由20个氨基酸残基组成的螺旋,同时该序列的跨膜蛋白N端位于细胞质中,C端位于细胞膜外。

图4 Vf_KCS 的跨膜拓扑结构预测Fig.4 Prediction of transmembrane topology in Vf_KCS
2.2.4 油桐KCS基因编码蛋白二级结构及三级结构的预测
在线工具SOPMA 软件预测分析Vf_KCS氨基酸序列的二级结构,结果显示Vf_KCS氨基酸残基组成为:α-螺旋(Hh)为 45.07%、β-折叠(Ee)为 15.25%、β-转 角(Tt)为5.61%、随机卷曲(Cc)为34.08%。并且可以推测α-螺旋最有可能的位置是3~35,60~ 70,106~ 120,185~ 195,245~255,283~ 300,307~ 322,349~ 359,364~372,383~393,415~423;β-折叠最有可能的位置是42~47,138~144,168~173,198~ 204,278~ 282,403~ 408,且 Vf_KCS的二级结构以α-螺旋和β-折叠为主。
根据EsyPred3Dwebserver1.0在线软件对Vf_KCS进行三维结构预测结果如图5,并利用本地软件UCSF Chimera 进行结构如图6分析显示:Vf_KCS编码蛋白由7个α螺旋和4个β折叠组成。从疏水表面立体图可以看到酶的整个轮廓,其中橘黄色为α螺旋,占主要部分。

图5 Vf_KCS带状立体模型Fig.5 Ribbons spatial model of Vf_KCS

图6 Vf_KCS疏水表面立体模型Fig.6 Hydrophobic surface graphic model of Vf_KCS
2.3 油桐KCS基因氨基酸序列同源性和系统进化树分析
将克隆得到的Vf_KCS的cDNA序列推导出的氨基酸序列与其它物种的KCS基因所编码的氨基酸通过在线软件clustalw2和GENDOC进行同源性分析,结果如图7所示;用软件MEGA4.0构建进化树(见图8)。结果显示此类蛋白质C端及中间区域保守性较高,如239~247,264~271,453~471,485~505等区段高度保守,N端保守性较差,N端物种之间的差异也比较大。同时,Vf_KCS与葡萄的KCS基因相似度最高达89.69 %,与其它植物的KCS基因所编码的氨基酸同源性大多在 80 %以上,但从不同物种KCS种系进化分析图来看,Vf_KCS与拟南芥的亲缘关系是最为接近。
3 结论与讨论
β-酮脂酯-CoA合酶是在内质网中催化超长链脂肪酸合成的第1步缩合反应限速酶,在植物脂肪酸合成途径中起着极其重要的作用,同时也是角质层蜡质生物合成的前体物质。本文从基因辨别、蛋白质的理化特性分析、结构预测、序列比对、分子进化等方面对油桐β-酮脂酯-CoA合酶基因及其编码的蛋白质序列进行了分析,获得了一些该
序列的相关数据和信息,如Vf_KCS的CDS长度为1 338 bp,编码446个氨基酸,所形成的蛋白相对分子质量分别为50.15 kDa,等电点为9.21;它有3个高度保守区为“PTPSLSAM”、“FGNTSSSS”、“GSGFKCNSAVW”和1个β-酮脂酯-CoA合酶基因的催化区域为“GMGCSA”,其中半胱氨酸cys为其催化位点;Vf_KCS存在2个跨膜域和3个疑似跨膜域,二级结构以α-螺旋和β-折叠为主;与其它物种来源的β-酮脂酯-CoA合酶基因及其编码序列具有高度的同源性,并且在结构及特性上非常相符合,从而进一步验证了本试验所得的基因为油桐的β-酮脂酯-CoA合酶基因全长cDNA,为以后的油桐β-酮脂酯-CoA合酶基因的研究工作提供了理论依据。下一步将通过各种试验验证该基因在油桐中的各种功能。

图7 不同物种KCS氨基酸序列比对结果Fig.7 Amino acid sequences alignment in KCS from different species
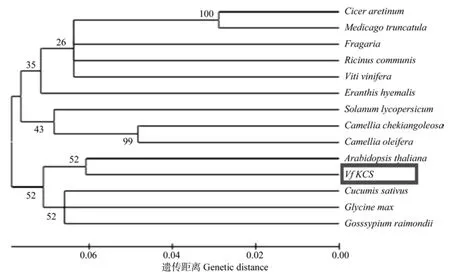
图8 不同物种KCS种系进化分析Fig.8 Phylogenetic analysis of KCS from different species
参考文献:
[1] 谭晓风.油桐的生产现状及其发展建议[J].经济林研究,2006, 24(3): 62-64.
[2] 何 方,何 柏,王承南,等.油桐产品质量等级标准制订说明[J].经济林研究, 2005, 23(4): 118-122.
[3] 孙 颖,卢彰显,李建安.中国油桐栽培利用与应用基础研究进展[J].经济林研究, 2007, 25(2): 84-87.
[4] Lassner MW, Lardizabal K, Metz JG.A jojobabeta-Ketoaeyl-CoA synthase cDNA Complements the canola fatty acid elongation mutation in transgenic plants[J].Plant Cell,1996,8(2):281-292.
[5] 倪 郁,郭彦军.植物超长链脂肪酸及角质层蜡质生物合成相关酶基因研究现状[J].遗传,2008,30(5):561-567.
[6] Toubes J, Raffaele S, Bourdenx B,et al.The VLCFA elongase gene family inArabidopsis thaliana: phylogenetic analysis, 3D modelling and expression profiling[J].Plant Mol Biol, 2008,67(5): 547-266.
[7] Todd J, Post-Beittenmiller D, Jaworski JG.KCS1 encodes a fatty acid elongase β-ketoaeyl-CoA synthase affecting wax biosynihesis inArabidopsis thaliana[J].Plant J,1999,17(2):119-130.
[8] Hooker TS, Millar AA, Kunst L.Signi fi cance of the expression of the CER6 condensing enzyme for cuticular wax production inArabidopsis[J].Plant Physiol, 2002,129: 1568-1580.
[9] Fiebig A.Alterations in CER6, a gene identical to CUT1,differentially affect long-chain lipid content on the surface of pollen and stems[J].Plant Cell, 2000, 12:2001-2008.
[10] Millar AA, Clemens S, Zachgo S,et al.CUT1, anArabidopsis generequired for cuticular wax biosynthesis and pollen fertility,encodes a very-long-chain fatty acid condensing enzyme[J].Plant Cell, 1999, 11(5): 825-838.
[11] Pruitt RE.FIDDLEHEAD, a gene required tosuppress epidermal cell interactions inArabidopsis, encodes a putative lipid biosynthetic enzyme[J].PNAS,2000, 97: 1311-1316.
[12] Yephremov A.Characterization of the FIDDLEHEAD gene ofArabidopsisreveals a link between adhesion response and cell differentiation in the epidermis[J].Plant Cell,1999,11:2187-2201.
[13] Millar AA, Kunst L.Very-long-chain fatty acid biosynthesis is controlled through the expression and specificity of the condensing enzyme[J].Plant J, 1997, 12(1): 121-131.
[14] James DW Jr, Lim E, Keller J,et al.Directed tagging of theArabidopsisfatty acid elongation 1 (FAE1) gene with the maize transposon activator[J].Plant Cell, 1995, 7(3): 309-319.
[15] Das S, Roseoe TJ, Delseny M,et al.Cloning and molecular charaeterization of the fatty acid elongase l (FAE1) gene from high and low erucic acid lines ofBrassica campestrisandBrassica oleraeea[J].Plant Sci, 2002, 162:245-250.
[16] Rossak M, Smith M, Kunst L.Expression of the FAEI gene and FAEI promoter aecivity in developing seeds ofArabidopsis thaliana[J].Plant Mol Biol, 2001, 46(6):717-725.
[17] 郭经宇.甘蓝型油菜脂肪酸延长酶1基因FAE1的克隆、载体构建、原核表达及转基因拟南芥的初步研究[D].成都:四川大学,2006.
