ELPs-PDOR融合基因表达条件的优化
2012-12-29王文研张光亚
王文研,张光亚
(华侨大学 化工学院,福建 厦门 361021)
ELPs-PDOR融合基因表达条件的优化
王文研,张光亚
(华侨大学 化工学院,福建 厦门 361021)
利用均匀设计法和二次多项式逐步回归,对重组大肠杆菌生产类弹性蛋白多肽-1,3-丙二醇氧化还原酶(ELPs-PDOR)重组蛋白的培养条件进行优化 .结果表明:在装液量为30%,诱导剂浓度为6.3mmol·L-1,诱导温度为30℃,诱导时间为2h的优化条件下进行培养,重组菌融合蛋白表达量是原始表达量的3.3倍,酶活力提高到10.84mkat·L-1,提高2.1倍.
类弹性蛋白多肽;1,3-丙二醇氧化还原酶;大肠杆菌;重组蛋白;均匀设计
类弹性蛋白多肽(ELPs)是由五肽重复序列单元构成,其中第4位为除Pro以外的任一氨基酸[1].ELPs具有依赖于自身的序列、盐度、温度敏感的可逆相变过程[2],并利用可逆相变过程纯化重组蛋白的纯化方法是一种新型、发展潜力大、应用前景广泛的非色谱纯化技术,具有操作简单、回收率高、可对目标蛋白进行浓缩富集等优势,可作为新纯化方法代替传统的色谱等传统的蛋白纯化方法[3].1,3-丙二醇氧化还原酶(PDOR,EC 1.1.1.202)是生产1,3-丙二醇(1,3-PD)的关键酶之一 ,具有广阔的应用前景,但目前仍未实现商品化.3-羟基丙醛(3-HPA)是甘油代谢中的一种中间产物 .由甘油经脱水酶作用后形成高浓度的3-羟基丙醛对菌体自身有很大的伤害.快速把产生的3-羟基丙醛转化为1,3-丙二醇是提高1,3-丙二醇产量和避免3-羟基丙醛大量积累的有效手段,与该转化密切相关的就是PDOR,因此PDOR的产出与纯化至关重要.ELPs标签的纯化方法为纯化重组蛋白提供了新方法[4].但由于ELPs自身结构的特点,其具备高度重复的序列,在基因表达过程中频繁使用某些特定的密码子而导致密码子疲劳,从而使得基因表达效率较低 .当连接到待分离的重组蛋白后,其基因序列进一步增长,转化到表达载体中表达往往效率不高,成为生产酶制剂的重要制约因素.本文利用均匀设计方法,对导入到大肠杆菌中ELPs-PDOR的基因表达条件进行优化,找出其最适的培养条件.
1 材料与方法
1.1 菌种
表达宿主菌Escherichia coli BLR (DE3),本实验室保存;质粒pUC19-ELPs-dhaT,pET22b(+)-ELPs-dhaT,由本实验室构建合成[5].
1.2 培养基及培养条件
1)TB培养基.蛋白胨12.0g·L-1,酵母提取物24.0g·L-1,NH4Cl 3g·L-1,甘油4.5mL·L-1,KH2PO41.7mmol·L-1,K2HPO47.2mmol·L-1,pH=7.0.
2)LB培养基.蛋白胨10.0g·L-1,酵母浸膏5.0g·L-1,NaCl 10g·L-1,pH=7.0.
1.3 培养条件优化
将含有pET-22b-ELPs-dhaT重组质粒的BLR(DE3)工程菌按1%的接种量接种到的5mL含100μmol·L-1氨苄青霉素的LB种子培养基中,于37℃,200r·min-1下过夜培养;然后,按1∶100的量将种子菌接种到含100μmol·L-1氨苄青霉素的TB培养基,于35℃,200r·min-1下培养至D(600)为0.8时,加入IPTG诱导剂,低温诱导.将发酵培养液于4℃,4 000r·min-1条件下离心15min,收获菌体;然后用预冷的磷酸盐(PBS)缓冲液(137mmol·L-1NaCl,2.7mmol·L-1KCl,4.2mmol·L-1NaH2PO4,1.4mmol·L-1KH2PO4,pH=7.3)重悬菌体,置冰浴中;最后用300W 超声破碎菌体4 min,其上清为无细胞抽提液 .通过测定可得到酶活力的变化.
1.4 PDOR 酶活力测定[6-7]
在含30mmol·L-1硫酸铵,1μmol·L-1的硫酸亚铁铵,0.1mol·L-1的1,3-PD,2.0mmol·L-1的 NAD+和0.1mol·L-1的碳酸钾缓冲液(pH=9.5)的1.5mL酶反应体系中,在45℃,340nm下连续测定并跟踪酶反应过程中的吸光值变化;然后,通过酶活计算式可得到PDOR的酶活(z).对于PDOR还原3-HPA为1,3-PD的正反应而言,一个酶活单位(kat)定义为1s内催化1mol底物3-HPA所需要的酶量.
2 结果与讨论
2.1 优化条件的选择
重组目的蛋白ELPs-PDOR在大肠杆菌中的表达受众多因素的影响,如接种量、培养温度、溶氧量、诱导剂IPTG浓度、诱导温度、诱导时间等.其中,氧是影响大肠杆菌生长和代谢的重要因素之一,大肠杆菌只能利用溶解在培养基中的氧气生长.当培养基中氧气不足时,大肠杆菌的呼吸链和三羧酸循环(TCA)就会被抑制,转而通过糖酵解途径来获取能量,同时大量积累乙酸;而乙酸的积累会抑制菌体的生长以及目的重组蛋白的合成.温度是影响重组菌生长和产物合成的又一重要因素,重组菌的生长速度和温度密切相关,随着温度的降低,重组菌的代谢速度明显下降,对氧和营养物质的消耗明显下降.
IPTG作为诱导剂,对于大肠杆菌的生长密度及代谢速度有明显的影响[8],加入IPTG后,菌体生长速度降低,降低生长压力,同时增加表达重要蛋白产物的速度.诱导温度对基因的调控机制影响很复杂,涉及DNA的复制、转录、翻译及低分子量调节分子合成等方面,以及菌体生长密度、代谢物的产生是不可或缺的考察因素,ELPs重组蛋白在大肠杆菌中表达已有文献报道,其诱导温度多在25℃.
基于上述原因,选取对重组蛋白蛋白表达产物有重要影响的4个因素:诱导剂浓度(异丙基-β-D-硫代半乳糖苷,IPTG)(X1)、诱导温度(X2)、诱导时间(X3)和装液量(X4)为自变量,以PDOR酶活力为因变量Y,采用U(144)不等水平均匀设计表设计实验[9],对重组菌产生重组蛋白酶的进行优化.
2.2 培养最优条件确定
考察培养基成分对ELPs-PDOR蛋白酶活力的影响,其均匀设计方案及实验结果如表1所示.通过二次多项式逐步回归分析,以调整相关数R最大为原则,对该模型进行显著性检验,建立回归方程为
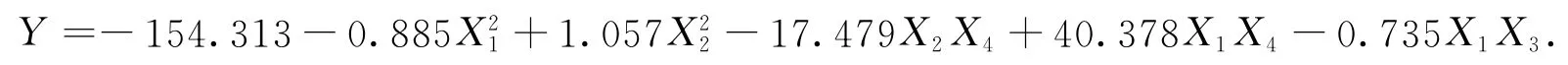
其中:相关系数R为0.996,F值为186.958,显著性水平小于0.001,回归极显著,表明该方程能很好地拟合各考察参数对于目标蛋白酶活力的影响过程.
通过规划求解,目标蛋白酶活力Y 的最大值为11.40mkat·L-1,此时各因素值:X1=6.3,X2=30,X3=2,X4=0.3.表明,当诱导剂量为6.3mmol·L-1,诱导温度为30℃,诱导时间为2h,装液量为30%时,目标蛋白酶活力理论酶活力可达11.40mkat·L-1.

表1 培养条件优化均匀设计表Tab.1 Uniform design table of cultivation parameters optimization

续表Continue table
2.3 培养条件对产酶量的影响
由表1可知:第8组实验组得到酶活力为10.27mkat·L-1,是14组中产酶量最高的一组;第4组实验得到的酶活力为9.16mkat·L-1,仅次于第8组 .这两组培养条件的诱导温度(X2)均为各水平中最大值30℃,在此温度下的培养中,菌体生长,代谢处于活跃状态,对于目标蛋白的产生具有优势.初步得到X2对产酶量有较大影响.另由回归方程得到X22系数的t值为26.21,此因素对Y值影响最大,故诱导温度对于E.coli产生重组蛋白影响显著.第10组实验组得到的酶活力仅为4.69mkat·L-1,由于其培养时诱导温度为18℃,温度较低,菌体生长受抑制,表达蛋白的速度减缓,因此造成酶活力较低.同时,此实验组的培养基装液量为90%,菌体培养时,由于溶氧量缺乏,使得菌体代谢速度缓慢.上述两种因素是造成第10组实验组产生目标蛋白的基因表达效率降低,PDOR酶活力较低的主要原因.
诱导剂量也是影响重组菌产生目标蛋白的关键因素,加入IPTG诱导剂后,随着诱导时间的增加,产生蛋白酶活力有所下降.这主要是由于IPTG对菌体有一定毒性,甚至过量表达的蛋白也会使细胞致死;同时,长时间存在且不能被分解的IPTG会引起酶活力和稳定性的下降及酶蛋白的分解[10].由于优化后菌株表达了更大量的外源蛋白,因此菌体受到的毒性更大,酶活力也就受到了更大的影响.因此,诱导剂剂量及诱导时间也是对酶活力影响的重要因素.经过均匀设计得到的最佳诱导剂量为6.3 mmol,且诱导时间为2h,相较于传统的原始的培养条件(诱导剂量为1.0mmol,诱导时间为10h),增加了诱导剂量,减少了诱导时间,对目标蛋白的酶活力抑制降低.
2.4 优化条件的验证
通过上述均匀设计实验和二次多项式逐步回归,得到重组菌发酵产生目标蛋白最优培养条件,以及在此优化条件下ELPs-PDOR酶活力预测值为11.40mkat·L-1.在原始培养条件(诱导剂量为1.0 mmol·L-1,诱导温度为18℃,诱导时间为10h,装液量为60%)培养得到的ELPs-PDOR重组蛋白,其酶活力为5.15mkat·L-1;而在优化条件下,ELPs-PDOR的酶活力达到10.84mkat·L-1,与预测值的相对误差仅为5%.相较于优化前培养条件,优化后目标蛋白酶活力提高了110.21%.
大肠杆菌优化后,对其培养液的无细胞抽提液进行SDS-PAGE分析,结果如图1所示.由图1可知:优化培养条件前的ELPs-PDOR蛋白表达量为7.4%,而优化后的ELPs-PDOR蛋白表达量增加为24.1%,表达量是优化前的3.3倍,优化的培养条件对产生重组的ELPs-PDOR有明显的促进作用.验证实验表明:在优化条件下,培养的重组菌对于序列较长,含高度重复序列的重组基因的表达,以及对其酶活力性能等方面有显著的提高.
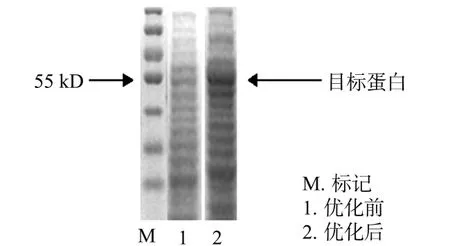
图1 优化前后大肠杆菌表达ELPs-PDOR的SDS-PAGE分析Fig.1 SDS-PAGE analysis of ELPs-PDOR before and after optimization
3 结束语
利用均匀设计实验和二次多项式逐步回归的方法,优化了菌种发酵产ELPs-PDOR重组蛋白培养条件:诱导剂为6.3mmol·L-1,诱导温度为30℃,诱导时间为2h,装液量为30%.此条件得到的酶活力可达到1.84mkat·L-1,为优化前的2.1倍,且ELPs-PDOR产量明显优于原始培养条件产生的重组蛋白量,具有较大的优势.
[1]URRY D W.Physical chemistry of biological free energy transduction as demonstrated by elastic protein-based polymers[J].J Phys Chem,1997,101(51):11007-11028.
[2]URRY D W.Free energy transduction in polypeptides and proteins based on inverse temperature transitions[J].Prog Biophys Mol Biol,1992,57(1):23-57.
[3]LIM D W,KIMBERLY T C,MACKAY J A,et al.Improved non-chromatographic purification of a recombinant protein by cationic elastin-like polypeptides[J].Biomacromolecules.2007,8(5):1417-1424.
[4]黄凯宗,李晶晶,李巍,等.类弹性蛋白多肽的从头设计、非色谱纯化及盐效应[J].生物工程学报,2011,27(4):553-658.
[5]LI Wei,FANG Bai-shan,ZHANG Guang-ya,et al.Codon optimization of 1,3-propanediol oxidoreductase expression in Escherichia coli and enzymatic properties[J].Electronic Journal of Biotechnology(in press).
[6]AHRENS K,MENZEL K,ZENG A,et al.Kinetic,dynamic,and pathway studies of glycerol metabolism by Klebsiella pneumoniae in anaerobic continuous culture(Ⅲ):Enzymes and fluxes of glycerol dissimilation and 1,3-propanediol formation[J].Biotechnol Bioeng,1998,59(5):544-552.
[7]陈宏文,聂金峰,方柏山.克雷伯杆菌1,3-丙二醇氧化还原酶的分离纯化及其酶学性质[J].华侨大学报:自然科学版,2009,30(1):62-66.
[8]MIROUX B,WALKER J E.Over-production of proteins in Escherichia coli:Mutant hosts that allow synthesis of some membrane proteins and globular proteins at high levels[J].J Mol Biol,1996,260(3):289-298.
[9]李志西.试验优化设计与统计分析[M].北京:科学出版社,1994.
[10]KWEON D H,HAN N S,PARK K M,et al.Overproduction of phytolacca insularis protein in batch and fed-batch culture of recombinant Escherichia coli[J].Process Biochem,2001,36(6):537-542.
Optimization of Cultivation Conditions of ELPs-PDOR Fusion Gene
WANG Wen-yan,ZHANG Guang-ya
(College of Chemical Engineering,Huaqiao University,Xiamen 361021,China)
The optimal parameters of cultivation condition of eastin-like polypeptides-1,3-propanediol oxidoreductase(ELPs-PDOR)by recombinant Escherichia coli were obtained through uniform-design and two polynomial stepwise regression.The optimal conditions of experiment were as follows:the medium volume was 30%,the induction concentration was 6.3mmol·L-1,the cultural temperature was 30℃and the induction temperature was 2h.On the basis of this,the yield of ELPs-PDOR is 3.3times higher than the primitive expression,and the enzymatic activity of PDOR is 10.84mkat·L-1which is 2.1times of the enzymatic activity of primitive expression.
eastin-like polypeptides;1,3-propanediol oxidoreductase;Escherichia coli;recombinant protein;uniformdesign
TQ 923;Q 554
A
1000-5013(2012)03-0296-04
2011-11-22
张光亚(1975-),男,教授,主要从事酶学与酶工程的研究.E-mail:zhgyghh@hqu.edu.cn.
国家自然科学基金资助项目(20806031);福建省自然科学基金资助项目(2009J01030);福建省高校新世纪优秀人才支持项目(07176C02)
(责任编辑:钱筠 英文审校:刘源岗)
