基于基因组DNA混合池的EST-SSR引物快速筛选方法研究
2012-12-29谢耀坚张党权谷振军
刘 果,谢耀坚,张党权 ,谷振军
(1.国家林业局桉树研究开发中心,广东 湛江 524022;2.中南林业科技大学;a.林业生物技术湖南省重点实验室;b.经济林育种与栽培国家林业局重点实验室;c.经济林培育与保护省部共建教育部重点实验室,湖南 长沙 410004)
基于基因组DNA混合池的EST-SSR引物快速筛选方法研究
刘 果1,谢耀坚1,张党权2,谷振军2
(1.国家林业局桉树研究开发中心,广东 湛江 524022;2.中南林业科技大学;a.林业生物技术湖南省重点实验室;b.经济林育种与栽培国家林业局重点实验室;c.经济林培育与保护省部共建教育部重点实验室,湖南 长沙 410004)
林木SSR标记的引物筛选工作量大,十分费时且消耗大量实验材料。采用基因组DNA混合池技术,将研究对象的若干个基因组DNA样品等量混合后,作为单一模板进行SSR的PCR扩增,能显著降低实验的工作量和成本。以选择的150对EST-SSR引物为对象,对6个桉树基因组DNA样品进行等量混合,进行PCR扩增筛选引物,结果表明,与常规筛选方法相比,基因组DNA混合池方法大幅度缩短了实验周期,显著减少了基因组DNA的消耗,可用于大量林木SSR引物的快速高效筛选。
基因组DNA混合池;引物筛选;EST-SSR,PCR
简单重复序列(Simple Sequence Repeats, SSR)又称微卫星序列,广泛分布于整个基因组。SSR作为DNA遗传标记具有高度变异性、共显性、重复性好、基因组覆盖率高等优异特性而被广泛应用[1-4]。近些年,利用大量公共ESTs数据库来开发EST-SSR标记已成为一种非常有效的途径。与传统SSR标记开发相比,基于数据库的EST-SSR标记开发无需构建基因组文库或EST文库,也无需再进行克隆测序,而且还充分利用了现有的数据资源,极大地降低了开发成本。EST-SSR来自于基因组的转录区域,因此表现的更为保守为,在其他相近物种中通用性更好[5-7],且与很多重要的功能位点连锁不平衡[8]。在大多数群体的遗传分析中表明,EST-SSR标记能很好的揭示高水平的遗传变异[7-11]。EST-SSR标记在构建遗传图谱、群体遗传多样性、进化遗传学已经种质资源收集等方面有很大的研究潜力。
桉树是世界上著名的经营利用树种之一,由于其生长速度快、轮伐期短、耐干旱、耐瘦瘠、适应性广,且用途广泛,经济效益高,现已被世界近百个国家与地区引种,遍布于中南海地区、东南亚、美洲和非洲,目前已经成为世界人工林的第二大类造林树种[12-13]。在桉属植物中SSR标记有高度的保守性和遗传多样性,所以对桉树进行SSR标记分析,非常有利于对桉树遗传结构、遗传多样性与分子标记辅助选择育种研究[14]。截至2012年5月,在公共数据库NCBI中已有16万多条桉树的EST序列,采用传统筛选方法进行大量SSR引物开发耗时费力。近年来在国际上逐渐被广泛应用的DNA混合池技术[15]能有效解决这个问题,可以最大限度的减少研究人员的工作量和试剂成本,使得大量引物的开发工作成为可能。DNA混合池技术是把多个样本的DNA溶液混合在一起进行PCR扩增、基因扫描、基因分型、最后用统计学方法对数据进行分析[16]。对组成混合池的样本数量没有固定的要求,少到几个、多到几千个样本都可以组成DNA混合池[17-18]。 DNA混合池技术近年来在国际上已被广泛应用于医学上各种疾病的研究,但应用于植物上的研究还甚少。本研究采用基因组DNA混合池技术,通过构建6个桉树样品的基因组DNA混合池快速筛选EST-SSR标记,并与常规方法筛选方法进行验证,为林木SSR引物的大量开发提供一种新的途径。
1 材料与方法
1.1 实验材料
设计的引物由英潍捷基(上海)贸易有限公司公司合成。本实验所用桉树叶片采集于国家南方重要种苗基地和广东省雷州林业局,其中细叶桉(1号)、巨桉(2号)和粗皮桉(6号)采集于广东省遂溪县南方国家级重要种苗基地,尾叶桉(3号)、赤桉(4号)和柠檬桉(5号)采集于广东省雷州市。
1.2 ET-SSR标记的引物选择
1.2.1 EST数据的获得和预处理
从美国NCBI公共数据库dbEST提供的桉树EST序列中下载了2010年和2011年更新(截至2011.11.7)的EST序列共16566条,其中巨桉16008条,蓝桉558条。
在开发之前需要对下载的数据进行预处理,包括去除一些低质量的片段(<100 bp)和带有少量载体序列及末端存在polyA/T“尾巴”的序列。这些数据会影响相关信息的分析。只有无冗余的EST数据库的SSR频率才能更真实的反应SSR在基因组转录部分的密度。本实验利用DNAStar软件下的SeqManⅡ操作系统对所有下载的序列分别树种进行去冗拼接和聚类。
1.2.2 EST-SSR位点的查找及引物的设计和评估
利用SSRHunter软件对Contigs序列进行SSR位点查找。查找标准为含有2,3,4,5,6碱基重复的SSR位点,其重复次数分别不小于9,6,5,4,3,即碱基重复至少要达到18个碱基长度。
对所有筛选出的含有SSR位点的Contigs序列利用Primer5.0软件进行引物设计。引物设计遵循3条基本原则:首先引物与模板的序列要紧密互补;其次引物与引物之间避免形成稳定的二聚体或发夹结构; 再次引物不能在模板的非目的位点引发DNA聚合反应(即错配)。最后利用Oligo6.0软件对所设计出的引物进行评价分析并筛选出评价高的引物,评价分析的标准为:1) ΔG值反映了序列与模板的结合强度,最好引物的ΔG值在5′端和中间值比较高,而在3′端相对低。2) Tm值曲线以选取72℃附近为佳,5′到3′的下降形状也有利于引物引发聚合反应。3) Frq曲线揭示了序列片段存在的重复机率大小。选取引物时,宜选用3′端Frq值相对较低的片段。
1.3 基因组DNA提取与混合池构建
桉树叶片样品的DNA提取采用QIAGEN公司的植物DNA基因组提取试剂盒(DNeasy Plant Mini kit)。 取 DNA 样 品 3.0 μL 与 1.0 μL 6×Loading Buffer混匀后用1.5%琼脂糖凝胶上电泳检测DNA样品浓度,检测染料为GelRedTM(100ml琼脂糖凝胶加3μL),用100 bp DNA ladder标定,初步判断2μL中含有DNA的量,然后根据检测结果稀释DNA,以供后续PCR扩增实验用,并保存于-20℃备用。
将6个桉树基因组DNA样品等量混合,构建一个基因组DNA混合池,取混合池样品3.0 μL与1.0 μL 6×Loading Buffer混匀后用1.5%琼脂糖凝胶上电泳检测其浓度,根据DNA样品浓度稀释混合池样品,供后续实验利用。
1.4 EST-SSR引物的筛选
1.4.1 SSR-PCR扩增体系优化及引物快速筛选
以各样品的基因组浓度一致的基因组DNA混合池为模板进,扩增体系参照李发根[19]设计的SSR引物筛选程序进行PCR优化和扩增。通过调整EST-SSR引物的Mg2+浓度和退火温度(Tm)来进行PCR优化,使扩增产物具有较好的特异性及较高的浓度。20 μL的PCR反应体系中,Mg2+浓度设置为1.5、2.0、2.5 mmol 3个梯度,退火温度(Tm)设置为50、56、60℃ 3个梯度。先将所有引物在Mg2+浓度为2.5 mmol、Tm为56℃条件下进行扩增,PCR扩增产物用1.5%的琼脂糖凝胶电泳检测和凝胶成像观察,扩增结果为明显的单一条带或两条条带,其中亮带较弱带强2倍以上的引物保留。再将剩下的引物继续在Mg2+浓度为2.0 mmol或1.5 mmol条件下进行第二轮扩增,保留结果较好的引物。最后改变Tm温度为50℃或60℃条件,并与三种Mg2+浓度相互交叉,反复筛选,每一轮筛选保留扩增结果较好的引物,筛选到最后仍然得不到较理想扩增产物的引物舍弃。具体的PCR扩增体系如表一所示。
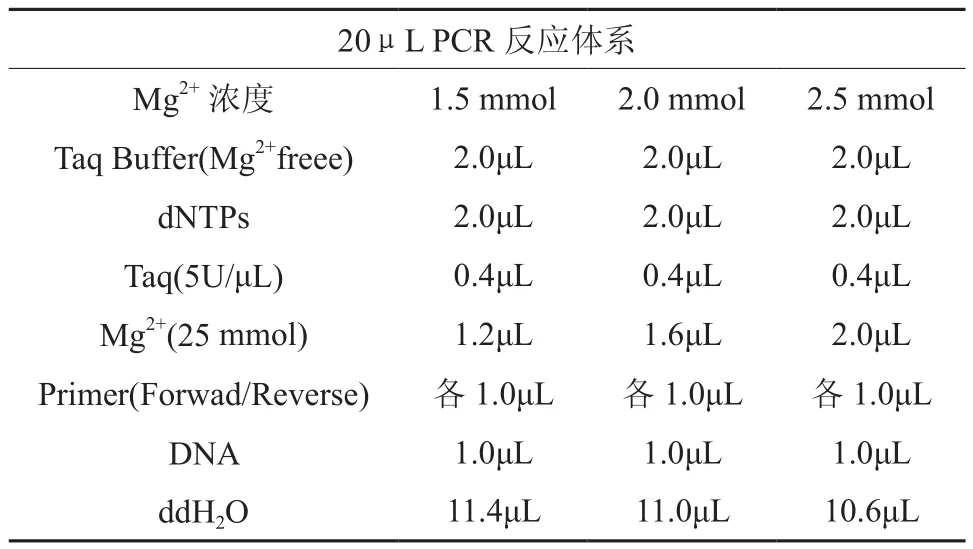
表1 20 μL PCR反应体系Table 120μL PCR reaction system
PCR扩增循环条件是: 94℃预变性4 min;94℃变性30 s,Tm退火(56℃、50℃、60℃)30s,72℃延伸1min(35个循环);ƒ72℃后延伸10 min。
2.4.2 常规EST-SSR引物筛选与快速筛选的对比分析
常规引物筛选是对代表性的基因组DNA样品(6个)进行筛选验证。本实验安排了在快速筛选实验中随机选出3对扩增结果好的引物(eSSR-GR029、eSSR-GR035、eSSR-GR074)进行常规PCR技术筛选验证;同时,在快速筛选实验中随机选出3对扩增结果不好的引物(eSSR-GL001、eSSR-GR010、eSSR-GR117)进行常规PCR筛选验证。扩增体系及循环条件与快速筛选实验相同。
2 结果与分析
2.1 基因组DNA样品浓度的检测
由QIAGEN公司的基因组提取试剂盒(DNeasy Plant Mini kit)提取的桉叶DNA质量较好,没有条带拖尾和杂带的现象。图1为6个桉树DNA样品和DNA混合池样品的电泳检测图,点样为 3.0 μL 与 1.0 μL 6 × loading buffer的混合。由图可得到,1、2、3号桉树DNA样品的浓度比较一致,4、5、6号桉树DNA样品的浓度比较一致。按照基因组DNA样品终浓度10 ng/μL,将1、2、3号样品稀释一倍后与4、5、6号样品等量混合,待用。
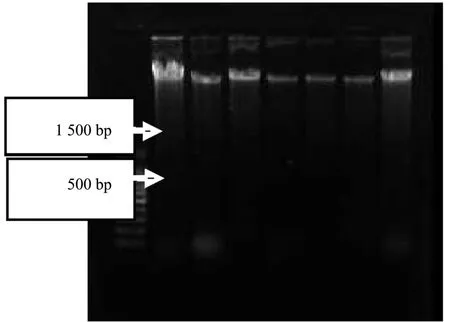
图1 桉叶基因组DNA提取电泳效果Fig. 1Eucalyptus genomic DNA detected by electrophoresis
2.2 EST-SSR引物设计
经软件分析含有SSR位点的可以用来设计引物的EST-Contigs序列一共有820条,其中蓝桉的序列有24条,巨桉的序列有796条。随机挑选部分设计引物,共设计EST-SSR引物150对用于本次实验,其中由蓝桉的EST序列设计的引物有9对,由巨桉的EST序列设计的引物有141对。EST-SSR引物长度均为18~22bp,引物设计的目的片段大小均为200~500 bp。
2.3 EST-SSR引物快速筛选的PCR体系优化
经 PCR 扩增后,取其产物 3.0 μL 与 1.0 μL 6× Loading Buffer混合用来电泳检测,选取扩增出明亮单一或者有两条条带,其中亮带比弱带强2倍以上的片段的引物,该引物可以直接用来后续实验,其余杂带较多,条带比较模糊的引物或者无条带的引物都要进行Mg2+浓度和Tm退火温度的优化。如图2,其中竖箭头指出的是较好的条带,横箭头指的是100 bp DNA Ladder。
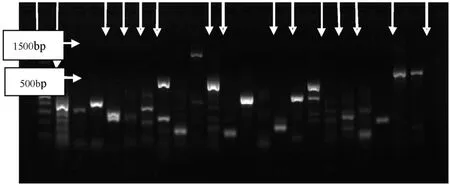
图2 部分EST-SSR引物快速筛选Fig. 2Fast screening result of partial EST-SSR primers
3.4 EST-SSR引物快速筛选分析
9轮PCR体系优化的筛选后,成功扩增的引物有136对,获得比较单一明亮的片段,扩增成功率达90.667%。说明了蓝桉和巨桉的EST序列在桉树种中的通用性较好。从表1中可以得到,第一轮筛选(2.5 mmol和56℃)和第二轮筛选(2.0 mmol和56℃),基本成功扩增的就达到了70.667%,与李发根[19]和郭勇[20]中的引物筛选结果相一致,其余扩增效果不好的进入下一轮的筛选。

表1 EST-SSR引物筛选结果Table 1The results of EST-SSR primer screening

图3 eSSR-GR029、eSSR-GR035、eSSR-GR074三对引物单一样本扩增效果Fig.3Three pairs primers (eSSR-GR029, eSSR-GR035, eSSR-GR074) single template PCR detected by electrophoresis

图4 eSSR-GL001、eSSR-GR010、eSSR-GR106引物单一样品扩增效果Fig.4 Primers(eSSR-GL001, eSSR-GR010, eSSR-GR106)single sample PCR detected by electrophoresis
2.5 引物常规筛选与快速筛选的比较分析
在与快速筛选相同体系和循环条件下,进行了6对引物的常规筛选验证,3对为快速筛选结果较优的引物,其余3对为快速筛选中结果较差的引物。如图3分别为eSSR-GR029、eSSR-GR035、eSSR-GR074用6个DNA样品PCR扩增后的产物检测图。图中,第一个白色箭头指的是3(a)为引物eSSR-GR029的单一样品逐一扩增图,引物在1、2、3、4、6号样品中扩增效果都比较好,在DNA混合样品中也有很好的扩增效果,但5号样品的扩增效果不佳;3(b)为引物eSSR-GR035的单一样品逐一扩增图,引物在6个样品中的扩增效果都比较好;3(c)位引物eSSR-GR074的单一样品逐一扩增图,引物在1、2、3、6号和DNA混合样品中都有很好的效果,但4号和5号样品基本检测不到条带。图4为 eSSR-GL005、eSSR-GR010、eSSR-GR106用6个DNA单一样品和DNA混合池逐一PCR扩增后的产物检测图,其中,白色箭头指的是DNA混合池样本PCR扩增后的产物。由图可看出,三对引物的扩增效果在单一样本和DNA混合池中均不理想,都没有出现明显的单一条带,得到的条带模糊不清晰,两种方法得到的结果一致,eSSR-GL005、eSSR-GR010、eSSR-GR106不是理想的引物。
3 结论与讨论
本研究基于基因组DNA混合池技术,建立了一种适用于EST-SSR标记特异性引物的快速高效筛选方法,适用率达到83.33%,工作量比常规单样本逐一筛选引物方法降低了约80%。
(1)基因组DNA混合池的构建。本实验中基因组DNA混合池是选用的6个桉树基因组DNA样品进行等量混合,保证了各个样品在混合池中的浓度和数量,使得构建出来的DNA混合池能够相等的代表其中的6个样品。经1.5%琼脂糖凝胶电泳检测得到的浓度也充分显示出构建的DNA混合池与六个样品的浓度和片段大小都比较一致,可以作为模板进行PCR扩增筛选引物。
(2)基因组DNA混合池的引物快速筛选的可靠性。本实验中采用的是6种常见桉树的DNA样品作为模板进行混合构建的DNA混合池,并以混合池为PCR扩增反应的模板进行EST-SSR引物的筛选。从筛选结果可以得出,DNA混合池模板PCR扩增的条带清晰明亮,与各样品分开PCR扩增得到的条带,片段大小和条带清晰度一致。在常规筛选实验中,三对较好引物的条带在1号(细叶桉),2号(巨桉),3号(尾叶桉),6号(粗皮桉)四种桉树中条带均单一明亮,通用性很好。4号(赤桉)在eSSR-GR074中没有条带出现,5号(柠檬桉)在eSSR-GR029和eSSR-GR074两对引物中都没有条带出现,适用率达到83.33%。
(3)基于基因组DNA混合池的EST-SSR引物快速筛选方法的效率及适应性。本方法的研究基础是基于基因组DNA混合池技术与简单重复序列(SSR)研究的相结合[14,18-19]。由于SSR标记技术属于特异性引物的分子标记技术[20],对DNA模板的要求不是很高,而且结果稳定可靠,重复性好[21-27],利用基因组DNA混合池代替各个样本进行PCR反应,满足SSR标记技术对DNA模板的要求。因此,具有特异性引物的分子标记技术,使用DNA混合池可以进行引物的快速筛选。
常规SSR引物的筛选,为保障有效性需对至少5个样本逐一进行PCR扩增反应,对于大量的引物设计,工作量非常大,且消耗大量基因组DNA样品。使用DNA混合池对大量引物进行筛选,工作量仅为原来工作量的约五分之一,能切实有效的提高工作效率,显著降低基因组DNA消耗和实验成本。
[1] Tautz D. Hypervariability of simple sequences as a general source for polymorphic DNA markers [J].Nucleic Acids Res.,1989,17(16): 6463-6471.
[2] Philippe J, Pierre J, Lagoda L.. Microsatellites, from molecules to populations and back [J]. Trends in Ecology & Evolution,1996, 11(10): 424-429.
[3] Powell W, Machray G C, Provan J. Polymorphisms revealed by simple sequence repeats [J]. Trends in Plant Science, 1996, 7(1):215-222.
[4] Gupta P K, Varshney. R K. The development and use of microsatellite markers for genetic analysis and plant breeding with emphasis on bread wheat [J]. Euphytica, 2000, 113(3): 163-185.
[5] Varshney R K, Graner A, Sorrells M E. Genic microsatellite markers in plants: features and applications [J]. Trends Biotechnol, 2005, 23(1): 48-55.
[6] Pashley C H, Ellis J R, McCauley D E, et al. EST databases as a source for molecular markers: lessons from Helianthus [J]. J.Hered., 2006, 97(4): 381-388.
[7] Ellis J R, Burke J M. EST-SSRs as a resource for population genetic analyses [J]. Heredity, 2007, 99(2): 125-132.
[8] Ayres N M, Larkin P D., Park W D, et al. Microsatellites and a single nucleotide polymorphism differentiate apparent amylose classes in an extended pedigree of US rice germplasm [J]. Theor.Appl. Genet., 1997, 94(6-7): 773-781.
[9] Eujayl I , Sorrells M E, Baum M, et al. Isolation of EST-derived microsatellite markers for genotyping the A and B genomes of wheat [J]. Theor. Appl. Genet., 2002, 104(2-3): 399-407.
[10] Simko I. Development of EST-SSR markers for the study of population structure in lettuce (Lactuca sativa L.) [J]. J. Hered.,2009, 100(2): 256-262.
[11] Kim K S, Ratcliffe S T, French B W, et al. Utility of EST-derived SSRs as population genetic markers in a beetle [J]. J. Hered.,2008, 99(2): 112-124.
[12] 欧阳乐军,曾富华. 桉树分子育种研究进展[J]. 分子植物育种, 2008,6(6): 1146-1152.
[13] 陈少雄,吴志华. 桉树分子育种研究进展[J]. 中南林业科技大学学报, 2008, 28(4): 42-48.
[14] 刘 果,谢耀坚. 分子标记技术在桉树育种中的应用研究[J].世界林业研究, 2012,25(3):19-25.
[15] 吕 筠,李立明. DNA混合分析技术[J]. 中国公共卫生,2002, 28(12): 1517-1519.
[16] 王 博,陈 刚. DNA混合池技术及其在精神分裂症遗传研究中的应用[J].精神医学杂志, 2007, 20(6): 404-407.
[17] 刘文文,郝转芳,翁建峰,等. 一种基于PCR技术混合样本高效定性筛查转基因阳性样本的方法[J]. 中国农业科学,2012, 45(2): 399-404.
[18] 何 柳,唐 迅,胡永华. DNA混合分析技术的单体型频率估计方法[J]. 中南大学学报(医学版), 2011, 36(5): 457-461.
[19] 李发根. 尾叶桉和细叶桉STS标记连锁图谱构建及生长状况QTL定位研究[D]. 北京:中国林业科学研究院, 2010.
[20] 郭 勇. 利用EST-CAPS标记构建桉树遗传图谱[D]. 北京:中国林业科学研究院, 2008.
[21] 祝全东,张党权,李晓云, 等. 油茶SRAP标记的PCR体系建立与优化[J]. 中南林业科技大学学报, 2010, 30(3): 57-62.
[22] 张党权,明付焕,江 平, 等. 绵毛优若藜冷诱导SSH 文库构建研究[J]. 中南林业科技大学学报, 2010, 30(2): 65-69.
[23] 明付焕,张党权,宋志丹, 等. 绵毛优若藜冷胁迫均一化全长cDNA文库的构建[J]. 中南林业科技大学学报, 2011, 31(7):169-173.
[24] 张党权,宋志丹,田晔林, 等. 抗逆模式灌木沙冬青的研究进展[J]. 中南林业科技大学学报, 2012, 32(2): 16-22.
[25] 韩 欣,张党权,王志平, 等. 基于SRAP的赣南油茶良种分子鉴别研究[J]. 中南林业科技大学学报, 2012, 32(3):147-151.
[26] 张党权,田 华,谢耀坚, 等. 细叶桉遗传多样性的ISSR分析[J]. 中南林业科技大学学报, 2009, 29(5): 91-94.
[27] 张党权,田 华,谢耀坚, 等. 桉树4个种遗传多样性的ISSR分析[J]. 中南林业科技大学学报, 2010, 30(1): 12-17.
Studies on quick screening of EST-SSR primers based on genomic DNA mixing pool
LIU Guo1, XIE Yao-jian1, ZHANG Dang-quan2, GU Zheng-jun2
(1.China Eucalypt Research Centre, Zhanjiang 524022, Guangdong, China; 2.a. Hunan Provincial Key laboratory of Forestry biotechnology; 2b. Key Laboratory of Non-wood Forest Products of State Forestry Ministry; 2c. Key Laboratory of Non-wood Forest trees and protection of Ministry of Education, Central South University of Forestry and Technology, Changsha 410004, Hunan, China)
The screening of SSR primers for forestry is a heavy research which is very time consuming and consume intensive experimental materials. By employing genomic DNA pooling technology, a method of mixing several genomic DNA templates equivalently as the single template for PCR, was used to quickly screen SSR primers, which will signif i cantly reduce the experimental workload and cost. Genomic DNA pooling constructed by equivalently mixing 6 Eucalyptus genomic DNA templates was used to screen 150 pairs of EST-SSR primers. The analytical results show that comparing with conventional screening method, the genomic DNA pooling method can greatly shorten experimental period and signif i cantly reduce the consumption of genomic DNA, suggesting that this method is a fast and eff i cient method for screening substantive SSR primers used in trees.
genomic DNA mixing pool; primer screening; EST-SSR; PCR
S792.39
A
1673-923X(2012)09-0124-06
2012-08-15
林业公益性行业专项(201104003);863课题(2011AA100202);湖南省科技厅重点项目(2010NK2008,2011NK2019);国家863计划课题(2011AA100203)
刘 果(1988-),女,硕士生,研究方向为林木遗传育种;Email:liuguopz@163.com
谢耀坚,男,博士,研究员,研究方向为林木遗传育种;Email:Xieyj@21cn.com
[本文编校:欧阳钦]
