基于SRAP的赣南油茶良种分子鉴别研究
2012-12-28张党权王志平宋志丹谷振军王金路邝先松田晔林
韩 欣,张党权,王志平,宋志丹,谷振军,王金路,邝先松,田晔林
基于SRAP的赣南油茶良种分子鉴别研究
韩 欣1,张党权1,王志平2,宋志丹1,谷振军1,王金路1,邝先松3,田晔林4
(1.中南林业科技大学 a.经济林育种与栽培国家林业局重点实验室;b.林业生物技术湖南省重点实验室;c.长沙市经济林工程技术研究中心,湖南 长沙 410004;2.抚州市林业科学研究所,江西 抚州 344000;3.赣州市林业科学研究所,江西 赣州 341000;4.北京农学院 园林学院,北京 102206)
油茶是我国南方的主要木本食用油料树种,赣南油茶良种是我国重要的油茶资源组成部分。利用SRAP(序列相关扩增多态性)标记技术,对我国油茶良种分子鉴别进行探索性研究。以14个赣南油茶良种为对象,提取叶片基因组DNA作为模板,筛选出稳定性高的引物组合; 采用SRAP-PCR的优化反应体系,对获得的清晰可重复的条带进行{0,1}数据转换,并根据SRAP的谱带特征建立了赣南油茶良种的分子鉴别体系,为油茶良种的分子鉴定提供了理论依据和技术基础。
赣南油茶;分子鉴别;SRAP;良种
油茶Camellia oleifera又名茶子树、油茶树,是山茶属植物中油脂含量较高且具有栽培经济价值的一类植物的总称[1],是我国特有的食用木本油料树种,主要分布在湖南、江西、广西等地。油茶适应力强,是我国南方低山和丘岗地区开发的主要经济林树种之一[2]。从油茶种仁中制备的茶油是一种高品质纯天然植物食用油,而茶枯和茶壳还可用于工业和医药原料。我国研究人员对油茶科学进行了长期的研究,在良种培育、丰产栽培、病虫害防治等方面取得了丰硕的研究成果,但在分子遗传学方面还处于起始阶段[3]。
DNA分子标记技术被广泛应用于林木遗传学研究[4]。近年来,一种基于外显子与内含子、启动子序列差异的新型分子标记技术——SRAP(Sequence Related Amplified Polymorphism,相关序列扩增多态性)[5],其原理是通过独特的引物设计,对目的基因组DNA的开放阅读框进行扩增[6]。由于SRAP所获得的多态性DNA条带大部分是与基因表达相关[7],因而特别适用于不同良种之间的分子遗传学研究[8]。SRAP的正向引物(长度为17 bp)由5′端的填充序列和3′端CCGG序列组成核心序列,3′端再设计3个选择性碱基对外显子进行特异性扩增[9]。SRAP的反向引物(长度为18 bp)5′端前11 bp为填充序列,与后面的AATT组成核心序列,3′端亦连接3个选择性碱基,对内含子区域、启动子区域进行特异性扩增[10]。因此,SRAP标记的扩增产物因不同物种、不同基因型的内含子、启动子与间隔长度不等而产生稳定的多态性[11]。因而,SRAP分子标记技术特别适用于不同种质特异性状遗传多样性及性状相关差异标记的研究[12-13]。
从20世纪60年代末开始,江西赣南地区系统开展了赣南油茶选优和良种培育的科学研究[14]。经数十年努力,先后选育出“GLS赣州油”、“GLR赣州油”等20多个油茶良种,其中,“GLS赣州油”是国家林木品种审定委员会审定的国家级良种,“GLR赣州油”是江西省林木品种审定委员会审定的林木良种,以及部分认定的油茶丰产良种[15]。多年来推广栽培证明,用这些赣南油茶良种造林,丰产期平均亩产茶油可达50 kg,最高达67.25 kg,为国内较优的油茶良种。将这些新品种、新技术应用于生产,有着巨大的经济、生态和社会效益[16]。
然而,目前油茶良种的形态标记极少[17],而且,即使可通过形态标记对林木进行初步鉴定,但当叶片、枝条、种子等材料脱离油茶植株个体后,良种无性系或种源在形态上很难区别,时常出现假冒良种,对油茶产业造成较大损失,制约了我国油茶良种化的进程,不利于油茶产业的又好又快发展。因而,需开发新型分子标记对赣南油茶良种进行离体材料的有效、快捷、准确鉴定[18]。本研究采用SRAP分子标记技术,以油茶叶片为材料,提取基因组DNA为模板,探索油茶良种的分子鉴别技术体系,可为油茶良种的推广栽培和分子标记辅助选择育种提供理论基础和技术支撑。
1 材料与方法
1.1 材料与试剂
基于Li and Quiros (2001)提出的SRAP引物设计原则[5],从16个SRAP正向引物和20个SRAP反向引物中,筛选出多态性好、条带特异、稳定性高的8对引物组合(见表1),由深圳华大基因科技有限公司合成。
采取江西省赣州市林业科学研究所、抚州市林业科学研究所提供的14个油茶高产优良品种(见表2)的新鲜叶片,洗净后于-70℃冷冻保存备用。所用试剂都为分子生物学纯。
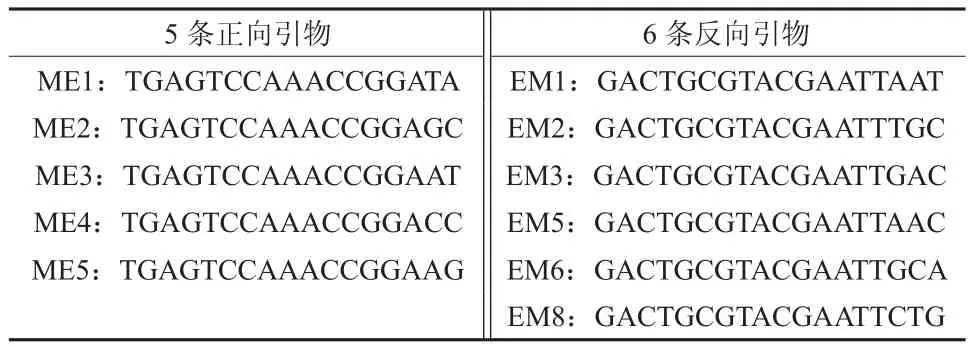
表1 SRAP标记8对引物组合的正反引物序列Table 1 Sequences of SRAP forward and reverse primers for 8 groups
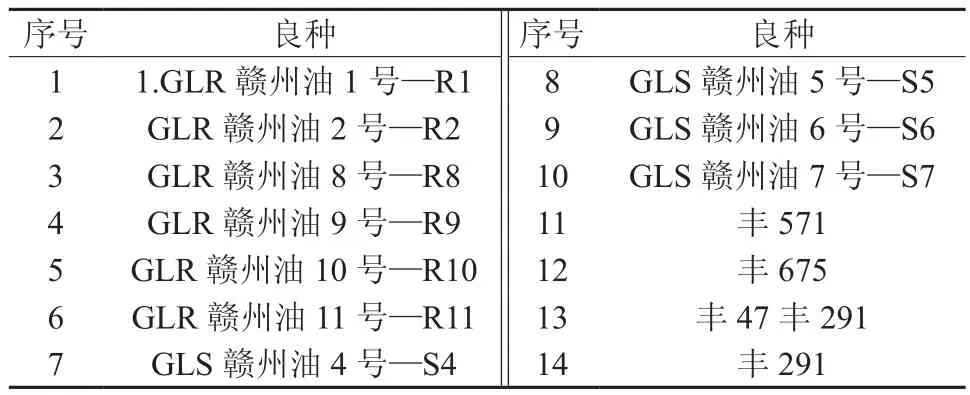
表2 赣南油茶14个良种及序号Table 2 The14 elite varieties of Camellia oleifear and their number
1.2 油茶基因组DNA的提取
采用改良的CTAB法[19-20]提取赣南油茶叶片的基因组DNA,即采集足量的油茶嫩叶,抽提时加入β-巯基乙醇,抽提过程中使用2.5%CTAB,最后用预冷的无水乙醇沉淀DNA,溶解后稀释至10 ng/μL,于零下20℃保存备用。
1.3 SRAP-PCR体系
本实验采用优化的SRAP-PCR反应体系[10,18]进行扩增,PCR反应总体积为20μL,循环程序为:94 ℃预变性 5 min;94 ℃变性 1 min,35 ℃复性1 min,72℃延伸1 min,5个循环;94 ℃变性1 min,50 ℃复性1 min,72 ℃延伸1 min,35个循环;最后72 ℃延伸10 min;4 ℃保存。扩增结果用1.2%琼脂糖凝胶电泳检测。
1.4 SRAP-PCR的数据处理
采用8对引物组合对14个油茶良种的模板DNA进行SRAP-PCR扩增。按照相同迁移位置上有扩增条带记为“1”、无则记为“0”的方法记录每个引物的电泳谱带,且仅记录清晰、重复性好的扩增条带,并将“1”、“0”数据转化成基因型数据,用于后续的分子鉴别分析。
2 结果与分析
2.1 油茶良种的8对SRAP引物组合扩增结果
通过改良CTAB法提取了高质量的赣南油茶14个良种叶片基因组DNA,采用8对SRAP引物组合进行PCR扩增,获得了22条多态性条带,长度范围150~1 200 bp(见图1)。
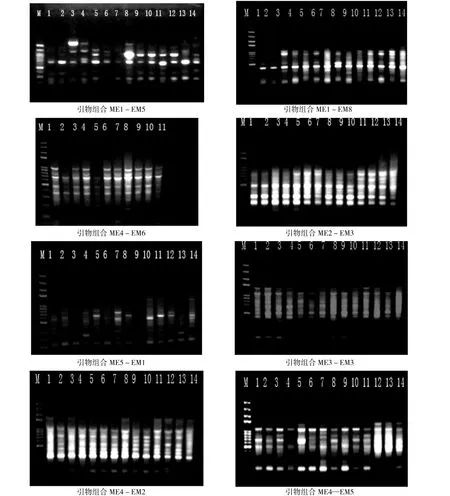
图1 赣南油茶14个良种的8个引物组合的SRAP-PCR扩增结果Fig.1 SRAP-PCR results of 14 elite C.oleifera cultivars by using 8 primer groups
2.2 赣南油茶良种的基因型及分子鉴别
根据SRAP引物的扩增图谱,绘制了油茶14个良种的分子标记基因型{0,1}数据表(见表3)。根据特异分离谱带确定了个品种的标记基因型,以此实现对油茶14个良种DNA水平上的鉴别。
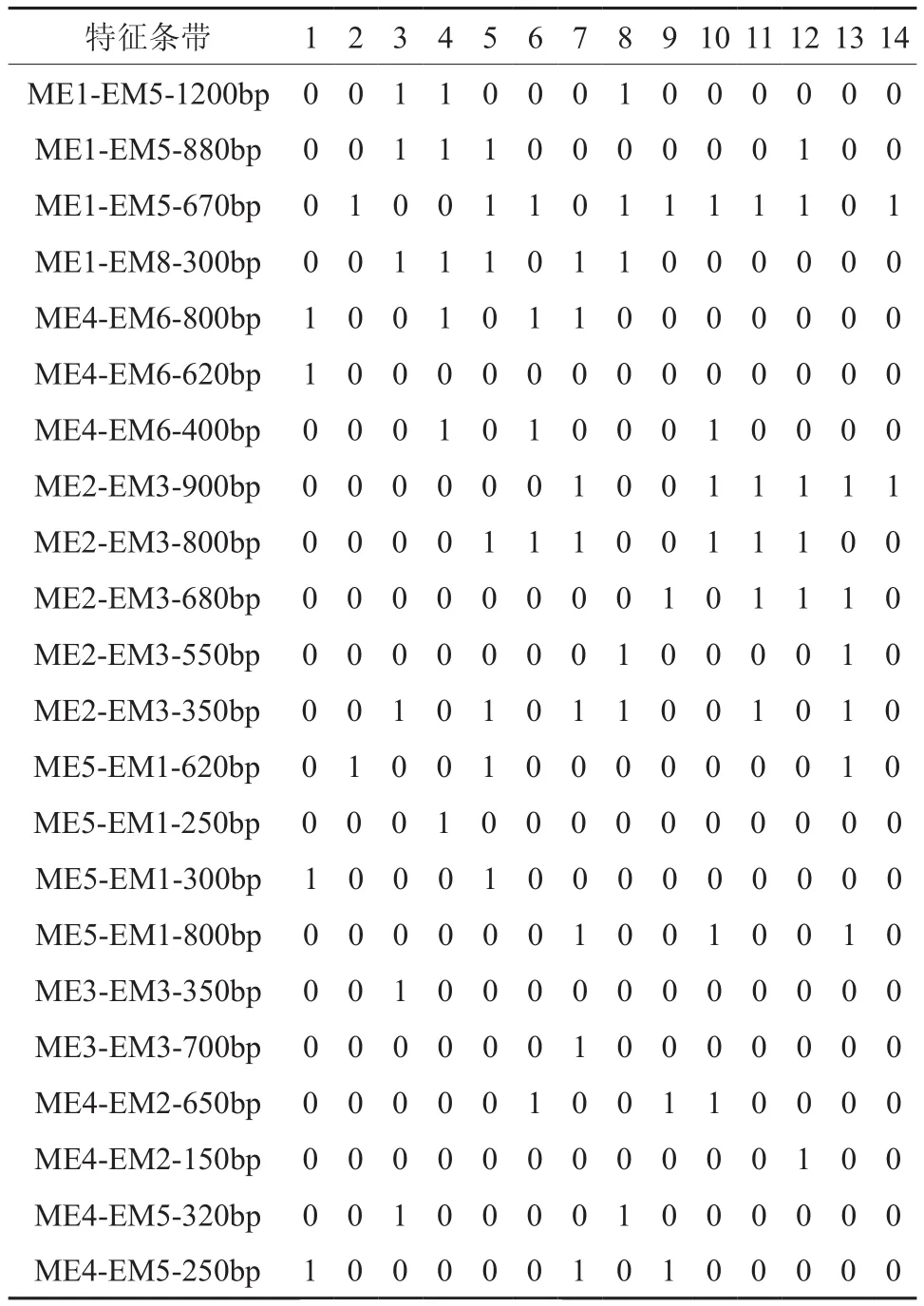
表3 赣南油茶14个良种的分子鉴别0-1基因型数据Table 3 The 0-1 genotype data for molecular identification of 14 Camellia elite cultivars from south Jiangxi province
1号样品的特征谱带组合是ME4-EM6-800bp,ME4-EM6-620bp,ME5-EM1-300bp,ME4-EM5-320bp。2号样品的特征谱带组合是ME1-EM5-670bp,ME5-EM1-620bp。3号样品的特征谱带组合 是 ME1-EM5-1200bp, ME1-EM5-880bp, ME1-EM8-300bp, ME2-EM3-350bp, ME3-EM3-350bp,ME4-EM5-320bp。4号样品的特征谱带组合是ME1-EM5-1200bp,ME1-EM5-880bp,ME1-EM8-300bp,ME4-EM6-800bp,ME4-EM6-400bp, ME5-EM1-250bp。5号样品的特征谱带组合是ME1-EM5-880bp,ME1-EM5-670bp,ME1-EM8-300bp,ME2-EM3-800bp,ME2-EM3-350bp, ME5-EM1-300bp,ME5-EM1-620bp。6号样品的特征谱带组合是ME1-EM5-670bp,ME4-EM6-800bp,ME4-EM6-400bp,ME2-EM3-800bp, ME4-EM2-650bp。7号样品的特征谱带组合是ME1-EM8-300bp,ME4-EM6-800bp,ME2-EM3-900bp,ME2-EM3-800bp,ME2-EM3-350bp,ME5-EM1-800bp,ME3-EM3-700bp,ME4-EM5-250bp,ME3-EM3-700bp。8号样品的特征谱带 组 合 是 ME1-EM5-1200bp,ME1-EM5-670bp,ME1-EM8-300bp,ME2-EM3-550bp,ME2-EM3-350bp,ME4-EM5-320bp。9号样品的特征谱带组合是ME1-EM5-670bp,ME2-EM3-680bp,E4-EM2-650bp,ME4-EM5-250bp。10号样品的特征谱带组合是 ME1-EM5-670bp,ME4-EM6-400bp,ME2-EM3-900bp,ME2-EM3-800bp,ME5-EM1-800bp,ME4-EM2-650bp。11号样品的特征谱带组合是ME1-EM5-670bp,ME2-EM3-900bp,ME2-EM3-800bp,ME2-EM3-680bp,ME2-EM3-350bp。12 号样品的特征谱带组合是ME1-EM5-880bp,ME1-EM5-670bp,ME2-EM3-900bp,ME2-EM3-800bp,ME2-EM3-680bp。13号样品的特征谱带组合是ME2-EM3-900bp,ME2-EM3-680bp,ME2-EM3-550bp,ME2-EM3-350bp,ME5-EM1-250bp,ME5-EM1-800bp。14号样品的特征谱带组合是ME1-EM5-670bp,ME2-EM3-900bp。
其中,ME4-EM6-620bp为1号样品特有的特征条带,ME5-EM1-250bp为4号样品特有的特征条带,ME3-EMs3-350bp为3号样品特有的特征条带,ME3-EM3-700bp为7号样品特有的特征条带,ME4-EM2-150bp为12号样品特有的特征条带,可以作为简单鉴别依据。
3 结论与讨论
采用优化的SRAP-PCR反应体系,对赣南14个油茶良种进行了SRAP-PCR分析,从16个正向引物和20个反向引物中筛选出条带特异、多态性好、稳定性高的8对引物组合。利用这些引物组合对赣南油茶14个良种扩增出22条多态条带,并转换成{0,1}基因型数据,初步建立了这些油茶良种的分子鉴别体系。
20世纪90年代以来,国内外许多研究者已将RAPD、ISSR、SSR等分子标记应用于农作物(如水稻、小麦等)、果树(如苹果等)、林木(如杨树、桉树等)和园林花卉(如月季、菊花等)的品种分子分类和分子鉴定[21],这些研究均以谱带的差异作为鉴别的依据[22]。而SRAP是基于外显子与内含子、启动子序列差异的一种新型分子标记技术,通过独特的引物设计对开放阅读框进行扩增,所获得的多态性DNA条带大部分是与基因表达相关,因而比RAPD、ISSR等标记更适用于不同良种之间的分子遗传学研究[23],特别是在油茶遗传图谱构建、油茶数量性状位点定位、分子标记辅助选择育种、综合图谱构建和基因图位克隆提供有效的框架,为其它相关分子遗传学研究提供有益参考。
目前,可应用于油茶优良种质分子鉴别研究的SRAP引物较少,因而还需加大引物开发力度[24]。作为随机引物标记的一种,SRAP的稳定性不如特异引物标记如SSR[25],因而,需继续进行将SRAP标记开发成高稳定性的SRAP-SCAR特异标记,以实现我国油茶良种的精准分子鉴定,从分子水平上提升我国油茶良种化进程。
[1] 胡芳名, 谭晓风, 刘惠民.中国主要经济树种的栽培与利用[M].北京:中国林业出版社, 2006: 370-383.
[2] 庄瑞林.中国油茶(第2版) [M].北京:中国林业出版社,2008: 1-13.
[3] 邹 峰, 谭晓风, 袁德义, 等.RAPD分子标记及其在油茶遗传育种研究中的应用(综述)[J].亚热带植物科学, 2009,38(1): 70-73.
[4] 张党权, 田 华, 谢耀坚, 等.桉树4个种遗传多样性的ISSR分析[J].中南林业科技大学学报,2010, 30(1): 12-17.
[5] Li G, Quiros C F.Sequence-related amplified polymorpgism(SRAP) a new marker system based on a simple PCR reaction:its application to mapping and gene tagging in Brassica [J].Theor.Appl.Genet., 2001, 103: 455-461.
[6] Riaz A, Li G, Quresh Z, et al.Genetic diversity of oilseed Brassica napus lines based on sequence-related amplified polymorphism and its relation to hybrid performance [J].Plant Breeding, 2001, 120(5): 411-415.
[7] 郭大龙, 张君玉, 李 猛, 等.葡萄SRAP反应体系优化及引物筛选[J].基因组学与应用生物学, 2010, 29(2): 379-387.
[8] Ferriol M, Pico B, Nuez F, et al.Genetic diversity of a germplasm collection of Cucurbita pepo using SRAP and AFLP markers [J].Theor.Appl.Genet., 2003, 107(2): 271-282.
[9] Yu M Y, Ma B, Luo X, et al.Molecular Diversity of Auricularia polytricha Revealed by Inter-Simple Sequence Repeat and Sequence-Related Amplified Polymorphism Markers [J].Curr.Microbiol., 2008, 56: 240-245.
[10] 祝全东, 张党权, 李晓云, 等.油茶SRAP标记的PCR体系建立与优化[J].中南林业科技大学学报, 2010, 30(3): 57-62.
[11] Primmer C R, Ellgren H.Patterns of molecular evolution in avian microsatellite [J].Mol.Biol.Evol., 1998, 15(8): 997-1008.
[12] Gunasekare M T K.Applications of molecular markers to the genetic improvement of Camellia sinensis L.(tea)-A review [J].The Journal of Horticultural Science & Biotechnology, 2007,82(2): 161-169.
[13] Han X Y, Wang L S.Molecular Characterization of Tree Peony Germplasm Using Sequence-Related Amplified Polymorphism Markers [J].Biochem.Genet., 2008, 46:162-179.
[14] 刘作梅, 赵 倩, 谭晓风, 等.赣南油茶高产优良无性系的ISSR分子鉴别[C]// 第二届中国林业学术大会—S2功能基因组时代的林木遗传与改良论文集, 2009.
[15] 陈永忠, 张智俊, 谭晓风.油茶优良无性系RAPD的分子鉴定[J].中南林学院学报, 2005, 25(4): 40-45.
[16] 张国武, 钟文斌, 乌云塔娜, 等.油茶优良无性系的ISSR分子鉴别[J].林业科学研究, 2007, 20(2): 278-282.
[17] 沈程文, 黄意欢, 黄建安, 等.SRAP标记体系优化及在茶树种质资源研究中的应用[J].经济林研究, 2009, 27(3): 38-41.
[18] 彭邵锋, 张党权, 陈永忠, 等.14个油茶良种遗传多样性的SRAP分析[J].中南林业科技大学学报, 2011, 31(1): 80-85.
[19] 张党权, 明付焕, 江 平, 等.绵毛优若藜冷诱导SSH 文库构建研究[J].中南林业科技大学学报, 2010, 30(2): 65-69.
[20] 明付焕, 张党权, 宋志丹, 等.绵毛优若藜冷胁迫均一化全长cDNA文库的构建[J].中南林业科技大学学报,2011, 31(7):169-173.
[21] Pasquali M, Komjati H, Lee D, et al.SRAP Technique Generates Polymorphisms in Puccinia striiformis Isolates[J].Journal of Phytopathology, 2010, 10: 708-711.
[22] Rahman M, Li G Y, Schroeder D, et al.Inheritance of seed coat color genes in Brassica napus (L.) and tagging the genes using SRAP, SCAR and SNP molecular markers [J].Molecular Breeding, 2010, 10: 439-454.
[23] Tang L H, Xiao Y, Li L, et al.Analysis of Genetic Among Chinese Auricularia auricula Cultivars Using Combined ISSR and SRAP Markers [J].Current Microbiology, 2010, 8: 132-141.
[24] Ren X P, Huang J Q, Liao B S, et al.Genomic affinities of Arachis genus and interspecific hybrids were revealed by SRAP markers [J].Genetic Resources and Crop Evolution, 2010, 8:903-914.
[25] Chen M M, Feng F J, Sui X, et al.Construction of a framework map for Pinus koraiensis Sieb.et Zucc.using SRAP, SSR and ISSR markers [J].Trees-Structure and Function, 2010, 24(4):685-693.
SRAP-based molecular identification of elite cultivars of Camellia oleifera from south Jiangxi province
HAN Xin1, ZHANG Dang-quan1, WANG Zhi-ping2, SONG Zhi-dan1, GU Zheng-jun1, WANG Jin-lu1, KUANG Xian-song3, TIAN Ye-li4
(1 a.Key Lab.of Non-wood Forest Products of State Forestry Administration; 1b.Hunan Provincial Key Laboratory of Forestry Biotechnology; 1c.Non-wood Engineering Institute of Changsha; Central South University of Forestry and Technology, Changsha 410004, Hunan, China; 2.Fuzhou Forestry Research Institute, Fuzhou 344000, Jiangxi, China; 3.Ganzhou Forestry Research Institute,Ganzhou 341000, Jiangxi, China ; 4.School of Landscape Architecture, Beijing University of Agriculture, Beijing 102206, China)
Camellia oleifera is one of main tree species of woody edible oil in South China, and important elite cultivars resource in southern Jiangxi province.The researches on molecular identification of the elite cultivars in south Jiangxi were carried out by SRAP(sequence related amplification polymorphism).By Using the leaf genomic DNA extracted from 14 elite Camellia oleifera cultivars in south Jiangxi province, the improved CTAB method as template, the high-stable primer groups were selected out based on the improved SRAP-PCR system, which can bring specific DNA bands, good polymorphism and high stability.According to the SRAP-based band pattern from these primer groups, the polymorphic DNA bands were transformed in range of {0,1} data, which was used to establish the primary system of molecular identification of elite cultivars from south Jiangxi, thus providing theoretic basis and technique foundation of molecular identification of Chinese elite Camellia oleifera cultivars.
Camellia oleifera; molecular identification; SRAP; elite cultivars
S794.4
A
1673-923X(2012)03-0147-05
2011-10-20
国家“十一五”科技支撑计划课题(2009BADB1B02,2009BADB1B10)
韩 欣(1983—),女,硕士研究生,研究方向为植物分子遗传学
张党权(1976—),男,副教授,博士,研究方向为植物分子生物学与林产品加工利用;E-mail:zhangdangquan@163.com
[本文编校:谢荣秀]
