miR-144/451基因簇调控网络的生物信息学分析
2012-12-25聂伟伟陈龙邦张辰宇李冬海管晓翔
聂伟伟,唐 林,韦 凤,陈龙邦,张辰宇,朱 琳,李冬海,管晓翔
(基础研究)
miR-144/451基因簇调控网络的生物信息学分析
聂伟伟,唐 林,韦 凤,陈龙邦,张辰宇,朱 琳,李冬海,管晓翔
目的 生物信息学方法是预测miRNA二级结构及miRNA靶基因的有用工具。miRNA、转录因子和靶基因之间的调控网络参与多种生理、病理活动的分子机制。文中用生物信息学方法预测mir-144/451基因簇的调控网络结构。 方法运用多个在线数据库,研究miR-144/451基因簇的上游转录因子及下游靶基因间的多个反馈和前反馈环路,对参与miR-144/451基因簇调控环路的基因进行功能聚类分析,绘制miR-144/451基因簇的核心调控网络图。结论miR-144/451基因簇与其上游转录因子GATA1有17个共调控的靶基因,与生物体蛋白磷酸化、性腺发育、上皮细胞分化、葡萄糖代谢等生化学过程密切相关,并参与胰岛素信号通路、哺乳动物雷帕霉素靶蛋白(mammalian target of rapamycin,mTOR)信号通路、血管内皮生长因子(vascular endothelial growth factor,VEGF)信号通路、自然杀伤细胞介导的细胞毒作用、Toll样受体信号通路、ErbB信号通路及黄体酮调节卵细胞成熟等体内重要生理过程。结果对miR-144/451基因簇调控网络的生物信息学分析有助于理解其在细胞发育、细胞周期调控和肿瘤发生过程中的作用机制,为后续实验提供了理论依据。
微小RNA;miR-144/451基因簇;生物信息学;反馈环路;基因功能
0 引 言
微小RNA(microRNA,miRNA)是具有基因转录后水平调控作用的非编码单链小RNA[1],在细胞增殖、分化、发育以及细胞凋亡等生理过程以及疾病发生发展中发挥重要作用[2]。miRNA的表达水平及活性在不同组织及不同时间内表现出明显差异[3],其确切机制尚未明确。
靶基因的转录水平和转录后水平分别受转录因子和miRNA调控,同时miRNA的表达水平依赖于转录因子的调控[4]。因此靶基因、转录因子和miRNA三者之间存在由多条反馈环路和前反馈环路构成的复杂的调控网络[5]。miRNA参与的反馈环路是指miRNA可以反作用于调控其自身表达的转录因子。miRNA参与的前反馈环路是指miRNA上游的转录因子除了通过miRNA间接调控靶基因,还可以直接调控靶基因,即miRNA和其上游转录因子共调控同一靶基因。
miR-144/451基因簇编码miR-144和miR-451 2个miRNA,参与基因调控、哺乳动物红细胞生成[6]等生理活动和白血病、淋巴瘤[7]、肺癌[8]等肿瘤发生发展,并与药物耐药现象密切相关[9]。本研究采用生物信息学方法,综合多个在线数据库的信息,探索miR-144/451基因簇与上游转录因子(transcription factor,TF)及下游靶基因间的调控关系,分析寻找miR-144/451基因簇与其上游转录因子之间存在的反馈和前反馈环路,并对其进行功能综合分析,精确地绘制出该基因簇调控网络,深入了解miR-144/451基因簇相关疾病的发生发展机制,为临床治疗提供依据。
1 材料与方法
1.1 在线数据库本研究使用的数据库如下:转录因子数据库TransmiR数据库(http://202.38.126.151/hmdd/mirna/tf);转录因子数据库 TRANSFAC (http://www.gene-regulation.com/pub/databases);靶基因数据库miRWalk(http://www.ma.uni-heidelberg.de/apps/zmf/mirwalk);启动子序列数据库UCSC(http://genome.ucsc.edu);jaspar-core数据库(http://jaspar.cgb.ki.se)。功能聚类分析采用DAVID基因功能富集分析数据库(http://david.abcc.ncifcrf.gov)。
1.2 方法
1.2.1 获取miR-144/451基因簇的上游TF-1从调控microRNA的TF数据库TransmiR[10]获得调控miR-144/451基因簇的上游TF-1。
1.2.2 获取miR-144/451基因簇的靶基因从综合性数据库miRWalk[11]获得经实验证实的 miR-144/451基因簇各个miRNA成员靶基因。
1.2.3 获取miR-144/451基因簇靶基因的转录因子用加州大学圣克鲁斯分校(University of California Santa Cruz)的基因参考序列数据库 UCSC搜索miR-144/451基因簇各成员靶基因的上游-2000到200 bp启动子序列中转录因子结合位点,选取相关系数分值大于0.9(最大相关系数分值为1)的转录因子,并与jaspar-core数据库及TRANSFAC数据库中人种属的转录因子进行校对,得到miR-144/451基因簇靶基因的TF-2。
1.2.4 获取参与miR-144/451基因簇调控网络的反馈环路和前反馈环路比较调控miR-144/451基因簇的上游转录因子和调控miR-144/451基因簇靶基因的转录因子,获得重叠转录因子,从而获得既受重叠转录因子调控又受miR-144/451基因簇调控的共调控靶(join target)基因。综合分析获得参与miR-144/451基因簇调控网络的反馈和前反馈环路。
1.2.5 获取miR-144/451基因簇调控网络用DAVID数据库分析参与miR-144/451基因簇反馈和前反馈环路中的基因的功能,绘制miR-144/451基因簇的核心调控网络图。

图1 miR-144/451基因簇调控网络图Figure 1 Flow shart of the miR-144/451 cluster mediated regulation network
2 结 果
2.1 miR-144/451基因簇的上游转录因子转录因子通过与靶基因的启动子区结合在转录水平调控其表达。转录因子受miRNA调控,而miRNA的表达水平依赖于转录因子为主的转录调控机制,所以转录因子和miRNA之间存在协同作用和相互调控的关系。TransmiR数据库收集了大量经实验验证的调控miRNA转录因子。我们利用TransmiR数据库得到miR-144/451基因簇的4个上游转录因子GATA1、GATA4、SMAD3、Tcf3。
2.2 miR-144/451基因簇调控的靶基因通过结合靶基因mRNA的3'-UTR端,miRNA抑制mRNA表达,并促进其降解,从而在转录后水平调控靶基因的表达[12]。miRWalk数据库是miRNA的综合数据库,数据来源于软件预测和文献挖掘,收录了包括miRNA靶基因以及 miRNA与信号通路、疾病及组织特异性表达相关的数据信息。利用miRWalk数据库得到miR-144/451基因簇各miRNA成员的靶基因共103个,其中miR-144和miR-451共同调控的靶基因有17个,见表1。

表1 miR-144/451基因簇的靶基因Table 1 Target genes of the miR-144/451 cluster
2.3 调控miR-144/451基因簇靶基因的转录因子采用一致序列匹配方法在Match数据库搜索调控miR-144/451基因簇的103个靶基因的转录因子,见表1。其中26个基因未找到对应的转录因子。为了避免过多的假阳性结果,我们只筛选出转录因子结合位点相关系数分值大于0.9的转录因子,最大相关系数为1。结合JASPAR数据库和TRANSFAC数据库收集的294个人的转录因子。筛选得到的调控miR-144/451基因簇转录因子共17个,分别为Arnt、Ap-4、c-Myb、ER、GATA1、GATA2、GATA3、HLF、MyoD、NKx2-5、Oct-1、P54、P65、pax6、SOX9、USF、YY1。
2.4 miR-144/451基因簇调控网络中反馈和前反馈环路转录因子既可调控相关靶基因,又可调控miRNA。将调控miR-144/451基因簇的转录因子和调控miR-144/451基因簇靶基因的转录因子进行对比,得到既调控miR-144/451,又调控miR-144/451基因簇靶基因的重叠转录因子GATA1。同时筛选出GATA1所调控的miR-144/451基因簇的靶基因共14个,见表2,进而得到miR-144/451基因簇各成员靶基因和GATA1参与的前反馈环路。

表2 转录因子、miR-144/451和共调控靶基因组成的前反馈环路Table 2 Feedforward loops among TFs,miR-144/451 and joint targets
2.5 参与miR-144/451基因簇反馈和前反馈环路的基因功能分析结果将参与miR-144/451基因簇反馈和前反馈环路的14个基因映射到DAVID数据库,进行基因分类分析(gene ontology,GO)和KEGG信号通路功能聚类分析,结果见表3、表4,可以看出这14个基因参与调节蛋白磷酸化、性腺发育、上皮细胞分化、葡萄糖代谢等生化过程,并参与胰岛素信号通路、mTOR信号通路、VEGF信号通路、自然杀伤细胞介导的细胞毒作用、Toll样受体信号通路、ErbB信号通路及黄体酮调节的卵细胞成熟等体内重要生理过程。因此证明miR-144/451基因簇和上游转录因子的共调控靶基因在多种细胞生物学过程中发挥重要作用。

表3 miR-144/451基因簇调控的靶基因的GO功能分析结果Table 3 Gene ontology analysis on the targets of the miR-144/451 cluster
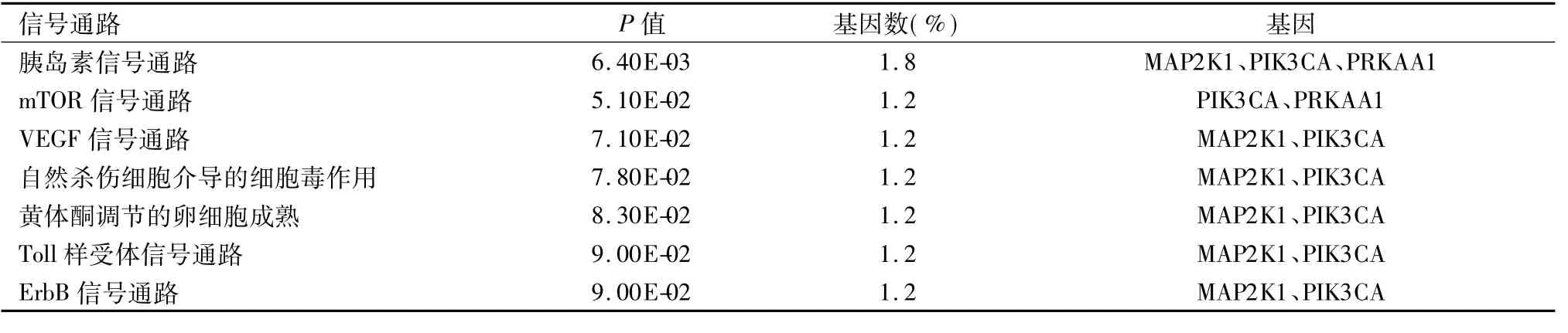
表4 miR-144/451基因簇调控的靶基因的KEGG pathway分析结果Table 4 KEGG pathway analysis on the targets of the miR-144/451 cluster
2.6 构建miR-144/451基因簇的核心调控网络综合以上分析结果,绘制出以miR-144/451基因簇的核心调控网络图。

图2 miR-144/451基因簇的核心调控网络Figure 2 Core regulation network for the miR-144/451 cluster
3 讨 论
miRNA在细胞增殖、分化、发育以及细胞凋亡等生理过程以及肿瘤形成等病理过程中发挥重要作用。生物信息学是处理大量生物信息的新的研究手段,应用生物学、数学和计算机知识,利用多种数据库,研究和分析生命体细胞、组织、器官的生理、病理和药理过程中的各种生物信息,从而全面、深刻地揭示生命科学的本质问题。生物信息学在miRNA二级结构预测、miRNA靶基因的预测等研究中起重要作用。本研究利用生物信息学技术揭示了miR-144/451与其上游转录因子及相应基因组成的复杂调控网络,有助于进一步研究该miRNA分子功能的理论和方法。
转录因子结合靶基因调控区的转录因子结合位点,既可激活基因的表达,也可抑制基因的表达。因此,转录因子既可以启动某些特定基因的表达,使细胞向特定方向分化;也可以关闭某些特定基因表达,解除细胞向特定细胞分化所受到的抑制作用,使细胞向特定方向分化。miRNA的作用方式与转录因子作用相似[13]。真核生物的转录因子可以与同源性的顺式作用元件相结合,但转录因子和这些顺式作用元件结合的亲合力并不相同。因此,某个转录因子可与多个顺式作用元件结合。反之,同一顺式元件也能和多个转录因子结合。miRNA与mRNA 3' UTR区的结合并不需要完全配对。因此,某个miRNA可与多个同源性结合位点结合,而同一顺式作用元件即miRNA结合位点也可以与多个miRNA相互作用[14]。研究发现某些miRNA可抑制[15-16]或者激活[17]其mRNA靶基因的表达水平,因而miRNA可在翻译水平发挥调节作用。随着对miRNA的深入研究,发现miRNA基因的表达受到复杂的转录因子调控,这种调控作用可以是直接作用,也可以是某种转录因子通过其他转录因子间接调控其相应的miRNA[18]。许多转录因子自身的表达受到 miRNA的调控。因此转录因子和miRNA是基因表达调控网络的重要组成部分和枢纽,协同调控靶基因的表达。转录因子和miRNA构成基因表达调控的反馈环路和前反馈环路。miRNA参与的反馈环路是指miRNA可以反作用于调控其自身表达的转录因子。反馈环路可有效降低细胞信号网络的噪音和波动,或者促进细胞从一种状态快速转换成另一种状态。miRNA参与的前反馈环路是指miRNA上游的转录因子除了通过miRNA间接调控靶基因,还可以直接调控靶基因,即miRNA和其上游转录因子共调控同一靶基因。前反馈环路又分为一致前反馈环路和不一致前反馈环路。在一致前馈环路中,TF通过调控miRNA能更有效地向下游共调控靶基因传递信号。在不一致前馈环路中,miRNA则可阻止噪音刺激引发的网络响应以及防止信号的过度激活。这些前反馈与反馈环路间的相互协同作用便于基因调控网络系统信号的放大、减弱和持续性,进而指导细胞对外界刺激做出最适应答[5]。
Re等[19]于2009年对人类转录因子、microRNA和基因之间存在的调控网络进行总结,为该构想理论提供了更多依据。c-Myc、E2F和miR-17-5p/miR-20a调控环路是经实验验证的调控网络之一。c-Myc可直接激活细胞周期相关转录因子(transeription factor1/3,E2F1/3)的转录,同时又可通过miR-17-5p和miR-20a转录后抑制 E2F1/3,进而精细调控E2F1/3蛋白表达水平,而E2F1/3也可激活髓细胞增生原癌基因(myelocytomatosis oncogene,MYC)和miR-17-5p的转录[20]。这些调控网络的存在一定程度上证实了miR-144/451基因簇调控网络存在的可能性。
GATA1是重要的造血转录因子,在网织红细胞和巨噬细胞的分化成熟中起着关键作用,并调控与该过程密切相关的基因[21]。我们的分析结果显示GATA1与miR-144/451基因簇共同调控的靶基因广泛地参与细胞的多种生物学过程。实验证明GATA1和miR-144/451之间的调控作用在红细胞生成中起关键作用[22]。这些被共调控的靶基因的具体功能尚有待实验研究进一步证实。
通过本研究我们得到miR-144/451基因簇与其上游转录因子及下游共调控靶基因构成精密复杂的基因表达调控网络,协调细胞对外界刺激做出适当的应答,进而维持细胞的正常功能。同时miR-144/451基因簇调控网络中的反馈和前反馈环路之相互间的稳定关系是维持细胞正常功能的关键因素,否则将会引起多个信号通路发生改变,甚至导致细胞的恶性转化。多项研究结果证明miR-144/451基因簇的功能异常可导致红细胞生成障碍,进而引起多种血液疾病[22-23]。因此,对miR-144/451基因簇调控网络进行详细而全面的研究,不仅能揭示miR-144/451基因簇的生物学功能,而且为阐明与其相关疾病的发病机制提供了新的理论基础,也为临床疾病治疗提供了新的治疗靶点[24]。
[1] Zamore PD,Haley B.Ribo-genome:the big world of small RNAs[J].Science,2005,309(5740):1519-1524.
[2] 程 文,高建平,张征宇.微小RNA与肿瘤相关性研究[J].医学研究生学报,2011,24(2):203-207.
[3] Lagos-Quintana M,Rauhut R,Yalcin A,et al.Identification of tissue-specific microRNAs from mouse[J].Curr Biol,2002,12 (9):735-739.
[4] Hobert O.Gene regulation by transcription factors and microRNAs[J].Science,2008,319(5871):1785-1786.
[5] Martinez NJ,Ow MC,Barrasa MI,et al.A C.elegans genomescale microRNA network contains composite feedback motifs with high flux capacity[J].Genes Dev,2008,22(18):2535-2549.
[6] Rasmussen KD,Simmini S.Abreu-Goodger C,et al.The miR-144/451 locus is required for erythroid homeostasis[J].J Exp Med,2010,207(7):1351-1358.
[7] Fabbri M,Croce CM,Calin GA.MicroRNAs in the ontogeny of leukemias and lymphomas[J].Leuk lymphoma,2009,50(2): 160-170.
[8] Wang R,Wang Z,Yang J,et al.MicroRNA-451 functions as a tumor suppressor in human non-small cell lung cancer by targeting ras-related protein 14(RAB14)[J].Oncogene,2011,30(23): 2644-2658.
[9] Zheng T,Wang J,Chen X,et al.Role of microRNA in anticancer drug resistance[J].Int J Cancer,2010,126(1):2-10.
[10] Wang J,Lu M,Qiu C,et al.TransmiR:a transcription factormicroRNA regulation database[J].Nucleic Acids Res,2010,38 (1):D119-D122.
[11] Wasserman WW,Sandelin A.Applied bioinformatics for the identification of regulatory elements[J].Nat Rev Genetics,2004,5 (4):276-287.
[12] Lim LP,Lau NC,Garrett-Engele P,et al.Microarray analysis shows that some microRNAs downregulate large numbers of target mRNAs[J].2005,433(7027):769-773.
[13] Hobert O.Common logic of transcription factor and microRNA action[J].Trends biochem Sci,2004,29(9):462-468.
[14] Liang R,Bates DJ,Wang E.Epigenetic control of microRNA expression and aging[J].Curr Genomics,2009,10(3):184-193.
[15] Doench JG,Sharp PA.Specificity of microRNA target selection in translational repression[J].Genes Dev,2004,18(5):504-511.
[16] Fabian MR,Sonenberg N,Filipowicz W.Regulation of mRNA translation and stability by microRNAs[J].Ann Rev Biochem,2010,79(6):351-379.
[17] Vasudevan S,Tong Y,Steitz JA.Switching from repression to activation:microRNAs can up-regulate translation[J].Science,2007,318(5858):1931-1934.
[18] Shalgi R,Lieber D,Oren M,et al.Global and local architecture of the mammalian microRNA-transcription factor regulatory network[J].PLoS Comput Biol,2007,3(7):1291-1304.
[19] Re A,Cora D,Taverna DM.Genome-wide survey of microRNA-transcription factor feed-forward regulatory circuits in human[J].Mol Biosys.2009,5(8):854-867.
[20] O'Donnell KA,Wentzel EA,Zeller KI,et al.c-Myc-regulated microRNAs modulate E2F1 expression[J].Nature,2005,435 (7043):839-843.
[21] Tang XB,Liu DP,Liang CC.Regulation of the transcription factor GATA-1 at the gene and protein level[J].Cell Mol Life Sci,2001,58(14):2008-2017.
[22] Kerenyi MA,Orkin SH.Networking erythropoiesis[J].J Exp Med,2010,207(12):2537-2541.
[23] Byon JC,Papayannopoulou1 T.MicroRNAs:Allies or foes in erythropoiesis?[J]J Cell Physiol,2011,227(1):7-13.
[24] 封 冰,陈龙邦.微小RNA与表观遗传调控:肿瘤治疗新策略[J].医学研究生学报,2011,24(1):92-95.
Bioinformatic analysis of the miR-144/451 cluster mediated regulation network
NIE Wei-wei1,TANG Lin1,WEI Feng1,CHEN Long-bang1,ZHANG Chen-yu2,ZHU Lin3,LI Dong-hai2,Guan Xiao-xiang1
(1.Department of Medical Oncology,School of Medicine,Nanjing University/Nanjing General Hospital of Nanjing Military Command,PLA,Nanjing210002,Jiangsu,China;2.Jiangsu Research Center for MicroRNA Engineering,State Key Laboratory of Pharmaceutical Biotechnology,School of Life Sciences,Nanjing University,Nanjing210093,Jiangsu,China;3.Institute of Discovery Biology,Jiangsu Simcere Pharmaceutical R&D Co.,Ltd,Nanjing210042,Jiangsu,China)
Objective Bioinformatic methods are useful tools for predicting the secondary structure and target gene of miRNA.The regulation network of miRNAs,transcription factors and target genes is involved in molecular mechanisms of physiological and pathological activities.This study is to predict the miR-144/451 cluster regulation network using online bioinformatic tools.Methods We studied some of the feedforward and feedback loops by integrating compressive information from different online databases.After function analysis of the genes involved in the circuits regulated by the miR-144/451 cluster,we determined the regulation network of the gene cluster. Results There were 17 joint targets of the miR-144/451 cluster and its upstream transcriptional factor GATA1,which played important roles in such biological processes as protein amino acid phosphorylation,gonad development,epithelial cell differentiation and glucose metabolism,as well as in some important signaling pathways,including insulin signaling pathway,mTOR signaling pathway,VEGF signaling pathway,natural killer cell mediated cytotoxicity,progesterone-mediated oocyte maturation,Toll-like receptor signaling pathway,and ErbB signaling pathway. Conclusion Bioinformatic analysis of the regulation network of the miR-144/451 cluster potentially provide a good guideline for deeper insights into its mechanisms of regulating cell development and tumorigenesis.
microRNA;miR-144/451 cluster;Bioinformatic analysis;Feedforward loop;microRNA feedback loop;Gene function
Q522
A
1008-8199(2012)03-0229-05
国家自然科学基金(81141094,31000323);江苏省自然科学基金(BK2011656);高等学校博士学科点专项科研基金(20100091120023,20100091120026),高校基本科研业务费专项资金(1095020823)
210002南京,南京大学医学院临床学院(南京军区南京总医院)肿瘤内科[聂伟伟(医学硕士)、唐林、韦 凤、陈龙邦、管晓翔];210093南京,南京大学生命科学院医药生物技术重点实验室,江苏省小核糖核酸工程研究中心(张辰宇、李冬海); 210042南京,南京市先声药业研究院生物评价研究所(朱 琳)
管晓翔,E-mail:xxguan@hotmail.com
2011-10-17;
2012-01-16)
(责任编辑:齐 名;英文编辑:罗永合)
