本氏针茅SRAP-PCR反应体系的建立及引物筛选
2012-12-24井赵斌程积民
井赵斌,俞 靓,魏 琳,程积民,,3
(1.西北农林科技大学动物科技学院,陕西 杨凌712100;2.西北农林科技大学资源与环境学院,陕西 杨凌712100;3.中国科学院水利部水土保持研究所,陕西 杨凌712100)
本氏针茅(Stipabungeana)为禾本科(Gramineae)针茅属多年生草本植物,又叫长芒草,俗称蓑草,广泛分布于我国西北、华北、西南和东北各地,亚洲中部及北部也有分布,是黄土高原典型草原的代表性植物和优势种植物。其根系发达,须根较多,草质柔软,营养丰富,适口性好,是一种牲畜较喜食的天然牧草,同时又是优良的水土保持植物[1]。以本氏针茅为优势种和建群种的典型半干旱气候带的云雾山草原自然保护区为我国黄土高原保留最完整、面积最大、原生性最强的典型区域。国内外对针茅属植物分子水平的研究较多,但国内对本氏针茅光合生理生态学方面的研究报道较少[1-6],尚未见基于分子生物学基础的研究报道。
基于DNA分子水平上的植物遗传多样性和遗传分化在物种多样性保护中的作用已得到公认。目前,对林草的遗传多样性研究由以濒危稀有物种为主逐渐向以优势种和建群种并重发展,优势种和建群种遗传多样性的研究不但对物种的保护和利用具有重要意义,而且可以为生态系统遗传健康的评估提供基础。国内外对针茅属植物遗传多样性的研究已有较多报道[7-12],已有的研究基本上以内蒙古草原为主体,对全国其他生态分布区的研究基本上属于空白,研究所用的标记以 RAPD[9-11]和ISSR[13]标记为主。SRAP标记(Sequence-Related Amplified Pol y mor phis m,相关序列扩增多态性)是由Li和Quiros[14]于2001年开发的一种基于PCR的新型随机引物标记系统。目前,SRAP标记已被成功用于遗传多样性分析、遗传图谱构建、QTL定位分析、品种鉴定和杂种优势等的研究中,结果表明较RAPD、SSR、ISSR、AFLP等标记具有更丰富的多态性且操作简便、重复性高、成本较低,特别是对序列信息未知的物种省去了开发引物的过程[15-17]。
本研究以黄土高原区本氏针茅为材料,在建立适于本氏针茅DNA提取方法的基础上,采用正交试验设计和单因素分析相结合的方法对SRAP反应体系进行优化,在最优反应体系建立的基础上对部分SRAP引物进行了多态性筛选,以期为利用SRAP标记进一步开展本氏针茅种质资源遗传多样性研究和黄土高原植被恢复及生态建设提供理论基础。
1 材料与方法
1.1 材料 供试材料于2010年7-9月采自陕西、宁夏、内蒙古和甘肃不同生态分布区(表1)。采样时同一生态区不同个体之间的距离保持在10 m以上,每个样品选取幼嫩无病斑叶片,采集编号后装塑料袋密封,置冰盒中带回实验室,存于-80℃冰箱中备用。优化体系材料为采集于陕西铜川和宁夏固原云雾山的本氏针茅资源,编号分别为SX-1-TC-1和NX-1-Y WS-1,单因素筛选试验材料为陕西铜川本氏针茅,编号为SX-1-TC-1,体系验证材料为甘肃、宁夏和内蒙古选出的8个采样点中的8个单株,编 号 分 别 为 GS-1-HX-1、NX-2-XJ-1、NX-3-H MS-1、NX-4-HC-1、NX-5-Y WS-1,NX-6-LPS-1、NMG-1-ETC-1和NMG-2-ETC-1,引物筛选从4个省区中各选取1个采样点(不同生态区)中的1个单株,编号分别为 SX-2-JB-1、NMG-3-ETC-1、NX-7-YC-1和GS-2-JY-1。

表1 供试材料Table 1 List of experi mental materials
1.2 方法
1.2.1 DNA提取及检测 取采集的本氏针茅幼嫩叶片,参考文献[18]的方法略作修改后提取基因组DNA。分别用0.8%的琼脂糖凝胶电泳和紫外分光光度计检测DNA质量和浓度后,稀释工作液至20 ng·μL-1,保存母液和工作液于-20℃冰箱中备用。
1.2.2 SRAP-PCR反应体系的优化 SRAP引物参考 Guo和Luo[19]、Ferriol等[20]和 Budak等[21]发表的引物序列,共17条正向引物和21条反向引物(表2),引物由上海捷瑞生物工程公司合成。用于体系优化的引物组合为Me1+Em1(材料:SX-1-1)和 Me2+Em6(材料:NX-1-Y WS),体系验证的引物组合为Me3+Em1(材料:GS-1-HX-1、NX-2-XJ-1)、Me5+Em2(材 料:NX-3-H MS-1、NX-4-HC-1)、Me6+Em5(材料:NX-5-Y WS-1、NX-6-LPS-1)、Me12+Em9(材料:NMG-1-ETC-1、NMG-2-ETC-1)。

表2 引物序列Table 2 Sequence of pri mers
PCR反应程序采用复性变温法:94℃预变性5 min;前5个循环为94℃变性1 min,35℃退火1 min,72℃延伸1 min;后35个循环仅将复性温度变为50℃;循环结束后,72℃延伸10 min,4℃保存。PCR扩增在Eppendorf PCR仪上进行。
采用L16(45)正交试验设计,对影响SRAPPCR反应的主要5个因子:DNA、Taq酶、d NTPs、Mg2+和引物进行5因素4水平的优化筛选试验,重复2次(2对引物组合2个材料),各反应组分的因素水平正交试验见表3。反应体系总体积为20μL,其中10×Buffer为2.5μL,其他各组分按照表2加入,不足体积用dd H2O补足。以正交试验结果选出的最优体系为基础,对5个主要因子设置8个不同的浓度梯度处理(表4),在其他因子浓度保持不变的情况下按照8个梯度变化单个因子,逐个筛选出各个因子的最佳反应浓度。扩增产物用6%的变性聚丙烯酰胺凝胶电泳检测,银染显影,照相统计结果。
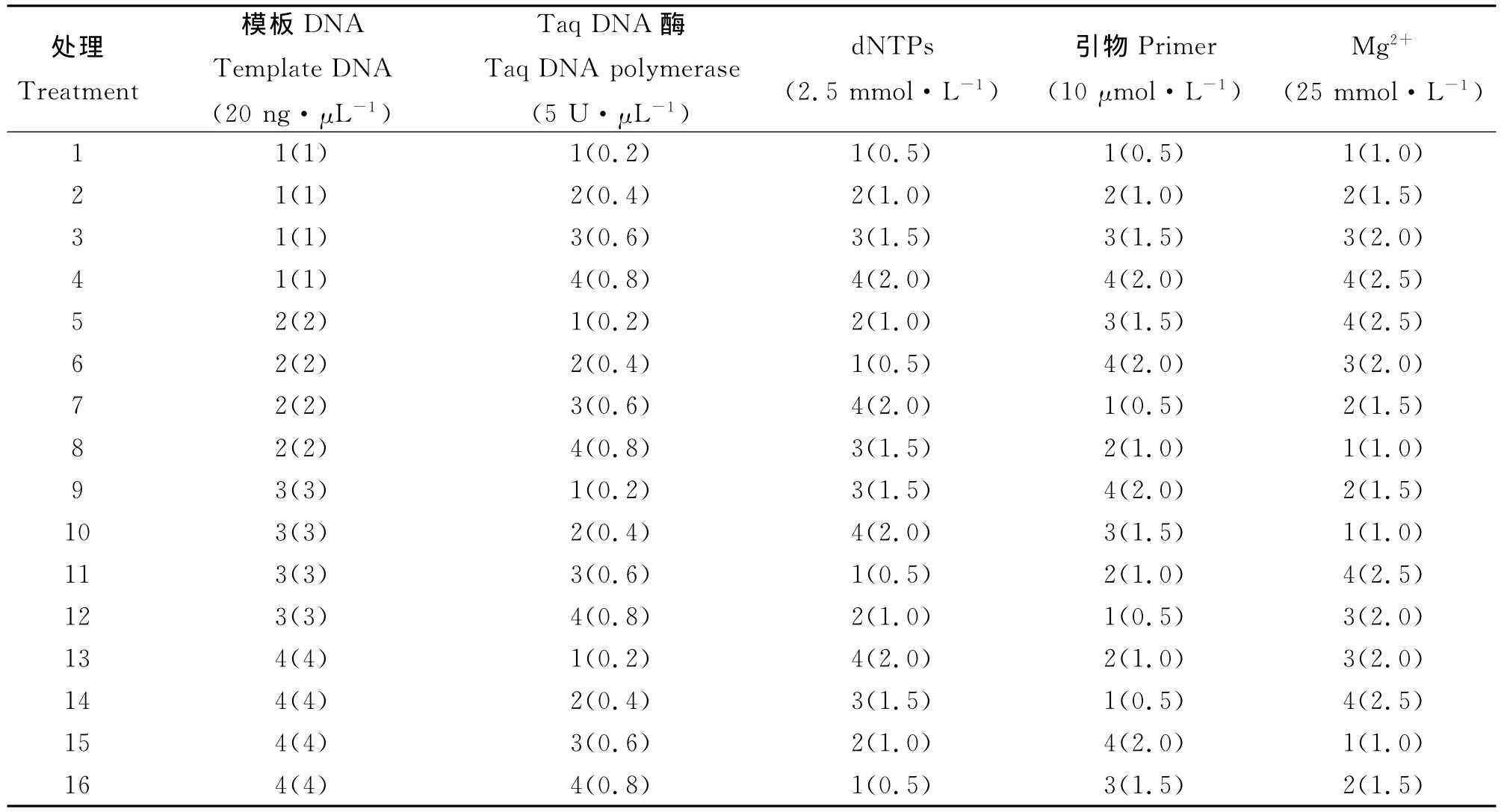
表3 SRAP-PCR反应中各试验组分因素水平L 16(4 5)正交试验设计Table 3 Factor and level of SRAP-PCR by orthogonal design[L 16(4 5)]

表4 SRAP-PCR反应体系优化单因素筛选试验Table 4 Optimal concentration gradient of each factor in SRAP-PCR system mL
1.2.3 数据分析 SRAP-PCR正交试验设计和单因素筛选试验最优反应体系扩增结果的选择采用直观法和统计分析法进行分析。直观法主要根据扩增条带数量、清晰度以及背景对16个处理进行直观分析。统计分析法采用郑轶琦等[22]方法分别计算出某水平下某因子参与反应所产生的扩增条带总数(Ki),某因子在某水平参与反应所产生的扩增条带的平均值(Xi)以及某因子在不同水平下最大平均值与最小值平均值之差(极差R),最后确定选出各个组分的最优水平。
1.2.4 SRAP-PCR优化体系的验证和确立 根据正交试验设计和单因素分析优化建立的最优反应体系,选取2个省份中8个采样点中的共8份材料和4对随机引物组合进行验证,进一步确立适合于本氏针茅种质资源遗传多样性SRAP分析的最优反应体系。
2 结果
2.1 基因组DNA提取 基因组DNA提取参考井赵斌[18]的SDS法提取本氏针茅DNA,经琼脂糖电泳检测后在点样孔处有亮带聚集产生,表明有部分多糖物质未去除干净,且有拖尾现象,说明DNA严重降解,如图1中样品6、7、8和9;用紫外分光光度计检测DNA浓度和质量,发现用SDS法提取的DNA其260 n m/280 n m处OD值均低于1.8,说明DNA中有蛋白质污染,这些结果均表明用SDS法提取本氏针茅基因组DNA效果较差。随后,在此方法的基础上进行改进,将SDS提取液改为CTAB提取液[100 mmol·L-1Tris-HCL(p H 值8.0),1.4 mol·L-1Na Cl,50 mmol·L-1EDTA(p H 值8.0),2%CTAB及20μLβ-巯基乙醇],再加入140 μL 20%的PVP充分混匀,同时增加了2个步骤(65℃水浴20 min和加无水乙醇后冰浴20 min)。用改良后的方法提取的DNA经电泳检测后,条带整齐,没有拖尾和降解现象出现,说明质量很好,如图1中样品1、2、3、4和5;紫外分光光度计检测结果表明,其OD值介于1.8~2.0,且浓度较高。本研究建立了适用于本氏针茅基因组DNA提取的改良CTAB法,用该方法提取的DNA经SRAP-PCR检测后,完全可以满足试验要求。

图1 基因组DNA琼脂糖电泳检测结果Fig.1 Agarose gel electrophoresis of genomic DNA
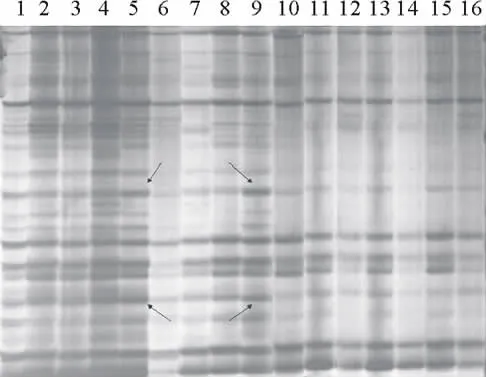
图2 SRAP-PCR正交试验设计扩增结果电泳图Fig.2 Amplification of SRAP-PCR by orthogonal design[L 16(4 5)]
2.2 SRAP-PCR正交试验设计优化体系结果分析 正交试验设计1 6个组合的SRAP-PCR扩增结果如图2所示。因正交试验设计具有均衡分散的特点,SRAP-PCR反应体系中各个试验组分的浓度组合彼此不同,因此扩增结果具有明显的差异(图2),组合6、10、11、12、14和16扩增条带数较低,且条带比较微弱模糊;组合1、2、3、4和7扩增结果较好,但与组合5、8、9和13相比,条带数较少且条带之间界限较为模糊;组合5、8、9和13之间,扩增条带数基本一致,组合8和13与组合5和9相比,如图2中箭头所示位置条带很微弱且模糊;最后组合5和9进入候选。比较分析2个组合中各个组分的浓度差异,组合5中的模板DNA、d NTPs和引物用量都较组合9少,最后确定第5组合为SRAP-PCR正交试验设计中的最优组合,即在20μL总的反应体系中,包括模板 DNA(20 ng·μL-1)2μL、Taq DNA 酶 (5 U·μL-1)0.2μL、d NTPs(2.5 mmol·L-1)1μL、引物(10μmol·L-1)1.5μL、Mg2+(25 mmol·L-1)2.5μL、10×Buffer 2.5μL、dd H2O 8.8μL。
参考郑轶琦等[22]方法,对正交试验设计中各个组合进行统计分析(表5),极差值R的大小反映了某组分对试验结果的影响,R值越大,影响越大。根据R值的大小,在设定的4个水平上,5个组分对扩增结果的影响由大到小依次为Taq DNA酶>模板DNA>引物>d NTPs>Mg2+。X值反映了各组合中各水平对反应体系的影响大小,X值越大,反应水平越佳。从表5中X值结果来看,模板DNA以水平1为佳,Taq DNA酶以水平1为佳,d NTPs、引物和Mg2+均以水平2为佳,即模板 DNA(20 ng·μL-1)2μL、Taq DNA 酶(5 U·μL-1)0.2 μL、d NTPs(2.5 mmol·L-1)1 μL、引 物 (10 μmol·L-1)1.5μL、Mg2+(25 mmol·L-1)2.5 μL,该结果与直观分析选出的最优组合5最为接近,最终确定组合5为正交试验设计中的最优组合。为进一步获得理想的反应体系,在组合5的基础上进行单因素筛选试验,以微调各组分的用量建立更优的SRAP-PCR反应体系。
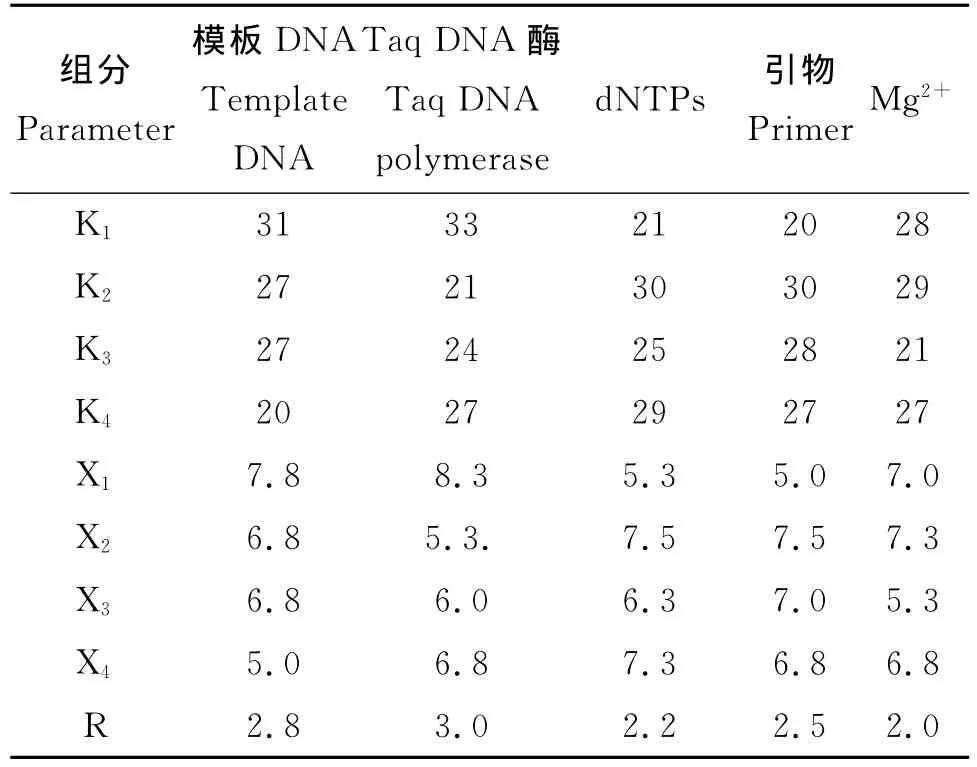
表5 SRAP-PCR正交试验设计各处理统计分析Table 5 Result of SRAP-PCR by orthogonal design
2.3 SRAP-PCR单因素筛选
2.3.1 模板DNA最优加入量的确定 合适的模板DNA加入量和质量是影响PCR扩增结果的关键因素。在设置的8个DNA加入量中均有条带产生,其中DNA加入量分别为1.5、2.0、2.5和3.5μL时,条带数量和清晰度表现较差;在加入量分别为0.5、1.0、3.0和4.0μL时,均可扩增出清晰的条带,但在3.0μL时条带清晰度和背景最好(图3),结合正交试验结果综合分析,确定20μL总反应体积中模板DNA的用量以3.0μL为最佳。

图3 模板DNA浓度对SRAP-PCR扩增结果的影响Fig.3 Effects of template DNA concentration on SRAP-PCR amplification
2.3.2 Mg2+最优浓度的确定 Mg2+的浓度直接影响着引物退火和产物的特异性以及酶的催化能力和准确性。在加入量为0.4和0.8μL时,扩增条带模糊,加入量在1.2~2.4μL时,背景清晰条带丰富,当加入量大于2.4μL时条带开始变微弱(图4)。直观分析总体上在加入量为2.0μL时扩增效果最优。因此,确定20 μL总反应体系中Mg2+最优加入量为2.0μL。
2.3.3 Taq DNA聚合酶最佳用量的确定 Taq DNA聚合酶的用量直接影响扩增反应的成败。8个Taq DNA聚合酶梯度都有扩增条带,用量在0.1和0.3μL时,条带较弱且缺失部分条带;用量在0.5~0.8μL时,均可得到理想的扩增条带;用量为0.2μL时,扩增产物全部出现(图5),但是条带稍有点微弱,结合正交试验结果最终以0.2μL作为体系入选最佳用量。
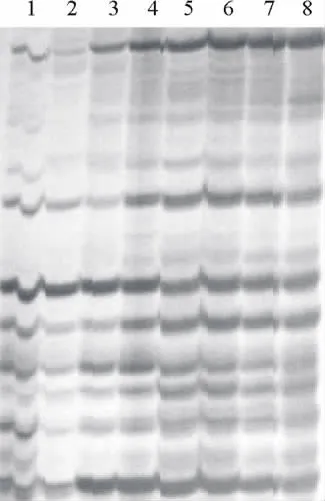
图4 Mg2+浓度对SRAP-PCR扩增结果的影响Fig.4 Effects Mg2+concentration on SRAP-PCR amplification

图5 Taq DNA聚合酶浓度对SRAP-PCR扩增结果的影响Fig.5 Effects of Taq DNA poly merase concentration on SRAP-PCR amplification
2.3.4 引物最优加入量的确定 引物加入量过高会引起非特异性扩增且易形成引物二聚体,而过低的引物加入量降低PCR扩增效率。从图6可以看出,除加入量为0.2μL时几乎没有扩增产物外,其他均有较清晰条带。结合正交试验候选组合引物加入量,最后确定1.5μL的引物为20μL反应体系中的用量。
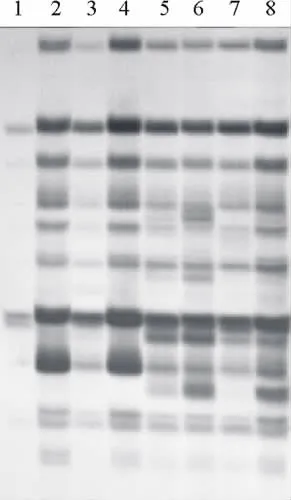
图6 引物加入量对SRAP-PCR扩增结果的影响Fig.6 Effects of pri mer concentration on SRAP-PCR amplification
2.3.5 d NTPs最优加入量的确定 高加入量d NTPs可加速反应,但同时增加错误掺入和试验成本;低加入量可提高精确性,而反应速度会降低。从d NTPs扩增结果(图7)来看,除加入量在1.6μL时条带微弱外,其他各梯度加入量均有扩增条带且清晰度和数量基本一致,但比较发现加入量为1.4μL时条带表现更清晰,结合正交试验结果最终选择1.4 μL为20μL反应体系中的最佳加入量。
2.4 最优反应体系的验证及多态性引物筛选根据SRAP-PCR正交试验设计和单因素筛选试验结果,得到本氏针茅SRAP标记的最优反应体系为20μL总的反应体系中包括:DNA(20 ng·μL-1)3μL、Taq DNA 酶(5 U·μL-1)0.2 μL、d NTPs(2.5 mmol·L-1)1.4μL、引物(10 μmol·L-1)1.5μL、Mg2+(25 mmol·L-1)2.0μL、10×Buffer 2.5μL、dd H2O 7.9μL。选取下列引物和材料进行最优体系验证:Me3+Em1、Me5+Em2、Me6+Em5、Me12+Em9。选取的4对引物组合在8份材料间均能扩增出很好的条带,且多态性特别丰富(图8)。选取4个省份差异较大生态区的材料4份对表2中组合的共357对引物进行SRAP-PCR扩增,共筛选出条带整齐、背景清晰的多态性引物共85对,最后从85对多态性引物中选出更优的20对进行本氏针茅遗传多样性分析。
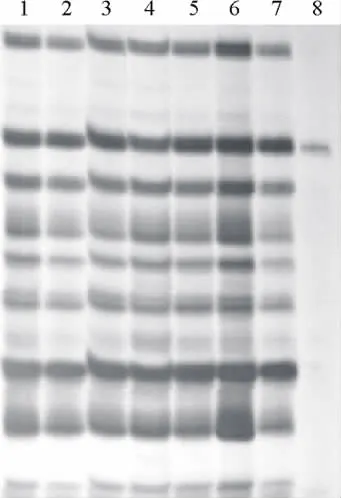
图7 d NTPs加入量对SRAP-PCR扩增结果的影响Fig.7 Effects of d NTPs concentration on SRAP-PCR amplification

图8 SRPA-PCR最优反应体系扩增结果验证Fig.8 Validation of amplification result by optimal SRPA-PCA system
3 讨论
3.1 本氏针茅基因组DNA提取方法 高质量的DNA是影响PCR扩增成败的关键因素,目前植物基因组DNA提取的方法已很成熟,但是不同植物体内含有的蛋白质、单宁、色素、酚类及多糖等物质成分差异较大,一般直接参考已有的方法不易成功。本氏针茅属于多年生草本植物,不同生态和地理分布区的植株持水量差异很大,本研究在提取本氏针茅基因组DNA过程中发现采集于荒漠草原类型区的样品,其叶片中含有的纤维物质特别多,不易研磨而直接影响了提取的DNA质量。基因组DNA提取开始时,直接参考用于水稻(Or yzasativa)基因组DNA提取的SDS法,电泳结果表明DNA全部降解,原因可能和研磨过程中未加液氮和叶片持水量较低有关。经过几次改进后,得到了适于本氏针茅基因组DNA提取的改良CTAB法,该方法具有提取DNA质量好、浓度高、时间短和易于操作等优点。SRAP-PCR扩增和聚丙烯酰胺凝胶电泳结果表明,用改良的CTAB法提取的基因组DNA完全可以满足本氏针茅遗传多样性分析的要求。
3.2 正交试验设计和单因素分析法及其在PCR反应体系优化中的应用 PCR反应体系的合适与否直接影响PCR扩增多态性位点的数量及遗传多样性结果的精确性,因此建立一个适宜的PCR反应体系是进行遗传多样性研究的首要条件。目前,应用于PCR体系优化的方法较多,其中以正交试验设计和单因素法应用最多,且在许多牧草PCR反应体系优化试验中得到了应用[19-20]。正交试验设计是统计数学的重要分支,运用这种方法可以达到减少试验次数,缩短试验周期,降低试验和生产成本,迅速找到优化方案,实现最大效益的目的;但该方法也有一定的局限性,如对试验结果本身优劣的判断具有主观性,不能很好地估计试验误差,也不能表明各试验因素水平的交互作用[22]。单因素分析法可直观快速地得到该因素对扩增程序的影响,但是忽略了各因素间的相互作用[23]。正交试验设计和单因素分析法相结合可以弥补彼此的缺点。前人[13,23]已发表的PCR体系优化报道基本上以单一的正交试验设计或者单因素分析法为主,本研究在正交试验设计的基础上,进一步对主要试验因素设置的合理浓度梯度进行微调,得到了更优的SRAP-PCR反应体系,其增效果表现更好。目前,基本上以直观法和统计分析法为标准对PCR体系优化试验结果进行评价分析,这2种方法的共同缺点是对结果的分析带有个人主观性,在扩增条带结果统计过程中难免会有人为设置标准的误差,所以更加科学和完善的评价分析方法亟需研究建立。
3.3 其他因素对PCR反应体系的影响 模板DNA、Taq DNA聚合酶、d NTPs、Mg2+和引物是影响PCR反应体系的主要因素,各个因素的主要作用及其之间的互作已有较多报道[19-20]。本研究在反应体系建立后,将Taq DNA聚合酶、d NTPs、Mg2+和10×Buffer进行了更换(不同生产商),结果发现相同的体系在不同的产品之间可以进行扩增,但是结果因产品质量不同会产生差异,如扩增条带数量和清晰度等;同时对其他组分和条件完全相同而采用不同公司合成的相同引物序列进行比较研究后发现,结果差异较大,所以引物合成质量的优劣是决定多样性丰富的一个关键因素。体系验证是对最优反应体系适宜性的进一步分析,因此应把选择不同生态和地理分布区的材料作为一个关键因素考虑,这样对最优反应体系在差异较大材料间的广适性可以得到更加精确的结论。
4 结论
本研究建立了适合本氏针茅基因组DNA提取的改良CTAB法,并在此基础上建立了本氏针茅SRAP-PCR反应体系,即在20μL总的反应体系中包括:DNA(20 ng·μL-1)3μL、Taq DNA 酶(5 U·μL-1)0.2μL、d NTPs(2.5 mmol·L-1)1.4 μL、引 物 (10 μmol·L-1)1.5 μL、Mg2+(25 mmol·L-1)2.0μL、10×Buff er 2.5μL、dd H2O 7.9μL,该体系经PCR扩增检测能很好地用于遗传多样性研究中。
[1]周秋平,程积民,万惠娥,等.干旱胁迫下本氏针茅光合特性水分利用效率日动态研究[J].草地学报,2009,17(4):510-514.
[2]王静,程积民,万惠娥.黄土高原本氏针茅光合特性及生产力的研究[J].中国生态农业学报,2005,13(4):71-73.
[3]胡相明,程积民.施水对本氏针茅光合作用和叶绿素荧光特征的影响[J].中国水土保持科学,2006,4(2):39-46.
[4]韩凤朋,董丽娜,罗文林,等.黄土高原侵蚀区长芒草对坡地土壤水分养分的影响[J].草地学报,2008,16(4):403-407.
[5]周梦华,程积民,万惠娥,等.云雾山本氏针茅群落根系分布特征[J].草地学报,2008,16(3):267-271.
[6]周秋平,程积民,万惠娥.本氏针茅与柳枝樱光合生理生态特征比较研究[J].水土保持通报,2009,29(2):129-133.
[7]Wu J B,Gao Y B,Bao X Y,etal.Genetic diversity ofStipa gr andisP.Smir n populations acr oss the species’range in t he Inner Mongolia Plateau of China[J].Biochemical Systematics and Ecology,2010,38(4):471-477.
[8]Liu W S,Dong M,Song Z P,etal.Genetic diversity pattern ofStipa pur pureapopulations in the hinterland of Qinghai-Tibet Plateau[J].Annals of Applied Biology,2009,154(1):57-65.
[9]Zhao N X,Gao Y B,Wang J L,etal.RAPD diversity ofStipa gr andispopulations and its relationship with some ecological factors[J].Acta Ecologica Sinica,2006,26(5):1312-1319.
[10]张红梅,赵萌莉,李青丰,等.内蒙古地区大针茅群体遗传多样性 RAPD研究[J].草地学报,2003,11(2):170-188.
[11]赵念席,高玉葆,王金龙,等.内蒙古中东部草原区克氏针茅种群遗传分化的RAPD研究[J].生态学报,2004,24(3):560-566.
[12]张庆,牛建明,董建军.内蒙古地区短花针茅(Stipa brevif lor a)种群遗传多样性[J].生态学报,2008,28(7):3447-3455.
[13]沈紫微,陈本建,康俊梅,等.红豆草ISSR体系优化及其在航天诱变种质鉴定中的应用[J].草业科学,2010,27(12):65-72.
[14]Li G,Quiros C F.Sequence-related amplified poly morphis m (SRAP),a new mar ker system base on a si mple PCR reaction:its application to mapping and gene tagging inBr assica[J].Theoretical and Applied Ge-netics,2001,103:455-461.
[15]刘丽娟,刘灶长,陈海荣,等.SRAP标记技术及其在蔬菜作物遗传多样性分析中的应用[J].中国农学通报,2009,25(21):43-48.
[16]李润芳,惠荣奎,邓瑞宁,等.三叶草遗传多样性的SRAP分析[J].草业科学,2010,27(12):53-57.
[17]鄢家俊,白史且,张新全,等.青藏高原老芒麦种质基于SRAP标记的遗传多样性研究[J].草业学报,2010,19(1):173-183.
[18]井赵斌.广东高州普通野生稻渗入系构建及产量性状的QTL定位[D].乌鲁木齐:新疆农业大学,2009:32-34.
[19]Guo D L,Luo Z R.Genetic relationships of some PCNA persimmons(DiospyroskakiThunb.)from China and Japan revealed by SRAP analysis[J].Genetic Resources and Crop Evolution,2006,53:1597-1603.
[20]Ferriol M,Pico B,Nuez F.Genetic diversity of a germplasm collection ofCucurbitapepousing SRAP and AFLP markers[J].Theoretical and Applied Genetics,2003,107:271-282.
[21]Budak H,Shearman R C,Parmaksiz I,etal.Comparative analysis of seeded and vegetative biotype buffalograsses based on phylo-genetic relationship using ISSRs,SSRs,RAPDs,and SRAPs[J].Theoretical and Applied Genetics,2004,109:280-288.
[22]郑轶琦,王志勇,郭海林,等.正交设计优化假俭草SRAP-PCR反应体系及引物筛选[J].草业学报,2008,17(4):110-117.
[23]王方,袁庆华.冰草ISSR-PCR反应体系的建立与优化[J].草地学报,2009,17(3):354-357.
