宁南山区本氏针茅自然种群遗传分化的ISSR分析
2012-12-24井赵斌程积民
俞 靓,井赵斌,魏 琳,程积民
(1.西北农林科技大学资源与环境学院,陕西 杨凌712100;2.西北农林科技大学动物科技学院,陕西 杨凌712100;3.中国科学院水利部水土保持研究,陕西 杨凌712100)
典型草原是温带内陆半干旱气候条件下形成的草地类型,针茅属植物是构成其草原植被的重要组分[1]。在严重的人为干扰和恶劣的气候影响下,作为建群种或优势种的针茅属植物大范围衰退[2],因此从物种保护角度对其遗传多样性和遗传结构进行研究具有重要意义。目前,利用DNA分子标记对针茅属植物的遗传多样性研究已有一些报道。赵念席等[3-6]、张庆等[7]和宋涛等[8]采用 RAPD 标记分别分析了内蒙古地区大针茅(Stipagrandis)和克氏针茅(S.krylovii)的遗传分化,短花针茅(S.breviflora)的遗传多样性以及内蒙古中东部地区贝加尔针茅(S.baicalensis)的遗传关系。但应用其他DNA分子标记对我国针茅属植物遗传多样性的研究尚未见报道。
本氏针茅(S.bungeana)又名长芒草,是一种喜暖旱生丛生禾草,属于禾本科针茅属无毛芒组(Sect.Leiostipa Dum),是黄土高原典型草原的建群种和优势种植物,同时也是治理水土流失、改善生态环境的优良草种,因其草质柔软、营养丰富、适口性较好,又是牲畜较喜食的天然饲草[9]。近年来,黄土高原地区水土流失日趋严重,生态环境恶化,使得大面积的本氏针茅种群已不多见,因此,迫切需要对本氏针茅天然草原进行保护。
分子标记被认为是研究遗传多样性的最有效途径,在各种DNA分子标记技术中,ISSR(Inter Simple Sequence Repeat)分子标记技术,因其不具有物种特异性的特点[10],目前被广泛用于遗传信息缺乏的牧草资源的遗传多样性研究中[11-15]。本研究对宁南山区6个本氏针茅种群的遗传多样性进行ISSR分析,旨在探讨本氏针茅种群遗传多样性及遗传变异产生的分子生态机理,以期为本氏针茅草原的合理利用和科学管理提供参考依据。
1 材料与方法
1.1 材料 2010年7月中旬在宁夏回族自治区宁南山区,选取6个本氏针茅自然种群(均为典型草原,其他生境状况详见表1。在各种群内,随机挑选20株植物,单株间距离不小于10m,每株剪取幼嫩、健康、无病斑叶片若干,编号装袋,置冰盒中带回实验室,于-80℃条件下保存备用。
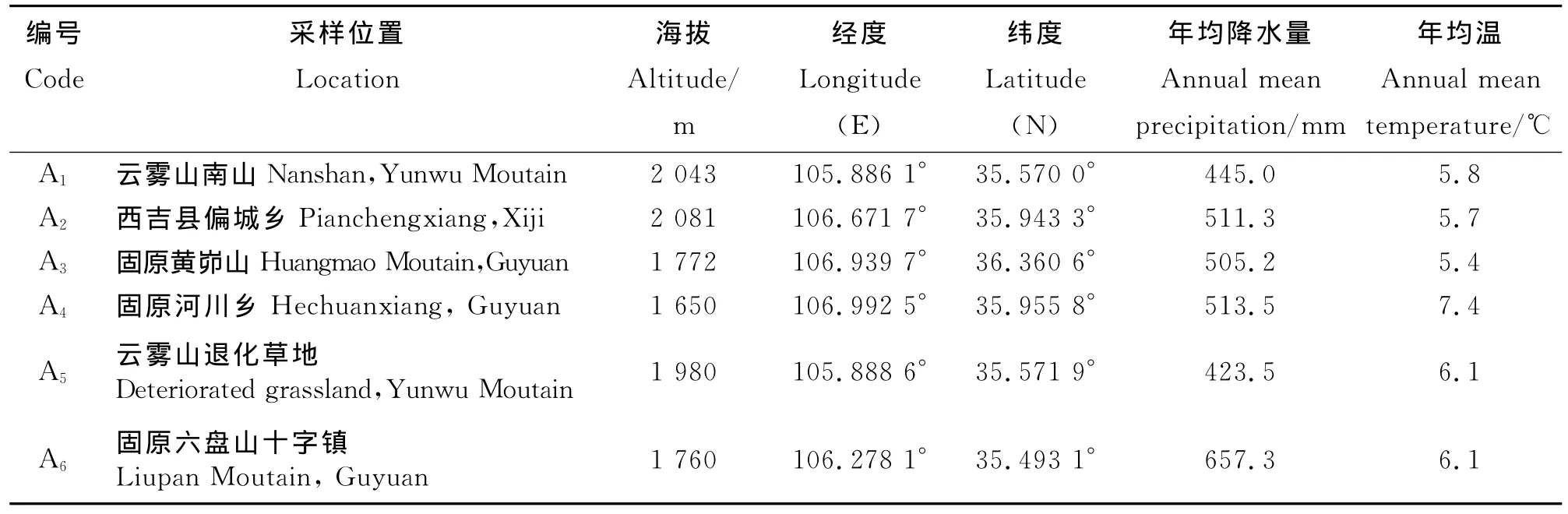
表1 采样点的生境特征Table 1 Collected locations and habitat characteristics
1.2 方法
1.2.1 DNA提取及检测 采用改良的CTAB法提取基因组DNA,经0.8%的琼脂糖凝胶电泳和紫外分光光度计双重检测。将各样品的基因组DNA工作液浓度用TE缓冲液稀释至20ng·μL-1作为PCR模板,保存母液和工作液于-20℃冰箱中备用。
1.2.2 引物筛选 引物选用加拿大哥伦比亚大学(University of British Columbia Biotechnology,UBC)公布的96个ISSR引物,由北京奥科生物工程公司合成。选取4个田间性状表现差异较大的种群,每个种群内随机选取一个单株,共计4个单株进行引物筛选,最终选取15个扩增产物条带清晰、多态性高且重复性好的引物用于种群遗传多样性的分析,引物序列见表2[16]。

表2 本氏针茅ISSR分析的引物序列Table 2 Primer sequences used in ISSR analysis of Stipa bungeana
1.2.3 PCR扩增及产物的检测 PCR扩增反应体系为:DNA 20ng、Taq DNA酶1U、dNTPs 0.18 mmol·L-1、引物0.75mmol·L-1、Mg2+1.87、10×Buffer(100mmol·L-1Tris-HCl,pH 值8.3;500mmol·L-1KCl),不足体积的用ddH2O补充至20μL[17]。PCR扩增反应程序:第1阶段,94℃预变性5min;第2阶段共35个循环,首先94℃变性45s,然后退火45s(退火温度因不同引物而定,表2),再者72℃延伸90s;第3阶段,72℃延伸5 min;最后4℃保存[18]。
扩增产物经6%的变性聚丙烯酰胺凝胶电泳分离后,用0.1%的AgNO3进行银染,并在NaOH溶液中显色,凝胶用高分辨率数码相机拍照保存。
1.2.4 数据处理与分析 用人工读带的方法,选择清晰可辨的扩增条带进行统计,模糊不清的条带忽略不计。按同一迁移位置上扩增产物的有无进行统计,扩增产物在相同迁移位置有带赋值为“1”,无带赋值为“0”,建立原始二元性状数据矩阵,通过软件DCFA 1.1[19]形成数据分析的原始文件,再依据不同的研究目的选取不同的软件对数据进行分析[20]。
在假设种群内基因频率处于Hardy-Weinberg遗传平衡的前提下,利用POPGENE 1.32[21]软件计算多态位点比率(PPB)、Nei’s基因多样性(H)和Shannon信息指数(I)等遗传多样性指数,用于估算各种群间的遗传多样性,种群内和种群间的遗传分化系数(φst)用软件 AMOVA 1.55[21]计算。
利用POPGENE 1.32软件,根据各条带的出现频率计算各种群间的遗传距离(D)和遗传一致度(I),并用非加权配对算术平均法(UPGMA)对各种群间遗传距离进行聚类分析,经POPGENE软件1 000次重抽样构建聚类图。
利用Pearson相关分析(SPSS 16.0)的方法计算各种群遗传多样性指数与生态因子间(海拔、经度、纬度、年均温和年降水量)的相关系数。
利用R语言对各种群间的遗传距离矩阵(POPGENE软件计算所得)与实际地理距离矩阵进行Mantel检验,以检验遗传距离与实际地理距离之间的相关性[22]。
2 结果
2.1 本氏针茅的遗传多样性 15条ISSR引物在6个本氏针茅种群的120个单株中共扩增出244条清晰的条带,扩增的DNA片段大部分集中在100~2 000bp(图1),其中多态性条带239条,多态性位点比率(PPB)为97.95%,Nei’s基因多样性(H)为0.254 4,Shannon信息指数(I)为0.396 6。在6个种群中,A2种群的遗传多样性最高(PPB=68.44%,H=0.213 2,I=0.324 4),而 A6种群的遗传多样性最低(PPB=41.39%,H=0.111 7,I=0.173 4)(表3)。
2.2 本氏针茅的群体遗传结构 本氏针茅种群的遗传变异通过POPGENE软件分析得出,其种群总遗传多样性Ht=0.254 4,种群内遗传多样性Hs=0.157 9,种群间遗传多样性 Dst=0.096 5(Dst=Ht-Hs),基因分化系数 Gst=0.399 6。结果表明,种群间的遗传变异较低于种群内的遗传变异,分别占总遗传变异的39.96%和60.04%。
分子变异方差分析(AMOVA)结果表明(表4),种群间的变异方差分量为16.142 6,种群内不同个体间的方差分量为22.346 1,遗传分化系数φst=0.419,表明本氏针茅种群的变异是由不同种群(遗传变异为41.90%)和种群内个体(遗传变异为58.06%)共同提供,即种群间的变异和种群内不同个体的变异共同构成了群体的遗传差异,贡献都达到了极显著(P<0.001)。上述结果与POPGENE的分析结果基本一致,均表明本氏针茅的遗传变异主要存在于种群内,种群间的遗传变异较小。
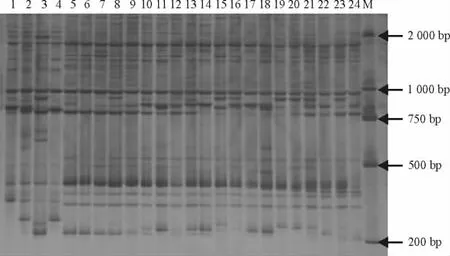
图1 引物UBC834对部分本氏针茅材料的ISSR扩增图谱Fig.1 ISSR fingerprints of some Stipa bungeana amplified with Primer UBC834
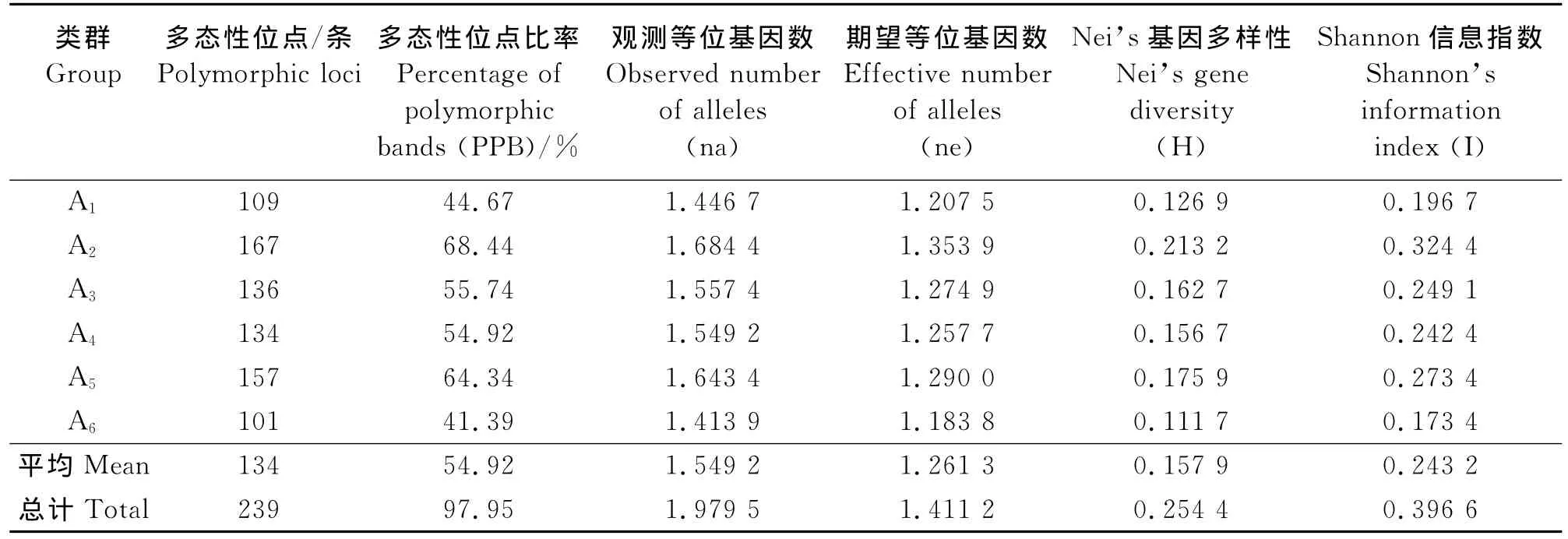
表3 6个本氏针茅种群的遗传多样性指数Table 3 Genetic diversity indexes of six populations of Stipa bungeana
2.3 种群间遗传关系及聚类分析 基于Nei氏遗传一致度和遗传距离可进一步分析种群间的遗传分化程度[23]。本氏针茅6个种群中(表5),A3与A4间遗传一致度最高,为0.977 7,相应的遗传距离最小,为0.022 5;而A1与A6间遗传一致度最低,为0.706 1,遗传距离最远,为0.347 9,说明A3与A4种群间的关系最近,A1与A6种群间的关系最远。
利用UPGMA法建立的系统树可以直接反映植物种内、种间或居群间的关系[24],因此对本氏针茅种群的Nei’s遗传距离构建遗传关系聚类图(图2)。此聚类图表明,6个自然种群聚成4支:A3和A4种群聚成一支;A5和A6种群聚成一支;A1和A2种群各聚成一支。
根据6个种群间的遗传距离矩阵值和地理矩阵值的Mantel检测结果(图2),在1 000次置换中,各种群间的遗传距离大小和地理距离远近均无显著相关性(r=-0.156 9,P=0.733>0.05),说明引起本氏针茅种群分化的主要原因不是地理距离的远近。
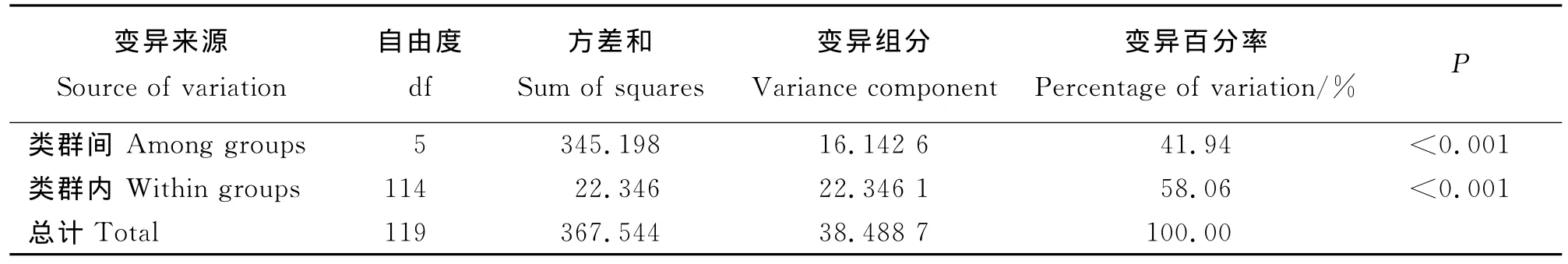
表4 本氏针茅种群的分子变异方差分析(AMOVA)Table 4 Analysis of molecular variance(AMOVA)of Stipa bungeana populations
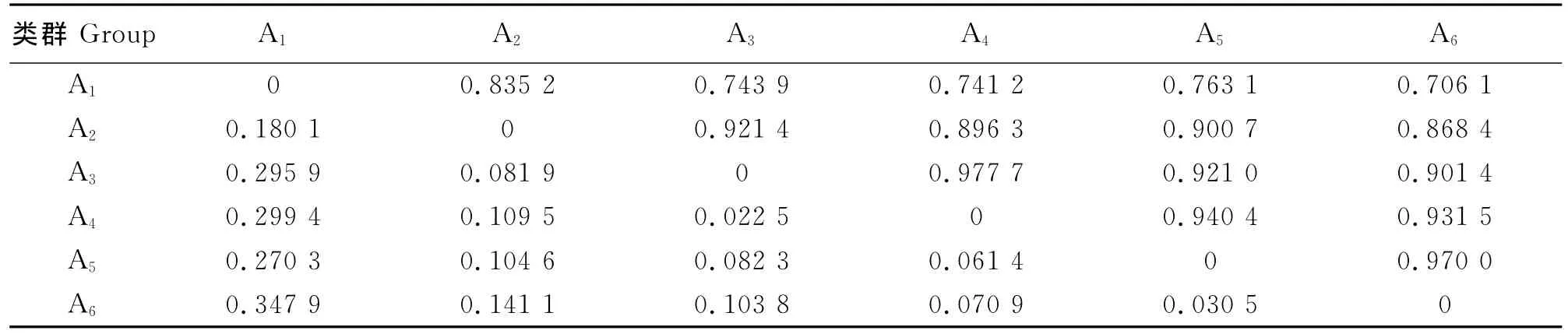
表5 本氏针茅种群遗传一致度和遗传距离Table 5 Genetic similarity coefficient and genetic distance of Stipa bungeana

图2 基于本氏针茅种群间Nei’s无偏差遗传距离构建的UPGMA聚类Fig.2 Dendrogram generated by UPGMA based on Nei’s unbiased genetic distances among Stipa bungeana populations
2.4 遗传多样性指数与生态因子之间相关性分析 对本氏针茅种群ISSR标记的遗传多样性指数与其所在生境的生态因子进行Pearson相关分析,结果表明,ISSR标记的遗传多样性指数与其所处生境的生态因子均未达到显著相关水平(表6),表明本氏针茅生境环境因子对本氏针茅的遗传多样性变化没有显著影响。
3 讨论
3.1 本氏针茅的遗传多样性 遗传多样性一般是指种内遗传多样性,即遗传特性的差异程度,用于评价某物种与其环境的相互作用方式及被改造和利用的潜力[23]。本研究利用ISSR分子标记对宁南山区6个本氏针茅自然种群120个单株材料进行PCR扩增,采用3种遗传多样性参数(多态性位点比率、Nei’s基因多样性和Shannon信息指数)对其遗传变异进行了分析。本氏针茅各种群的多态性位点比率在41.39%~68.44%,总多态性位点比率为97.95%。这可能是由于有些位点仅在个别群体中表现出多态或只在个别群体中可检测到,这些位点在各群体中分布的不均衡,导致总多态性位点比率很高[25]。一般认为,多态性位点比率在50%左右时,植物的遗传多样性是丰富的[26-28]。就多态性位点比率来看,本氏针茅种群具有较高的遗传多样性,但是,多态性位点比率简单直观,会受样本大小和条带总数的影响,且不能反映各条带的频率变化,对遗传多样性的评价只能是一个粗略的估计值[28]。因此,基于Hardy-Weinberg假设的Nei’s基因多样性和基于条带表型频率Shannon信息指数能更准确的评价种群遗传多样性[29]。本氏针茅种群Nei’s基因多样性(H=0.254 4)明显高于单子叶植物的平均值(H=0.190 0)[30],这个结果也说明本氏针茅种群具有较高的遗传多样性。在野外调查和取样的过程中发现,本氏针茅产生种子的能力并不低,但最后能萌发成实生苗的种子比例却较小。朱桂林等[31]发现针茅属植物的有性繁殖与水分因子密切相关,水分条件好,实生苗存活率高,有性繁殖能力强。在黄土高原恶劣的生境中,本氏针茅主要是以珠芽进行营养繁殖的。赵庆芳等[32]研究发现,种群内较高的遗传多样性可由补充较低比例的实生苗来维持。因此,宁南山区本氏针茅种群遗传多样性较高的主要原因可能与实生苗的不断补充有关。
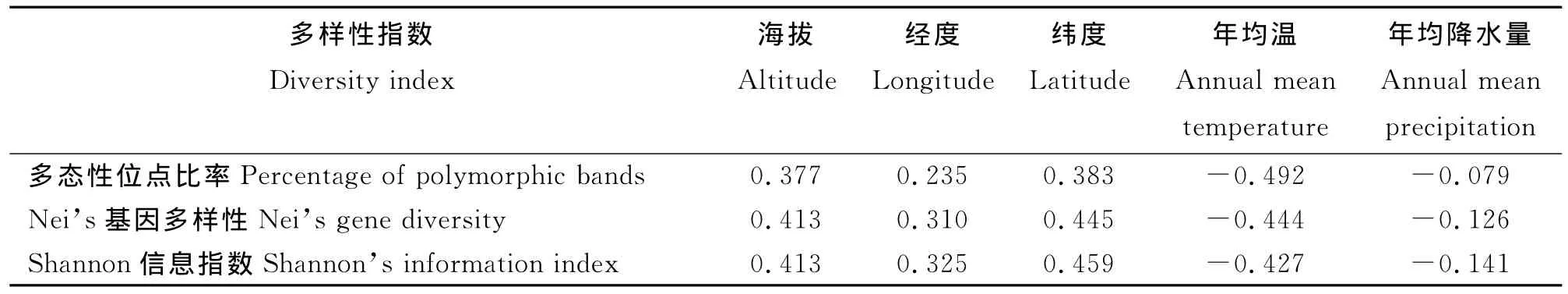
表6 本氏针茅种群遗传多样性指数与生态因子之间的Pearson相关性Table 6 Pearson correlation analyses for the relationships between genetic diversity indexes and ecological factors of Stipa bungeana
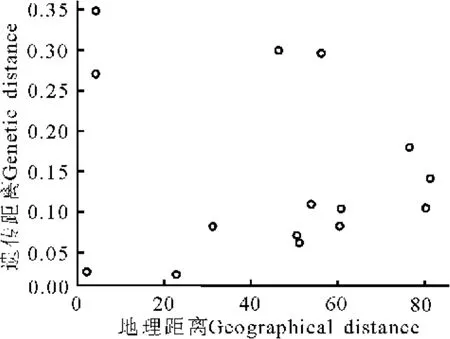
图3 6个本氏针茅种群间遗传距离与地理距离的相关关系Fig.3 The correlation between geneticdistance and geographic distance for six populations of Stipa bungeana
3.2 本氏针茅的遗传分化 群体遗传结构是指一个物种或群体的遗传变异在空间的非随机分布式样,即群体内、群体间的分布式样以及在时间上的变化,在很大程度上代表了其进化潜力[33]。因此,确定物种群体的遗传结构,有利于了解其生物学特性、探讨其进化过程和机制。群体遗传分化是衡量遗传结构的重要指标,表示种群间的变异占总的遗传变异中的比例[34]。利用分子方差变异AMOVA分析对本氏针茅种群的遗传结构进行分析,其遗传分化系数达到0.419,明显大于单子叶植物(Gst=0.231)和多年生草本植物(Gst=0.233)[35],说明本氏针茅种群发生了较大程度的遗传分化,其遗传变异主要存在于种群内,种群间的遗传变异相对较小。
Hamarick和 Godt[35]报道,植物的繁育系统、种子扩散机制、繁殖方式以及自然选择等因素对其遗传特征产生很大影响。作为典型的风媒传粉植物,本氏针茅的花粉和种子的传播与扩散无疑会影响其遗传分化。本研究的野外调查和取样生境为宁南山区,位于黄土高原半干旱区的典型草原,虽秋季多强风,但地形复杂,致使种子和花粉远距离传播受阻。另外,本氏针茅是牲畜所喜食的放牧型牧草,其春季萌发较早,在其他牧草尚未生长时就被牲畜大量采食,很难形成一个集中的高活力种子库,且无法依靠牲畜携带进行远距离传播。因此,本氏针茅种群内的遗传变异大于种群间的遗传变异的主要原因是种子无法远距离传播。
在对风媒植物的遗传分化研究中,许多学者认为[36-37],当种群间的遗传距离与地理距离之间不存在显著相关时,影响种群间遗传分化的主要因素则是遗传漂变或自然选择,而不是迁移或基因流。本研究中Mantel检验结果显示,在6个本氏针茅种群中,任意种群间的遗传距离和地理距离均没有显著的相关关系(P=0.733)。Volis等[38]曾认为,种群间的遗传分化与气候因子之间存在显著相关关系的关键就在于种群间的遗传分化是否因其遗传漂变引起。本研究中,来源于不同地理类群的本氏针茅,其遗传多样性指数与其不同生境的若干气候因子均未达到显著相关关系。综上所述,引起宁南山区本氏针茅种群间遗传分化较大的原因可能是由种子与花粉传播受阻以及遗传漂变。
3.3 种群保护措施 在黄土高原半干旱区特殊的生境中,本氏针茅有性繁殖能力较弱,以珠芽的营养繁殖为主,因此,一方面要采取相应的科学保护管理措施,加强对其原生境的保护,防止人为干扰,阻止种群遗传多样性的衰败;另一方面要加强对本氏针茅种子繁殖系统的深入研究,提高其在自然生境中的萌发力,以保护野生种质资源的可持续利用。
[1]卢胜莲,吴珍兰.中国针茅属植物的地理分布[J].植物分类学报,1996,34(3):242-253.
[2]汪诗平,李永宏.内蒙古典型草原退化机理的研究[J].应用生态学报,1999,10(4):437-441.
[3]赵念席,高玉葆,王金龙,等.大针茅种群RAPD多样性及其与若干生态因子的相关关系[J].生态学报,2006,26(5):1312-1319.
[4]赵念席,高玉葆,王金龙,等.内蒙古中东部草原大针茅的种群遗传分化[J].生态学报,2004,24(10):560-566.
[5]赵念席,高玉葆,王金龙,等.大针茅居群遗传分化和RAPD位点多态性分布的分析[J].云南植物研究,2008,30(5):557-564.
[6]赵念席,高玉葆,王金龙,等.内蒙古中东部草原区克氏针茅种群遗传分化的RAPD研究[J].生态学报,2004,24(3):2178-2185.
[7]张庆,牛建明,董建军.内蒙古地区短花针茅种群遗传多样性[J].生态学报,2008,28(7):3447-3455.
[8]宋涛,林枫,赵念席,等.利用RAPD技术分析内蒙古中东部草原三种针茅的遗传关系[J].草地学报,2006,14(3):328-332.
[9]周秋平,程积民,万惠娥,等.干旱胁迫下本氏针茅光合特性水分利用效率日动态研究[J].草地学报,2009,17(4):510-514.
[10]解新明,卢小良.SSR和ISSR标记及其在牧草遗传与育种研究中的应用前景[J].草业科学,2005,22(2):30-37.
[11]胡雪华,何亚丽,安渊,等.上海结缕草JD-1和结缕草属几个主要坪用草种的ISSR指纹分析[J].上海交通大学学报,2005,23(2):163-167.
[12]周国栋,李志勇,李鸿雁.老芒麦种质资源的研究进展[J].草业科学,2011,28(11):2026-2031.
[13]刘振虎,卢欣石,葛军.遗传标记在苜蓿遗传多样性研究中的应用[J].草业科学,2004,21(11):26-30.
[14]范彦,曾兵,张新全,等.中国野生鸭茅遗传多样性的ISSR研究[J].草业学报,2006,15(5):103-108.
[15]田青松,韩冰,杨劼,等.96份雀麦属材料遗传多样性的ISSR分析[J].中国草地学报,2010,32(1):18-25.
[16]俞靓,井赵斌,程积民.陕西地区本氏针茅自然种群遗传多样性的ISSR分析[J].草地学报,2012,20(3):512-517.
[17]Yu J,Jing Z B,Cheng J M.Genetic diversity ofStipa bungeanapopulations in the Loess Plateau of China using ISSR markers[J].African Journal of Biotechnology,2012,11(34):8425-8432.
[18]俞靓,井赵斌,魏琳,等.本氏针茅ISSR-PCR反应体系的建立及引物筛选[J].中国草地学报,2012,25(1):77-83.
[19]张富民,葛颂.群体遗传学研究中的数据处理方法I.RAPD数据的 AMOVA分析[J].生物多性,2002,10(4):438-444.
[20]Yeh F C,Boyle T J B.Population genetic analysis of codominant and dominant markers and quantitative traits[J].Belgian Journal of Botany,1997,129(1):157.
[21]Excofier L,Smouse P E,Quattro J M.Analysis of molecular variance inferred from metric distances among DNA haplotypes:Applications to human mitochondrial DNA restriction data[J].Genetics,1992,131:479-491.
[22]Mantel N A.The detection of disease clustering and a generalized regression approach[J].Cancer Research,1967,27:209-220.
[23]王东娜,牟长城,高卓,等.胡桃楸天然种群遗传多样性的ISSR分析[J].经济林研究,2011,29(2):22-29.
[24]高文伟,李培英,孙宗玖,等.新疆狗牙根种质遗传多样性的SSR分析[J].草业科学,2010,27(12):58-64.
[25]李钧敏,金则新,钟章成.不同海拔高度大血藤群体遗传多样性的RAPD分析及其与环境因子的相关性[J].生态学报,2004,24(3):567-573.
[26]马小军,汪小全,徐昭玺,等.人参不同栽培群体遗传关系的 RAPD分析[J].植物学报,2000,42(6):587-590.
[27]刘果厚,贾宝丽.浑善达克沙地榆遗传多样性的研究[J].干旱区资源与环境,2003,17(5):123-128.
[28]孙坤,陈纹,马瑞君,等.子午岭中国沙棘亚居群的遗传多样性研究[J].兰州大学学报,2004,40(3):72-75.
[29]钱韦,葛颂.居群遗传结构研究中显性标记数据分析方法初探[J].遗传学报,2001,28(3):244-255.
[30]Nybom H.Comparison of different nuclear DNA markers for estimating intra-specific genetic diversity in plants[J].Molecular Ecology,2004,13:1143-1155.
[31]朱桂林,卫智军,杨静,等.放牧制度对短花针茅群落植物种群地上生物量的影响[J].中国草地,2002,24(3):15-23.
[32]赵庆芳,李巧峡,马世荣,等.青藏高原东部矮生蒿草遗传多样性的 RAPD研究[J].生态学报,2006,26(8):2494-2501.
[33]Sun M.Effects of population size,mating system,and evolutionary origin on genetic diversity inSpiranthes sinensisandS.hongkongensis[J].Conservation Biology,1996,10:785-795.
[34]Hamrick J L,Loveless M D.Associations between the breeding system and the genetic structure of tropical tree populations[A].In:Bock J,Linhart Y B.Evolutionary Ecology of Plants[M].Boulder:Westview Press,1989:129-146.
[35]Hamrick J L,Godt M J W.Allozyme diversity in plant species[A].In:Brown A H D,Clegg M T,Kahler A L,etal.Plant Population Genetics,Breeding and Genetic Resources[C].Sunderland,Massachusetts:Sinauer Associates Inc.,1990:43-63.
[36]Wen C S,Hsiao J Y.Genetic differentiation ofLilium longiflorumThunb.var.ScabrumMasam.(Liliaceae)in Taiwan using Random Amplified Polymorphic DNA and morphological characters[J].Botanical Bulletin of Academia Sinica,1999,40:65-71.
[37]冯亮亮,唐红,李毅,等.甘肃红砂不同种群遗传多样性的ISSR分析[J].草业学报,2011,20(1):125-130.
[38]Volis S,Yakubov B,Shulgina I,etal.Tests for adaptive RAPD variation in population genetic structure of wild barley,HordeumspontaneumKoch[J].Biological Journal of Linnean Society,2001,74:289-303.
