基于宏基因组方法对西藏传统发酵牦牛奶中微生物多样性的研究
2012-12-07孙志宏刘文俊张和平
孙志宏, 刘文俊, 张和平
(内蒙古农业大学乳品生物技术与工程教育部重点实验室,内蒙古呼和浩特 010018)
基于宏基因组方法对西藏传统发酵牦牛奶中微生物多样性的研究
孙志宏, 刘文俊, 张和平
(内蒙古农业大学乳品生物技术与工程教育部重点实验室,内蒙古呼和浩特 010018)
采用454高通量测序技术从宏基因组角度对西藏地区自然发酵牦牛奶中微生物多样性进行分析,以细菌16S rRNA的V3区和真菌的ITS区序列为扩增、测序靶点,分别获得3 051和3 396条高质量序列.研究发现,西藏自然发酵牦牛奶样品中真菌的丰度要大于细菌.在门的水平上,细菌和真菌分别由硬壁菌门(Firmicutes)和子囊菌门(Ascomycota)组成,而在属的水平上细菌和真菌的优势菌分别为乳杆菌属(Lactobacillus)和耐碱酵母属(Galactomyces).
自然发酵牦牛奶;微生物多样性;454测序
酸牦牛奶(Kurut)是我国西藏地区由藏民制作的一种自然发酵乳制品,是青藏高原地区藏民重要的饮食和经济来源.Kurut呈纯白色、黏稠状,通常在10~20℃的自然环境中发酵3~6 d,产生具有酸度和酒精混合所特有的味道[1].其脂肪含量为5.37%,总蛋白含量约5.44%,平均大约是酸奶中脂肪和蛋白质含量的2倍.同时Kurut中钙(140 mg/100 g)、磷(146 mg/100 g)、镁(154 mg/100 g)、锌(5.74 mg/100 g)和B族维生素含量均高于普通酸奶[2].由于Kurut具有理想的营养成分以及特有的口感,许多研究机构不断模仿并设计该产品的生产加工工艺,以期产业化这一发酵乳制品.
全面深入地了解微生物多样性以及菌群组成是酸牦牛奶首要解决的问题.Kurut中的微生物主要是由乳酸菌和酵母菌组成,保加利亚乳杆菌(Lactobacillus delbrueckii subsp.bulgaricus)和嗜热链球菌(Streptococcus thermophilus)同样也是其主要的发酵剂成分[1-2].然而,关于发酵乳中微生物多样性及群落研究大多局限于传统的纯培养方法,这种方法虽然可以分离到部分微生物,但是由于培养条件的局限性,不能完全分析样品中微生物的组成,往往会低估了微生物多样性.
随着分子生物学技术的不断发展,基于聚合酶链式反应(polymerase chain reaction,PCR)技术出现了许多非培养的方法,像16S rRNA克隆建库、DGGE/TGGE以及芯片等方法被广泛应用于发酵食品和其他微生态环境中微生物多样性的研究.平行高通量焦磷酸测序技术(parallel high throughput pyrosequencing)是 Margulies等[3]优化改良焦磷酸测序技术后应用到微生物的多样性分析技术.由于其具有高通量、快速等优点,该方法一经出现就被广泛应用于各种生态系统中,比如深矿井,土壤,深海生物圈,慢性创伤部位、人类口腔和肠道等微生态环境中微生物多样性的检测和研究中.近年来,焦磷酸测序也逐渐应用到传统发酵食品中微生物的多样性研究,如开菲尔粒[4-6]、珍珠粟浆[7]、干酪[8]等发酵食品.
本研究以采集自我国西藏地区的2份自然发酵牦牛奶(酸牦牛奶)为研究材料,通过焦磷酸测序完成对样品中细菌以及真菌的多样性分析,全面了解自然发酵牦牛奶中微生物组成,为加快酸牦牛奶产业化提供理论基础.
1 材料与方法
1.1 材料与试剂
自然发酵牦牛奶样品XZ15和XZ16分别采集自西藏那曲县罗马镇和西藏当雄县纳木错(平均海拔4 300 m以上)的牧户.
PCR引物由上海桑尼生物科技有限公司合成.Taq酶(DR001C)由宝生物工程(大连)有限公司提供.
1.2 仪器与设备
5810R型冷冻离心机,德国 Eppendorf公司;GDS-8000型凝胶成像仪,美国UVP公司;DYY-12型电泳仪,北京六一公司;PTC-200型梯度PCR扩增仪,美国伯乐公司;2100型生物分析仪,美国安捷伦公司;Roche GS FLX型测序仪,美国Roche 454公司.
1.3 方法
1.3.1 基因组DNA的提取
采用CTAB-SDS冻融法提取样品中微生物宏基因组DNA.依据张春林等[9]的方法并进行了适当的调整.取1 mL发酵乳样品于7 mL离心管中,立即置于液氮中完全冻结,取出后放入65℃水浴中融化(5 min),反复3次,而后加3 mL DNA提取液(100 mol/L Tris-HCl;100 mmol/L EDTA;100 mmol/L Na2HPO4;1.5 mol/L NaCl,1%CTAB,pH 8.0)混合,然后加0.3 mL SDS(10%)和10 μL 蛋白酶K(10 mg/mL),于50℃恒温摇床中100 r/min摇动2 h,然后室温下10 000 r/min离心10 min,收集上清转移至另一离心管中,离心管中其余沉淀再加入0.9 mL DNA提取液和0.1 mL 20%SDS,涡旋10 s,65℃水浴5 min,于液氮中冷冻、65℃融化,反复3次重复上述操作,收集上清液与前一次合并.上清液与等体积的氯仿于10000 r/min离心10 min,吸取上清液转移至另一离心管中,加入等体积的V(酚)∶V(氯仿)∶V(异戊醇)=25∶24∶1,轻轻混匀 2 min,室温12 000 r/min离心5 min,收集上清液加入0.1倍体积的NaAc,轻柔混匀,加入1倍体积的冰异丙醇,混合、静置2 min,于12 000 g离心5 min沉淀DNA,然后加入70%乙醇洗涤沉淀,沉淀自然风干后,重悬于20 μL灭菌的去离子水中冷藏备用.
1.3.2 16S rRNA V3区PCR扩增及焦磷酸测序
为了能够把2个样品放在一次测序中完成,在引物的5'段加6个碱基的barcode(见表1)来区分样品.PCR扩增反应体系为1×PCR缓冲液(含有2.5 mmol·L-1MgCl2)、上下游引物 3 pmol、dNTPs 200 mmol·L-1和 1 μL 50 ng/μL 提取的 DNA 模板,用水补齐到50 μL.扩增循环条件为94℃变性30 s、55℃退火30 s、72℃延伸30 s,循环30次.扩增产物经1%琼脂糖凝胶电泳检验合格后,通过产物纯化、安捷伦2100生物分析仪定量混匀,上样测序.焦磷酸测序按照454 Roche GS-FLX的标准流程进行测试.

表1 所用PCR引物Tab.1 PCR primer used in this study
1.4 数据分析
获得的数据提取高质量序列、划分操作分类单元(operational taxonomic units,OTUs)等依据Mothur程序包[10]来完成.原始数据需要经过质量控制,需满足序列引物和Barcode序列完全匹配、序列长度大于150 bp、序列中不存在有争议的碱基、多聚体结构不得超过7个、每50个碱基质量值平均不低于35.通过序列的聚类以给定非相似度(cutoff)0.03水平上划分OTU,选用每一OTU代表序列进行注释,采用BLAST比对方法细菌于SILVA数据库[11]进行注释,真菌于UNITE数据库[12]进行注释.系统发育树构建由Megan程序包[13]依据OTU注释信息完成.
2 结果与分析
2.1 丰度和多样性分析
通过454高通量测序,XZ15号酸牦牛奶样品得到的高质量细菌和真菌序列分别为1 626条和2 101条,XZ16号样品为1 425条和1 295条.在给定非相似度97%的水平上进行OTU归并,XZ15和XZ16号样品分别得到32个和9个细菌OTU,63个和48个真菌OTU.两个酸牦牛奶样品细菌和真菌的稀疏曲线(rarefaction curves)均未进入平台期(见图1),这就意味着随着测序量的增加,新的细菌或者真菌种系将被发现.同时也表明XZ15酸牦牛奶样品其细菌和真菌的丰度要明显高于XZ16样品.2011年,Dobson等[4]采用焦磷酸测序对3个开菲尔粒样品的细菌组成进行分析,平均每个样品产生了5 805条序列,其结果表明已进入平台期.结合本研究结果,指示出后期实验还应进一步提高测序量,以尽可能的分析样品中微生物的组成.
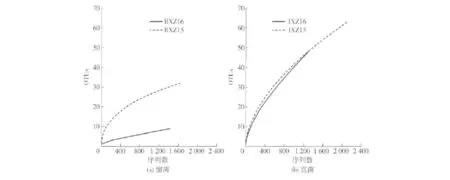
图1 两个酸牦牛奶样品中细菌和真菌的稀疏曲线Fig.1 Rarefaction analysis of pyrosequencing reads in bacteria and fungi from two Kurut samples
通过chao1指数对样品的丰度进行比较,XZ15号和XZ16号酸牦牛奶样品中细菌的chao1指数分别为58和16.5,真菌分别为129.1和130.7.由此可见,两份酸牦牛奶中真菌的含量均大于细菌,且XZ15号样品中细菌的含量要远大于XZ16号样品,而其真菌含量比较接近.通过simpson指数对样品的多样性进行比较,XZ15号和XZ16号酸牦牛奶样品中细菌的simpson指数分别为2.058和1.030,真菌分别为1.806和1.735.由此可见,XZ15号酸牦牛奶中虽然真菌的含量大于细菌,但其多样性和细菌相近,而XZ16号样品中细菌的含量和多样性均低于真菌.这与张春林[9]、Airidengcaicike[14]等报道kurut中真菌含量大于细菌的结论不同,可能是由于采用纯培养技术对真菌培养方法单一而致.
2.2 样品中微生物相对含量的比较分析
通过OTU的划分,选取出现次数较多的序列为一个OTU的代表序列,采用BLAST的比对方法完成序列的注释和分类学地位的确定.XZ15酸牦牛奶中的细菌分别属于硬壁菌门(Firmicutes)、变形菌门(Proteobacteria)和拟杆菌门(Bacteroidetes)3个细菌门,其含量分别为80.1%,12.9%和6.9%;而XZ16样品中的细菌分别属于硬壁菌门(Firmicutes)和变形菌门(Proteobacteria),其含量分别为99.8%和0.2%,参见图2、图3.
由图2、图3知,两个酸牦牛奶样品中的细菌主要隶属于硬壁菌门(Firmicutes).在属的水平上,XZ15酸牦牛奶中的细菌分别隶属于乳杆菌属(Lactobacillus)、乳球菌属(Lactococcus)、明串球菌属(Leuconostoc)、片球菌属(Pediococcus)、嗜热链球菌属(Streptococcus)和魏氏菌属(Weissella)等6个细菌属,其含量分别为 72.3%,7.0%,0.1%,0.1%,0.5%和0.2%;XZ16酸牦牛奶中的细菌分别隶属于乳杆菌属(Lactobacillus)、乳球菌属(Lactococcus)和嗜热链球菌属(Streptococcus)3个属,其含量分别为98.7%,0.1%和1.0%.由此可见,乳杆菌属(Lactobacillus)是两份酸牦牛奶中的优势菌属.此外,比较细菌的相对含量不难发现,XZ15酸牦牛奶样品中细菌的多样性要大于XZ16.Airidengcaicike等[14]通过传统的纯培养手段发现西藏地区酸牦牛奶中优势菌群为发酵乳杆菌(Lactobacillus fermentum)和干酪乳杆菌(Lactobacillus casei),与本研究取得结果一致.

图2 两个酸牦牛奶样品中细菌的组成Fig.2 Compositions of bacteria in two Kurut samples
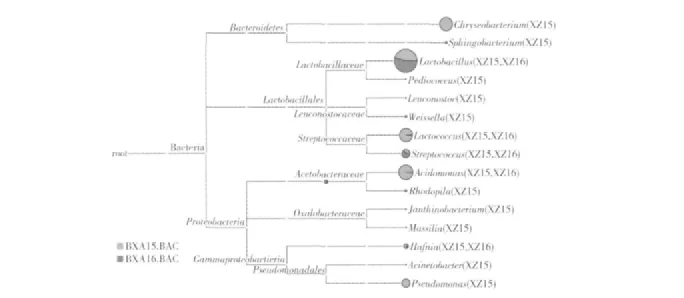
图3 两个酸牦牛奶样品中细菌相对含量的比较Fig.3 Relative abundance(percentage of sequences)of bacteria in two Kurut samples.
XZ15号酸牦牛奶中共发现14个真菌属,其中含量大于1%的真菌属分别为耐碱酵母属(Galactomyces)、克鲁维酵母菌属(Kluyveromyces)、红冬孢酵母属(Rhodosporidium)和双足囊菌属(Dipodascus),其含量分别为84.2%,8.0%,2.5%和2.0%;XZ16号酸牦牛奶中共发现10个真菌属,其中含量大于1%的真菌属分别为耐碱酵母属(Galactomyces)、克鲁维酵母菌属(Kluyveromyces)和酵母属(Saccharomyces),其含量分别为79.3%,17.2%和1.0%,参见图4、图5.

图4 两个酸牦牛奶样品中真菌的组成Fig.4 Compositions of fungus in two Kurut samples
由图4、图5可见,耐碱酵母属(Galactomyces)是2份酸牦牛奶中的优势真菌属,其次为克鲁维酵母菌属(Kluyveromyces).倪慧娟等分析了新疆地区酸马奶[15]、酸驼乳[16]中酵母菌的多样性,其结果都表明克鲁维酵母菌属(Kluyveromyces)和酵母属(Saccharomyces)是2种自然发酵乳的优势菌属.造成不同优势菌群的主要原因可能是样品来源不同、环境不同而造成.早在1996年,Montanari等[17]对来自不同地区的94份酸马奶样品进行了酵母菌分离鉴定后,发现多数样品中含有单孢酵母(Saccharomyces unisporus),并且它的数量随海拔位置的提高而增加,是传统酸马奶酒精发酵的主要微生物之一.本研究样品均采自海拔4 300 m以上自然发酵牦牛奶,可能耐碱酵母属(Galactomyces)更加适应当地的环境,经历了不断进化地选择,逐渐形成了西藏自然发酵牦牛奶的优势菌属.同时基于纯培养技术可能由于培养条件的限制,导致传统方法分析的菌群也具有片面性.
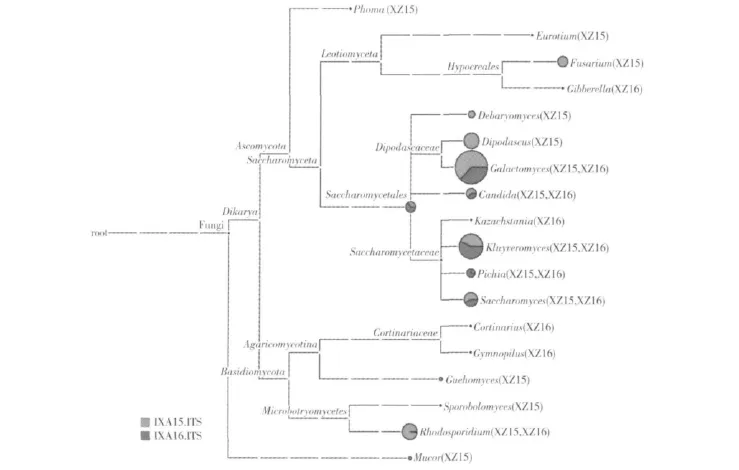
图5 两个酸牦牛奶样品中真菌相对含量的比较Fig.5 Relative abundance(percentage of sequences)of fungus in two Kurut samples
3 结论
本研究采用高通量测序技术对西藏自然发酵牦牛奶的微生物多样性及组成进行了分析,第一次从宏基因组水平对传统发酵牦牛奶中的微生物菌群进行探讨.研究发现,酸牦牛奶样品细菌的优势菌为乳杆菌属(Lactobacillus),真菌的优势菌为耐碱酵母属(Galactomyces),其中真菌的丰度要大于细菌.通过测序分析,不仅证实了前期采用基于纯培养方法的部分研究结果,也发现了以前未曾报道的现象,如真菌的含量以及多样性差异、细菌多样性等,均表明通过高通量测序可更全面地分析自然发酵牦牛奶中微生物的多样性.同时通过本研究,更加全面地了解了发酵牦牛奶的微生物组成和多样性,为加快这一传统发酵乳的产业化进程提供基础数据.
[1]Liu Wenjun,Sun Zhihong,Zhang Yanbin,et al.A survey of the bacterial composition of kurut from Tibet using a culture-independent approach[J].Journal of Dairy Science,2012,95:1064-1072.
[2]Zhang Heping,Xu Jie,Wang Junguo,et al.A survey on chemical and microbiological composition of kurut,naturally fermented yak milk from Qinghai in China[J].Food Control,2008,19:578-586.
[3]Margulies M,Egholm M,Altman W,et al.Genome sequencing in microfabricated high-density picolitre reactors[J].Nature,2005,437:376-380.
[4]Dobson A,O'Sullivan O,Cotter P D,et al.Highthroughput sequence-based analysis of the bacterial composition of kefir and an associated kefir grain[J].FEMS Microbiology Letters,2011,320:56-62.
[5]Leite A M O,Mayoa B,Rachid C T C C,et al.Assessment of the microbial diversity of Brazilian kefirgrains by PCR-DGGE and pyrosequencing analysis[J].Food Microbiology,2012,31:215-221.
[6]Humblot C,Guyot J P.Pyrosequencing of tagged 16S rRNA gene amplicons for rapid deciphering of the microbiomes of fermented foods such as pearl millet slurries[J].Applied and Environmental Microbiology,2009,75:4354-4361.
[7]Turpin W,Humblot C,Guyot J P.Genetic screening of functional properties of lactic acid bacteria in a fermented pearl millet slurry and in the metagenome of fermented starchy foods[J].Applied and Environmental Microbiology,2011,77:8722-8734.
[8]Wafa M,Monica T,Finn K V,et al.Characterization of bacterial populations in Danish raw milk cheeses made with different starter cultures by denaturating gradient gel electrophoresis and pyrosequencing[J].International Dairy Journal,2011,21:142-148.
[9]张春林,刘文俊,张彦斌,等.西藏地区传统发酵乳宏基因组DNA的提取及微生物群落多样性分析[J].食品与发酵工业,2010,36(4):15-20.
[10]Schloss P D,Westcott S L,Ryabin T,et al.Introducing mothur:open-source,platform-independent,community-supported software for describing and comparing microbial communities[J].Applied and Environmental Microbiology,2009,75:7537-7541.
[11]Pruesse E,Quast C,Knittel K,et al.SILVA:a comprehensive online resource for quality checked and aligned ribosomal RNA sequence data compatible with ARB[J].Nucleic Acids Research,2007,35:7188-7196.
[12]Abarenkov K,Henrik Nilsson R,Larsson K H,et al.The UNITE database for molecular identification of fungi-recent updates and future perspectives[J].The New Phytologist,2010,186:281-285.
[13]Huson D H,Auch A F,Qi J,et al.MEGAN analysis of metagenomic data[J].Genome Research,2007,17:377-386.
[14]Airidengcaicike,Xia Chen,Du Xiaohua,et al.Isolation and identification of cultivable lactic acid bacteria in traditional fermented milk of Tibet in China[J].International Journal of Dairy Technology,2010,63:437-444.
[15]倪慧娟,包秋华,孙天松,等.新疆地区酸马奶中酵母菌的鉴定及其生物多样性分析[J].微生物学报,2007,47:578-582.
[16]倪慧娟,孙天松,孙志宏,等.新疆自然发酵驼乳中酵母菌的分离鉴定及部分酶学特性研究[J].食品工业科技,2009,30(7):170-172.
[17]Montanari G,Zambonelli C,Grazia L,et al.Saccharomyces unisporus as the principal alcoholic fermentation microorganism of traditional koumiss[J].Journal of Dairy Research,1996,63:327-331.
(责任编辑:檀彩莲)
Study on Microbial Diversity in Naturally Fermented Yak Milk of Tibet Based on Metagenomics
SUN Zhi-hong, LIU Wen-jun, ZHANG He-ping
(Key Laboratory of Dairy Biotechnology and Engineering,Ministry of Education,Inner Mongolia Agriculture University,Huhhot 010018,China)
In this study,the microbial diversity of metagenomics in naturally fermented yak milk(kurut)were investigated using barcoded pyrosequencing.A total of 3 051 unique V3 variable regions of the 16S rRNA gene of bacterium and 3 396 unique ITS variable regions of fungi were analyzed from two kurut samples.The results indicated that the abundance of fungi was higher than bacteria in the kurut samples.The Firmicutes and Ascomycota were the most abundant bacteria and fungi phyla in kurut samples.At genus levels,the bacteria of kurut were largely composed of the Lactobacillus while Galactomyces was the dominant genus within the fungus of kurut.
naturally fermented yak milk;microbial diversity;454 pyrosequencing
TS201.3
A
1671-1513(2012)04-0019-06
2012-06-15
国家杰出青年基金资助项目(31025019);教育部创新团队发展计划资助项目(IRT0967).
孙志宏,男,助理研究员,硕士,主要从事乳酸菌分子生物学方面的研究;
张和平,男,教授,博士,主要从事乳品生物技术方面的研究.通讯作者.
编者按:微生物在乳制品的生产和贮藏过程中起着至关重要的作用.为了提高乳制品的品质和营养特性,一方面要全面掌握传统发酵乳制品的微生物特性,进而筛选、强化有益菌的作用;另一方面要采用现代乳制品高新加工技术抑制腐败菌的生长,达到保持营养品质、延长货架期的目的.在“基于宏基因组方法对西藏传统发酵牦牛奶中微生物多样性的研究”一文中,通过焦磷酸测序对我国西藏地区的传统发酵牦牛奶中细菌、真菌的多样性进行分析,从宏基因组水平对传统发酵牦牛奶中的微生物菌群进行探讨,证实了真菌丰度大于细菌的现象,为加快酸牦牛奶产业化提供了基础数据.在“不同贮藏温度对UHT乳品质的影响”一文中,研究了采用超高温瞬时灭菌之后液态奶的品质变化,分析不同贮藏温度和时间下UHT乳蛋白质水解、脂肪氧化等指标的变化规律,探讨造成UHT乳品质变化的根本原因,以期为UHT乳的运输、销售及贮藏环境的选择提供理论依据和参考.(栏目主持人:张德权研究员)
