某三级甲等医院肺炎克雷伯菌耐药性及耐药基因分析Δ
2012-11-28和晋渝王世博李迎丽邱景富
贾 燕,和晋渝,王世博,李迎丽,邱景富
(重庆医科大学公共卫生与管理学院,重庆 400016)
某三级甲等医院肺炎克雷伯菌耐药性及耐药基因分析Δ
贾 燕*,和晋渝,王世博,李迎丽#,邱景富
(重庆医科大学公共卫生与管理学院,重庆 400016)
目的:分析某三级甲等医院连续4年肺炎克雷伯菌的耐药性及耐药基因分布,了解耐药性变化趋势,指导临床合理用药。方法:使用VITEK-32全自动微生物分析仪进行细菌鉴定,用纸片法和最低抑菌浓度(MIC)试验进行药敏分析,用聚合酶链式反应(PCR)对产超广谱β-内酰胺酶(ESBLs)菌株可能携带的主要β-内酰胺酶耐药基因进行检测。结果:在182株临床分离的肺炎克雷伯菌中,痰和咽拭子标本分离率占首位(78.6%);对检测的13种抗菌药物耐药谱分析显示,该菌对哌拉西林耐药率最高,对亚胺培南和美罗培南敏感;产ESBLs菌株的检出率约30%;51%以上的产ESBLs菌株携带至少1种所检测的耐药基因,以blaTEM为主。结论:该院肺炎克雷伯菌分离株耐药范围广、产ESBLs菌检出率高,应引起重视。
肺炎克雷伯菌;耐药性;超广谱β-内酰胺酶;β-内酰胺酶基因
肺炎克雷伯菌是呼吸道感染中常见致病菌之一,也是医院感染和机会感染的重要病原菌[1]。近年来,肺炎克雷伯菌的耐药性呈上升趋势,临床大量、不合理使用抗生素造成了呼吸道感染菌群变化及其耐药性增加,其耐药性的变迁和现状也备受关注[2]。本文回顾性分析连续4年某三级甲等医院临床分离肺炎克雷伯菌的分布、耐药性、产超广谱β-内酰胺酶(ESBLs)菌株的检出率及β-内酰胺酶基因的存在情况,以了解临床常用抗菌药物的耐药性现状和耐药率的变化,为临床诊断及治疗、合理使用抗菌药物提供理论依据。
1 材料与方法
1.1 材料
1.1.1 标本来源。某三级甲等医院连续4年住院患者送检的痰液、咽拭子、尿、血等标本,按常规方法接种于血平板。同一人多次培养阳性结果只计1株。
1.1.2 仪器。VITEK-32全自动微生物分析仪(法国Bio-Merieux公司)。
1.2 方法
1.2.1 菌种鉴定。所有菌株分离纯化得到单菌落后鉴定到种。
1.2.2 药敏试验。采用纸片法和最低抑菌浓度(MIC)试验测定肺炎克雷伯菌对阿米卡星、庆大霉素、哌拉西林、哌拉西林/他唑巴坦、亚胺培南、美罗培南、头孢唑林、头孢吡肟、头孢噻肟、头孢他啶、头孢西丁、头孢呋辛、环丙沙星等13种抗菌药物的耐药性。判定标准依据美国临床实验室标准化协会(CLSI)2010年版的规定[3]。
1.2.3 产ESBLs菌株的确证试验。采用CLSI 2010年版推荐的药敏纸片扩散表型确证法[3]进行检测,判定标准为使用2组药敏纸片:头孢他啶(CAZ)、头孢他啶/克拉维酸、头孢噻肟(CTX)、头孢噻肟/克拉维酸(CTX/CA),当2种药物中任意一组加克拉维酸与不加克拉维酸的抑菌圈直径增大≥5 mm时,可确认为产ESBLs菌株。质控菌株为肺炎克雷伯菌ATCC 700603。
1.2.4 聚合酶链式反应(PCR)法检测产ESBLs菌株可能携带的主要6种耐药基因。用煮沸法提取细菌DNA,各型β-内酰胺酶基因引物设计参照文献合成。PCR产物经纯化进行双向测序,测序结果在GenBank进行比对分析。
2 结果
2.1 肺炎克雷伯菌在各类标本中的分布
对182株临床分离的肺炎克雷伯菌在各分离标本中的分布进行分析,来源于痰和咽拭子标本的共143株,占总数的78.6%,分离率占首位;分离自尿、血和其他标本的分别为12、11和16株,共占21.4%,远远低于痰和咽拭子标本的分离率。
2.2 肺炎克雷伯菌的耐药率
肺炎克雷伯菌对13种常用抗菌药物的耐药率见表1。
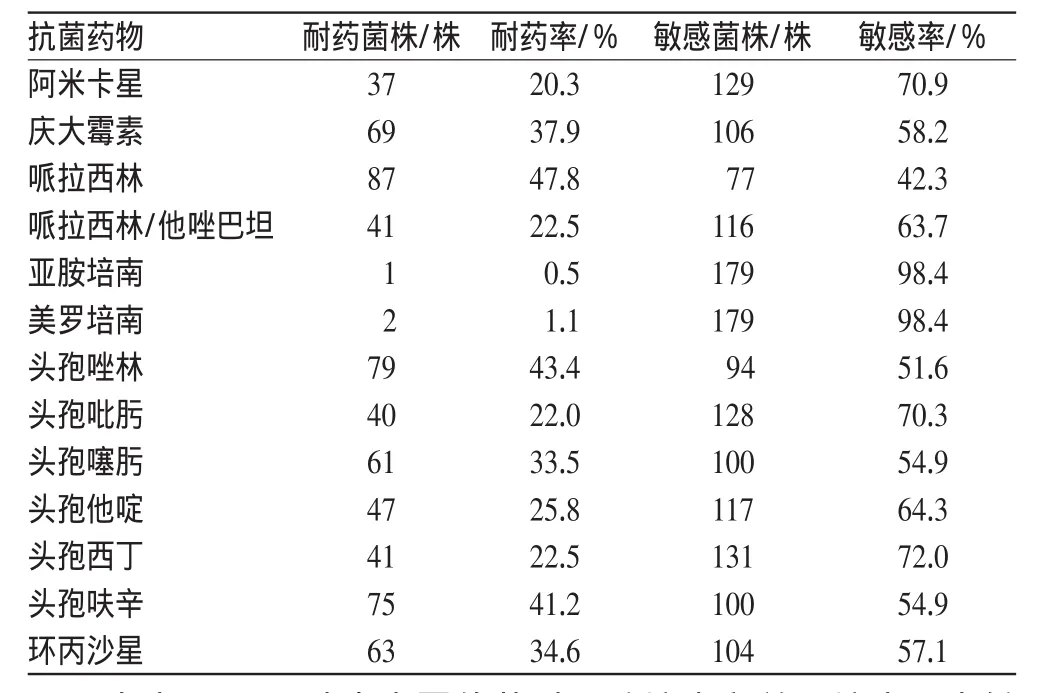
表1 182株肺炎克雷伯菌对13种抗菌药物的耐药率统计Tab 1 Resistance rates of 182 clinical isolates of K.pneumoniae to 13 kindsof antibiotics
由表1可见,肺炎克雷伯菌对亚胺培南和美罗培南最为敏感,敏感率均为98.4%;耐药率最高的为哌拉西林,高达47.8%。
2.3 产ESBLs与非产ESBLs菌株耐药率分析
临床分离的182株肺炎克雷伯菌中,产ESBLs菌株共54株,产ESBLs菌株的检出率约为30%(54/182=29.7%)。产ESBLs与非产ESBLs菌株对13种常用抗菌药物的耐药率见表2。
2.4 肺炎克雷伯菌产ESBLs菌株的主要耐药基因检测
用PCR法对43株产ESBLs肺炎克雷伯菌可能携带的主要β-内酰胺酶基因(blaTEM、blaSHV、blaOXA-1群、blaCTX-M-1群、blaCTX-M-2群和blaCTX-M-9群)进行分析,PCR扩增所用引物序列见表3。PCR扩增产物的电泳图见图1,扩增产物经测序后进行比对分析。结果表明,51%以上的产ESBLs菌株携带至少1种所检测的耐药基因,其中有6株菌同时携带2~3种耐药基因(blaTEM、blaSHV和blaCTX-M-1),有21株携带有blaTEM基因(48.8%),3株blaSHV基因扩增阳性(7.0%),5株blaCTX-M-1基因扩增阳性(11.6%),而bla-OXA-1、blaCTX-M-2和blaCTX-M-9未检测到阳性菌株。

表2 182株菌中产ESBLs与非产ESBLs菌株对13种抗菌药物的耐药率统计Tab 2 Resistance rates of ESBLs-producing and non-ESBLs-producing strains in 182 clinical isolates to 13 kinds of antibiotics

表3 产ESBLs菌株β-内酰胺酶基因检测引物统计Tab 3 The list of primers used forβ-lactamases gene detection in ESBLs-producing isolates
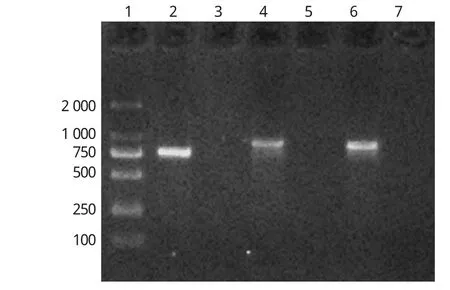
图1 PCR产物电泳图1.Marker DL2000;2、4、6.分别为blaTEM、blaSHV和blaCTX-M-1基因PCR扩增产物;3、5、7.分别为阴性对照Fig 1 Electrophoresis of PCR products1.Marker DL2000;2,4,6.PCR products for blaTEM,blaSHV and blaCTX-M-1 gene;3,5,7.negativecontrol
3 讨论
肺炎克雷伯菌是引起医院内感染尤其是呼吸道感染的常见条件致病菌之一,其引起的感染多发生在肺、泌尿道、血液和创伤面等部位,可引起肺炎、尿路感染、菌血症等。本文对182株临床分离肺炎克雷伯菌进行了分析,78.6%的分离株来自痰和咽拭子标本,提示该菌主要引起呼吸道感染,与文献报道一致[8]。
由于抗菌药物的广泛使用,肺炎克雷伯菌耐药菌株呈逐年上升趋势。本文数据分析表明,该菌对第2代头孢菌素有较高的耐药性,对第3、4代头孢菌素耐药性也在不断增加,而对亚胺培南、美罗培南较为敏感,碳青霉烯类可作为肺炎克雷伯菌感染的首选抗菌药物。
产ESBLs是肺炎克雷伯菌对β-内酰胺酶类抗生素主要的多重耐药机制,ESBLs不仅能分解广谱青霉素和第1、2、3代头孢菌素,而且还能分解第4代头孢菌素等β-内酰胺类抗生素,同时对氨基糖苷类和喹诺酮类有交叉耐药,给临床治疗感染性疾病带来很大困难。本研究结果显示,产ESBLs菌株的检出率约30%,与国内报道的发生率基本一致[9]。产ESBLs的肺炎克雷伯菌呼吸道感染最为多见,提示医院感染管理机构应加强对病患呼吸道的监控护理,警惕耐药株产生引起医院感染的发生。
本文对产ESBLs菌与非产ESBLs菌的耐药性进一步分析,结果显示产ESBLs菌的耐药性显著高于非产ESBLs菌株。产ESBLs菌株对除亚胺培南、美罗培南外其他11种检测抗菌药物都耐药。产ESBLs菌与非产ESBLs菌对亚胺培南均敏感,建议保护性使用此药,维持其抗菌活性,可用于治疗ESBLs菌株引起的重度感染。
ESBLs型别日益增多,新的β-内酰胺酶基因也不断地被报道,目前主要以blaTEM、blaSHV和blaCTX-M型为主。本文结果显示该院流行的主要基因型为blaTEM,与周军等[10]的报道一致,而与朱卫民等[4]的数据有些不同,推测可能与样本收集时间、来源等不一样有关。分析结果表明该院存在较为严重的ESBLs流行现状,β-内酰胺酶基因的存在和传播可能是导致产ESBLs菌株流行的主要原因之一。为降低产ESBLs菌株的发生率,应加强控制抗菌药物的使用。
抗菌药物的使用对细菌耐药性有明显的选择性压力,抗菌药物在医院、社区滥用是产生耐药的重要原因。应控制医院感染,开展常规病原学检测并定期公布,严格执行消毒隔离制度,避免或减少医源性交叉感染;循环使用抗菌药物,根据肺炎克雷伯菌耐药谱变化及年用量,限制用药范围和用量,抑制耐药菌株流行。医院应建立细菌耐药监测体系,及时开展细菌耐药谱的分析,掌握耐药趋势,以指导临床合理用药,控制多重耐药菌株的产生与传播。
[1]Gaynes R,Edwards JR.Overview of nosocomial infections caused by gram-negative bacilli[J].Clin Infect Dis,2005,41(6):848.
[2] 杨建良,吴龙章,潘美玉,等.96例下呼吸道感染肺炎克雷伯菌患者的临床表现与药敏结果分析[J].中国药房,2009,20(5):352.
[3] CLSI.Performance Standards for Antimicrobial Susceptibility Testing:Twentieth Informational Supplement[S].2010.
[4] 朱卫民,陈勇川,杨 春,等.产ESBLs和AmpC肺炎克雷伯菌医院感染分子流行病学研究[J].重庆医科大学学报,2010,35(2):304.
[5] 王冬国,周铁丽.产AmpC酶的克雷伯菌药敏结果分析及质粒介导的AmpC和ESBLs耐药基因检测[J].中国抗生素杂志,2007,32(4):239.
[6] 史伟峰,王玉月,李美忠,等.产超广谱β-内酰胺酶的肺炎克雷伯菌相关耐药基因研究[J].检验医学,2009,24(3):215.
[7] 王继东,钱小毛,糜祖煌,等.1株多重耐药肺炎克雷伯菌耐药基因和整合子、转座子遗传标记研究[J].微生物与感染,2006,1(4):229.
[8] 赵广玉,黄继勋.肺炎克雷伯菌耐药及抗菌药物应用分析[J].中国药房,2008,19(32):2 518.
[9] 万忠林,周环宇.产超广谱β-内酰胺酶大肠埃希菌和肺炎克雷伯菌的耐药性分析[J].中国药业,2010,19(13):22.
[10] 周 军,王 翎.肺炎克雷伯菌16种β-内酰胺酶基因检测研究[J].世界感染杂志,2007,7(2):113.
Analysis of Antibiotics Resistance of Klebsiella Pneumoniae and Drug Resistance Gene from a Class Three Grade A Hospital
JIA Yan,HE Jin-yu,WANG Shi-bo,LI Ying-li,QIU Jing-fu
(School of Public Health and Management,Chongqing Medical University,Chongqing 400016,China)
OBJECTIVE:To analyze the antibiotic resistance of Klebsiella pneumoniae and distribution of drug resistance gene from a class three grade A hospital in order to investigate the trend of drug resistance change and guide the rational use of antibiotics in the clinic.METHODS:The identification of K.pneumoniae isolates from clinical samples was performed by the VITEK-32 system.The susceptibility test to antimicrobial agents was determined by Disk Diffusion and MIC testing.Detection ofβ-lactamases gene in the ESBLs-producing isolates was performed using PCR.RESULTS:The highest detection rate of K.pneumoniae was found from sputum and throat swab(78.6%).As to K.pneumoniae,the resistance ratio to piperacillin was the highest,while imipenem and meropenem was sensitive to bacterial.The detection rate of ESBLs-producing strains was almost 30%.More than 51%of ESBLs-producing strains carried at least a kind of detectedβ-lactamases genes.Among them,blaTEM gene was the main prevalence gene.CONCLUSION:K.pneumoniae isolates show extensive drug resistance in the hospital with high detection rate of ES
BLs-producing strains,which should get more attention in the clinic.
Klebsiella pneumoniae;Drug resistance;ESBLs;β-lactamases gene
R978.1;R969.3;R378
A
1001-0408(2012)30-2824-03
DOI 10.6039/j.issn.1001-0408.2012.30.16
2012-02-21
2012-03-03)
