实时荧光定量RT-PCR监测沙门氏菌活菌
2012-10-24陈计峦
张 冲,刘 祥,陈计峦
(1.石河子大学食品学院,新疆石河子 832000;
2.国家加工食品质量监督检验中心,天津 300384)
实时荧光定量RT-PCR监测沙门氏菌活菌
张 冲1,刘 祥2,*,陈计峦1
(1.石河子大学食品学院,新疆石河子 832000;
2.国家加工食品质量监督检验中心,天津 300384)
沙门氏菌是引起食物中毒的最常见致病菌,而基于DNA水平的常规PCR(DNA-PCR)检测法虽然快速、灵敏,但在检测时无法区分死活菌,死亡的细菌会出现假阳性结果,严重影响日常检测结果的判断。提取沙门氏菌总RNA之后,采用一步法逆转录聚合酶链式反应(RT-PCR),以沙门氏菌invA mRNA为检测对象,发现只有活的沙门氏菌显阳性,死亡的沙门氏菌呈阴性。而且RT-PCR法的稳定性良好,能够准确定量活的沙门氏菌,在纯培养时,检出限可达1CFU/3mL。实验表明,建立的RT-PCR法是一种能够快速、精确检测沙门氏菌活菌的方法。
沙门氏菌,RT-PCR,死活菌,常规PCR
沙门氏菌是食品中最常见的致病菌,大量存在于牛肉、猪肉、鸡蛋、牛奶、海鲜等多种食物中,能引发多种沙门氏疾病,中、轻度感染会有腹泻、呕吐、腹部绞痛、发烧等症状产生[1]。在欧盟委员会、美、英、法、日等多个国际权威机构和国家发布的食品微生物限量标准中,对肉、蛋、奶类等动物性产品沙门氏菌的规定均为不得检出,在所有微生物限量的标准中限制最为严格[2]。传统培养检测法,操作复杂繁琐、耗时费力,完成整个检测过程至少需要5d的时间[3];而目前在食品微生物检测中广泛运用的定量PCR技术,大多是基于DNA水平的(DNA-PCR),虽然快速、特异性强、灵敏度高,但由于细菌死亡后,DNA降解缓慢,能够稳定存在,容易产生假阳性结果[4]。与DNA相比,mRNA的稳定性较差,在细菌死亡后能够快速降解;而在细菌还具有活性时,就会产生mRNA[5]。研究显示,选择适当的mRNA作为靶点基因,运用RT-PCR技术,便能够只检测到具有活性的细菌,避免死亡细菌假阳性的出现[6];invA mRNA在活的沙门氏菌中大量存在,在LB(Luria-Bertani)培养基中生长时,在其整个生长曲线中均能被检测到;同时,invA mRNA是沙门氏菌入侵哺乳动物活细胞,表达毒理蛋白的必备基因,所有致病性沙门氏菌中均含有invA基因[5]。因此,invA mRNA是非常适宜于RT-PCR检测沙门氏菌的靶点基因[7]。由此,本研究针对沙门氏菌invA基因设计TaqMan探针和引物,采用RT-PCR技术,拟建立一种快速、准确检测沙门氏菌活菌的方法。
1 材料与方法
1.1 材料与仪器
沙门氏菌salmonella(0246项目) 1株,上海之江生物科技;RNAprep pure 培养细胞/细菌总RNA提取试剂盒,北京天根生化公司;PCR反应试剂,Salmonella Real Time PCR Kit 上海之江生物科技; DNase I、RNase-free 美国Fermentas公司;以上试剂
均为分析纯试剂。
Bio-Rad PCR仪 配有 CFX96TMReal-Time System与C1000TMThermal Cycler,美国Bio-Rad公司。
1.2 引物与探针
设计依据:国标 SN/T1870-2007;合成厂家: Invitrogen;菌种名称:沙门氏菌;目标基因:invA;引物序列:YF-SMFY-1:5'-GCGGCGTTGGAGAGTGATA-3'、YF-SMFY-2:5'-AGCAATGGAAAAAGC AGGATG-3';探针序列:YF-SMFY-3:5'-CATTTCTTAAA CGGCGGTGTCTTTCCCT-3'。
1.3 实验方法
1.3.1 沙门氏菌培养 将活化后的菌液50μL接种于3mL LB培养液中,震荡培养3h,200r/min,37℃。
1.3.2 DNA与RNA提取 分别按提取试剂盒说明书进行。
1.3.3 DNase I消化 按DNase I说明书进行。
1.3.4 一步法RT-PCR扩增体系 扩增体系共40μL,其中包括10×Buffer 4μL,MgCl23.2μL,dNTP 0.8μL,YF-SMFY-1 0.5μL,YF-SMFY-2 0.5μL,YFSMFY-3 0.3μL,混合酶1μL,ddH2O 25.7μL,RNA提取液4μL。
扩增参数为:45℃ ×10min;95℃ ×15min;94℃ × 20s,60℃ ×20s,72℃ ×20s,40cycles;单点荧光在72℃;选用FAM通道检测。
1.3.5 DNA-PCR扩增体系 扩增体系共40μL,沙门氏菌荧光定量检测混合液35.6μL,Taq酶0.4μL,DNA提取液4μL。
扩增参数为:94℃ ×2min;93℃ ×15s,60℃ × 60s,40cycles;单点荧光在60℃;选用FAM通道检测。
1.3.6 区分死活菌 吸取两管100μL活化后的沙门氏菌液,一管设为活菌,不做任何处理;另一管设为死菌,采用食品常用的巴氏灭菌方法(65℃水浴30min)灭菌。提取后,采用一步法RT-PCR检测。
同时另取两管相同浓度的死、活菌直接提取DNA,然后采用常规荧光PCR(DNA-PCR)的方法检测。
比较两组扩增结果,若DNA-PCR扩增后活菌与死菌均为阳性,而RT-PCR扩增后活菌结果为阳性,死菌为阴性,则说明能够区分死活菌。
1.3.7 标准曲线制作 取1mL活化后的沙门氏菌,然后按步骤提取RNA,将提取液等比稀释:100、10-1,10-2、10-3、10-4、10-5、10-6,再进行 RT-PCR扩增绘制标准曲线,并将定量结果与传统培养法比对。
1.3.8 重复性 取不同体积的菌液进行RNA抽提,然后采用RT-PCR扩增,观察RT-PCR体系的重复性效果,每个体积重复6次。
1.3.9 检出限 在3mL培养基中分别加入菌量1、10、100CFU,37℃,200r/min,增菌3h后,RT-PCR扩增,观察其扩增曲线。
2 结果与分析
2.1 区分死活菌
活菌与死菌在经过不同方式扩增后,结果见图1。由图1(A)显示,DNA-PCR的扩增曲线死菌活菌均出现峰值;从图1(B)可以看出,经过RT-PCR扩增后,活菌扩增曲线出现峰值,死菌的扩增曲线未出现峰值。数据显示,经RT-PCR扩增后,活菌的Ct值为28.81,呈阳性,死菌未检出,为阴性;而另一组经DNA-PCR扩增后,活菌的 Ct值为15.13,死菌为15.22,两者均呈阳性。由此可以说明,RT-PCR具有区分死、活细菌的能力,能够避免死菌假阳性的出现。
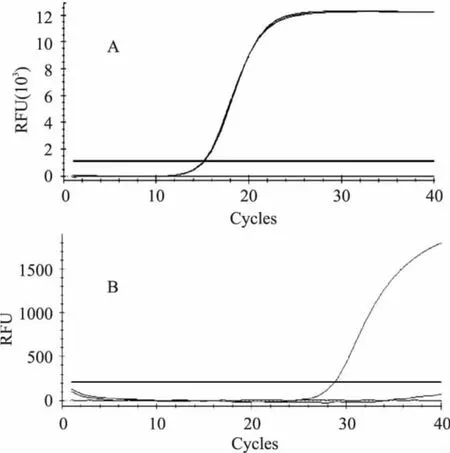
图1 活菌与死菌扩增曲线图Fig.1 Live and dead bacteria amplification curve
2.2 标准曲线绘制
将制备的等比稀释样品,采用本文所建立的实时荧光定量RT-PCR进行扩增,结果浓度在4×107~4×103CFU/mL的样品可以形成较为理想的曲线(图2(A))。以Ct值为纵坐标,对数浓度为横坐标,绘制标准曲线(图2(B))。经验证,RT-PCR与培养法对活菌的定量吻合率为100%。

图2 沙门氏菌标准曲线Fig.2 Standard curve of Salmonella
2.3 重复性
取不同体积的菌液,提取RNA后,经重复性扩增,结果见表1,由此可以看出该方法具有良好的重复性。
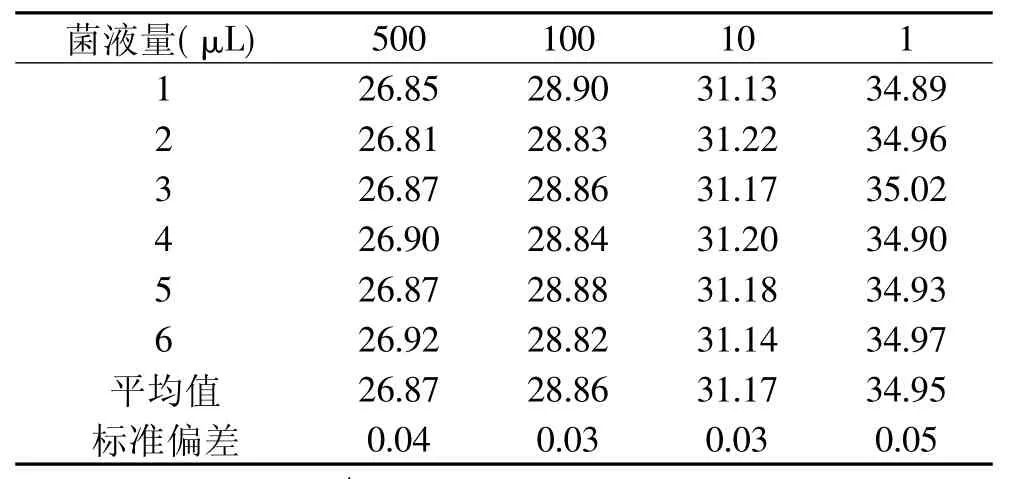
表1 RT-PCR扩增Ct值Table 1 RT-PCR amplification Ct value
2.4 检出限
增菌3h后,1、10、100CFU的沙门氏菌经 RTPCR扩增后,均呈阳性。因此,在纯培养时,增菌3h后的最低检出限为1CFU/3mL。

图3 RT-PCR检出限扩增曲线Fig.3 RT-PCR amplification curves of the detection limit
3 讨论
3.1 RNA的提取
高质量的RNA提取是后续RT-PCR检测的关键步骤[8],RT-PCR对RNA制品的要求极为严格,作为模板的 RNA分子必须是完整的,并且不含有DNA、蛋白质和其他杂质的高质量 RNA。细菌的RNA具有含量少,半衰期短,容易降解等特点,所以与动、植物等真核生物相比较,从细菌中提取、纯化出高质量的RNA具有一定的难度[9-10]。
在不同的生长时期,沙门氏活菌中invA mRNA的含量是不同的。在生长指数中期、指数晚期以及平台期时,invA mRNA的含量分别为 0.4、1、0.004copies/CFU[5]。因此,在提取RNA时,应选择处于生长指数期的沙门氏菌。
生长环境是影响沙门氏菌mRNA表达的重要因素。在实际食品中,存在一些抑制沙门氏菌生长的因素,会使其活性降低,mRNA的数量减少,不利于RT-PCR对活细胞的检测。通过前增菌,不仅可以使沙门氏菌数量增加,而且前增菌也是invA mRNA重新表达的过程[5]。因此,前增菌是RNA提取前必不可少的一步。
3.2 DNase I的使用
由于细菌死亡后,DNA仍然能够稳定存在,且含量远高于靶点mRNA的含量,会严重影响实验结果,所以去除RNA提取液中的残留DNA,是避免死菌假阳性出现的关键。研究表明,现有的DNase I难以一次完全消化掉残留的DNA,但在经过两次DNase I消化后,足以去除所有的残留DNA[8]。但在确认是否有必要再次使用DNase I时,需根据实验结果而定。因为随着消化次数的增加,体系中的RNA也会出现一定的降解。已有研究显示,Ct值相差在3.1~3.6时,同一体系中,其浓度大约有10倍左右的差异。因此,就本实验而言,在经RT-PCR扩增后,活菌与死菌的Ct值相差大于4时,可认为两者的Ct值存在差异,需再次使用DNase I消化提取液[5]。若活菌仍然为阳性,死菌为阴性,则说明区分死活菌实验成功。如图4(A)所示,经DNA-PCR检测,活菌与死菌均为阳性,Ct值分别为15.58、16.00。而消化后经RT-PCR扩增,活菌与死菌仍均为阳性,但活菌的Ct值(22.96)小于死菌的Ct值(35.22),且两者的差值为12.26,见图4(B)。因此,再次使用DNase I消化提取液,经RT-PCR扩增后,结果如图4(C)所示,活菌的Ct值为30.49,死菌未检出,呈阴性。
3.3 灭菌条件
mRNA的半衰期为0.5~50min[6],在细菌死亡后,mRNA也具有一定的稳定性,能够存在一定的时间,但这与灭菌方式以及灭菌后的存储温度有极大的关系[8]。因此,要充分降解死菌体内的mRNA,灭菌方式以及灭菌后的存放温度选择显得尤为重要。有研究指出[4-8,10-11],121℃灭菌15min;100℃灭菌10min,再室温放置50min;80℃灭菌10min,再室温或4℃放置1h;50%的乙醇溶液处理后,室温放置6h或4℃放置72h等均能有效降解细菌体内的靶点mRNA。但这些灭菌方式并不适用于实际食品中沙门氏菌的检测,因为这些方法在灭菌的同时会破坏食品物质的本身结构以及原有风味,所以本实验选用食品中常用的巴氏灭菌方式(65℃水浴30min),这样不仅能充分降解细菌体内的invA mRNA基因[12],而且能够保证食品的原有风味,具有重要的实际意义。

图4 活菌与死菌扩增曲线Fig.4 Live and dead bacteria amplification curve
虽然,目前RT-PCR技术在区分死活细菌上还存在着一定的争议[10,13-14],但是随着研究的深入,RT-PCR技术会日趋完善。目前已有文献表明,RT-PCR法不但能够检测到活性较强的细菌,而且还能够检测到处于活的非培养状态(VBNC)下的细菌,具有较强的区分死、活细菌的能力,其检测结果可作为菌体活性评估的重要参照[15],这是其它快速检测技术以及传统检测技术均无法比拟的优势,因而有着不可估量的发展前景。
[1]Alena Klotchko,MD Fellow,Mark R Wallace,et al. Salmonellosis[R].National Institute of Allergy and Infectious Diseases,2007,27.
[2]Thunberg R L,Tran T T,Bennett R W,et al.Microbial evaluation of selected fresh produce obtained at retail markets[J].Journal of Food Protection,2002,65(4):677-682.
[3]Wallace H Andrews,Thomas Hammack.Bacteriological analytical manual[M].U.S.Food and Drug Administration,Center for Food Safety and Applied Nutrition,April 12,2011.
[4]J T Keer,L Birch.Molecular methods for the assessment of bacterial viability[J].Journal of Microbiological Methods,2003,53(2):175-183.
[5]Narjol Gonzalez-Escalona,Thomas S Hammack,Mindi Russell,et al.Detection of LiveSalmonellasp.Cells in Produce by a TaqMan-Based Quantitative Reverse Transcriptase Real-Time PCR Targeting invA mRNA[J].Applied and Environmental Microbiology,2009,75(11):3714-3720.
[6]Takayama K,S A Kjelleberg.The role of RNA stability during bacterial stress responses and starvation[J].Environ Microbiol,2000,2(4):355-365.
[7]AxelFey,Stefan Eichler,CarlosA Guzmán,etal. Establishment of a real-time PCR-based approach for accurate quantification of bacterial RNA targets in water,using Salmonella as a model organism[J].Appl Environ Microbiol,2004,70(6): 3618-3623.
[8]Nancy Rijpens,Geert Jannes,Lieve Herman.Messenger RNA-based RT-PCR detection of viable Salmonella[J].International Dairy Journal,2002,12(2):233-238.
[9]Gingeras T R,Rosenow C.Studying microbial genomes with high-density oligonu-cleotide arrays[J].Am Soc Microbial News,2000,66(8):463-469.
[10]Junhui L,Chuanhong T.A simple,rapid and effective method for total RNA extraction from Lentinula edodes[J].Biotechnol Lett,2006,28:1193-1197.
[11]Lydia V Rump,Benedicta Asamoah,Narjol Gonzalez-Escalona.Comparison of commercial RNA extraction kits for preparation of DNA-free total RNA from Salmonella cells[J]. BMC Research Notes,2010,3(1):211.
[12]Carsten Suhr Jacobsen,William E Holben.Quantification of mRNA in Salmonella sp.seeded soil and chicken manure using magnetic capture hybridization RT-PCR[J].Journal of Microbiological Methods,2007,69(2):315-321.
[13]Fleige S,Pfaffl M W.RNA integrity and the effect on the real-time qRT-PCR performance[J].Mol Aspects Med,2006,27 (2):126-139.
[14]Schroeder A,Odilo Mueller,Susanne Stocker,et al.The RIN: An RNA integrity number for assigning integrity values to RNA measurements[J].BMC Mol Biol,2006,7(1):3.
[15]Matsuda K,Tsuji H,Asahara T,et al.Sensitive quantitative detection of commensal bacteria by rRNA-targeted reverse transcription-PCR[J].Appl Environ Microbiol,2007,73 (1):37-39.
Real-time quantitative reverse transcription polymerase chain reaction detection of live Salmonella
ZHANG Chong1,LIU Xiang2,*,CHEN Ji-luan1
(1.College of Food Science and Engineering,Shihezi University,Shihezi 832000,China;
2.National Quality Supervision and Inspection Center of Processed Foods,Tianjin 300384,China)
Salmonella was the most common pathogens that cause food poisoning.The conventional DNA-based PCR(DNA-PCR)detection method was rapid and sensitive,but dead bacteria could not be distinguished from the viable in the detection method,and the death may cause false-positive results.That could seriously affect the daily test results.Total RNA was extracted from Salmonella,by applying one-step reverse transcription polymerase chain reaction(RT-PCR),to detect Salmonella invA mRNA,only live Salmonella was found positive and death was negative.And the RT-PCR had good stability,it could quickly and accurately quantify the live Salmonella,the detection limit was 1CFU/3mL in pure culture.Experiments showed that the RT-PCR was a fast,accurate method to detect live Salmonella.
Salmonella;RT-PCR;dead and live bacteria;conventional PCR
TS207.3
A
1002-0306(2012)06-0091-04
2011-05-23 *通讯联系人
张冲(1988-),男,硕士研究生,主要从事食品科学与分子生物学研究。
国家质检总局科技项目(2010QK307),天津市质监局科技项目(10-11)。
