鲤45S rDNA的染色体荧光原位杂交定位
2012-09-19赵紫霞邓海霞徐鹏张研李炯棠孙效文
赵紫霞,邓海霞,徐鹏,张研,李炯棠,孙效文
(1.中国水产科学研究院生物技术研究中心,北京100141;2.大连海洋大学水产与生命学院,辽宁大连116023)
鲤Cyprinus carpio是世界上养殖范围较广的经济鱼类,在水产养殖业中占据着非常重要的地位。近年来,随着鲤基因组学和功能基因组学研究的逐步深入[1],已测定了大量的功能基因序列、分子标记序列、表达序列标签等数据,并构建了遗传连锁图谱[2]、细菌人工染色体物理图谱[3]等,然而这些基因组序列在鲤染色体上的定位信息却少见报道。目前,仅有5S核糖体DNA(ribosomal DNA,rDNA)[4]、重复序列 CR1[5]和生长激素转基因片段[6]等得到定位。
45S rDNA编码的3种核糖体RNA参与蛋白质的合成,是细胞内表达量最为丰富的基因[7],也是基因组内典型的中度串联重复序列,每个重复单元由18S rDNA、内转录间隔区1(internal transcribed spacer-1,ITS-1)、5.8S rDNA、内转录间隔区2(internal transcribed spacer-2,ITS-2)和28S rDNA组成,重复单元之间被非转录基因间隔区 (Intergenicspacer, IGS) 所 分 隔[8-9]。45S rDNA存在于所有真核生物中,拷贝数通常为数百个,不同物种间45S rDNA的染色体分布具有较大差异,因而其常被作为物种 (或品种)重要的遗传学特征进行研究。目前,有多个水产物种的45S rDNA定位信息已有报道,如栉孔扇贝Chlamys farreri[10]、泥鳅 Misgurnus anguillicaudatus[11]、多瑙哲罗鲑Hucho hucho[12]等。本研究中,作者通过荧光原位杂交(Fluorescence in situ hybridization,FISH)技术,对鲤45S rDNA序列进行染色体定位。
1 材料与方法
1.1 材料
试验用鱼采自中国水产科学研究院黑龙江水产研究所松浦实验基地,包括1龄散鳞镜鲤Cyprinus carpio var.scattered mirror 5尾,1龄松浦鲤Cyprinus carpio Songpu 5尾,体长为14.7~19.2 cm,体质量为42~106 g。
1.2 方法
1.2.1 有丝分裂中期染色体的制备 试验鱼在25℃恒温水族箱内暂养48 h后,腹腔注射植物血凝素(上海世泽生物科技有限公司),剂量为60 μg/g(鱼体质量)。18 h后腹腔注射秋水仙素(Sigma-Aldrich),剂量为6 μg/g(鱼体质量)。2 h后剪尾鳍放血,取肾组织,采用空气干燥法[13]制备有丝分裂中期染色体,置于冰箱(-80℃)中保存。
1.2.2 鲤45S rDNA序列的获取与验证 使用CLC Genomics Workbench 4.03数据分析平台,依据人45S rDNA内的相对保守区域18S rDNA(NCBI Reference Sequence:NR_003286.2,下同)、5.8S rDNA(NR_003285.2)、28S rDNA(NR_003287.2),在本课题组鲤基因组序列草图数据库中采用Blast软件进行序列同源性比对,对于命中的序列群进行必要的重新拼接和筛选,获得鲤45S rDNA完整序列。在该序列上设计引物 (由上海生工生物工程技术服务有限公司),分别以散鳞镜鲤和松浦鲤基因组DNA为模板进行PCR扩增,对部分扩增产物进行TA克隆,委托中美泰和生物技术 (北京)有限公司测序验证序列的准确性。
1.2.3 染色体荧光原位的杂交 分别使用引物CAAG1191、CAAG3602和 CAAG1845,采用 PCR标记探针合成试剂盒 (Roche公司产品)制备生物素标记探针和地高辛标记探针,杂交液成分为:5 ng/μL(探 针),15 ng/μL 鲑 精 DNA(Sigma-Aldrich),15 ng/μL 大肠杆菌 tRNA(Roche),2 μg/μL小牛血清白蛋白(上海生工生物工程技术服务有限公司),10%硫酸葡聚糖(Amresco)和4×SSC。按照文献 [11]中的方法进行杂交反应。
1.2.4 荧光染色及信号的放大 对于用生物素标记的探针杂交片,依次加入用绿色荧光染料FITC标记的亲和素 (Sigma-Aldrich)、用生物素标记的亲和素抗体 (Vector)、用FITC标记的亲和素进行反应;对于用地高辛标记的探针杂交片,依次加入鼠抗地高辛抗体 (Sigma-Aldrich)、用红色荧光染料TRITC标记的兔抗鼠抗体 (Sigma-Aldrich)、用TRITC标记的羊抗兔抗体 (Sigma-Aldrich)进行反应,使用各产品说明书推荐剂量。每次在37℃下避光反应1 h,反应结束后按文献 [10]中的方法洗去未结合的试剂。
1.2.5 杂交信号的荧光观察 每张载玻片上滴加50 μL 含有5 ng/μL DAPI、20 mg/mL DABCO、100 mmol/L Tris(pH 8.0)、90%甘油的荧光染色剂,室温下放置3 min后盖片,用甲油封片,在4℃下染色8 h后,在Imager M2荧光显微镜 (Zeiss)下观察并拍照。
2 结果
2.1 45S rDNA序列及其验证
获得的序列全长9 426 bp,其中包含45S rDNA完整序列和非转录IGS的部分序列,其功能注释见表1,序列信息已提交 GenBank,注册号为JN628435。
由于该序列是从鲤全基因组高通量二代测序的初步组装结果中获取,有可能存在拼接错误,因此在该序列不同位置处设计引物,分别以散鳞镜鲤和松浦鲤基因组DNA为模板进行PCR扩增,验证序列信息的准确性。设计的13对引物基本均匀覆盖该序列的大部分区域,扩增产物电泳结果均呈现清晰单带 (图1),实际扩增长度与预期相符,证明该序列组装结果较为可靠。

表1 鲤基因组序列JN628435功能注释Tab.1 Functional annotation of common carp genome sequence JN628435
为进一步验证序列的准确性,在以散鳞镜鲤基因组DNA为模板的PCR扩增产物中选取10条进行TA克隆,挑取单克隆进行Sanger测序,测序结果与组装序列的一致性均在95%以上,可转录区的一致性为99%或100%。扩增引物详细信息和目标片段测序结果见表2。
2.2 散鳞镜鲤和松浦鲤有丝分裂中期染色体组型
选取形态清晰、染色体分散良好的中期分裂相进行染色体计数,结果显示两个品种鲤的染色体二倍数均为2n=100。散鳞镜鲤共计数52个分裂相,其中37个分裂相染色体数目为100,其典型形态如图2-A所示,多数染色体数目完整的分裂相核型公式符合张克俭等[14]报道的公式20m+26sm+30st+24t;松浦鲤共计数46个分裂相,其中38个分裂相染色体数目为100,其典型形态如图2-B所示,多数染色体数目完整的分裂相核型公式符合尹洪滨[15]报道的公式30m+26sm+26st+18t。
2.3 标记探针的制备

图1 用于验证序列准确性的引物PCR扩增结果Fig.1 PCR amplification of the primers used in sequence accuracy verification

表2 用于验证序列准确性的引物信息及测序结果Tab.2 Primer information and sequencing of the primers used in sequence accuracy verification
选择位于JN628435序列上不同区域的3对引物,以PCR方法制备用于杂交反应的标记探针,其中探针CAAG1191和CAAG3602使用生物素标记,探针CAAG1845使用地高辛标记。探针的琼脂糖凝胶电泳结果见图3,与未进行标记的普通PCR扩增产物相比,标记后探针的电泳条带有微弱滞后,表明其标记成功,分子量增加,泳动速度下降。
2.4 通过单色FISH进行探针的染色体定位
使用绿色荧光染料FITC对生物素标记进行显色和信号放大,显微镜照片显示,5.8S rDNA探针CAAG1191和IGS探针CAAG3602(图4)在散鳞镜鲤和松浦鲤中的定位情况相同,仅定位于一对近端着丝粒染色体的短臂末端。
2.5 通过双色FISH进行探针的染色体共定位
将用地高辛标记的18S rDNA探针CAAG1845与用生物素标记的5.8S rDNA探针CAAG1191和IGS探针CAAG3602在染色体上共定位,使用红色荧光染料TRITC对用地高辛标记的进行显色和信号放大,使用绿色荧光染料FITC对用生物素标记的进行显色和信号放大,显微镜照片 (图5)显示,位于45S rDNA不同区域的3条探针在散鳞镜鲤和松浦鲤中均定位于同一对近端着丝粒染色体的短臂末端。图5-A、C显示探针CAAG1845的单独定位结果 (红色),图5-B、D显示3条探针共定位的结果,其中黄色信号由红色的TRITC和绿色的FITC混合而成。
3 讨论
3.1 45S rDNA探针的设计
国内报道的核糖体DNA定位研究多使用包含部分目的片段的质粒克隆作探针[8,11],通过不同实验室之间相互馈赠而获得。由于探针获取途径受限,一定程度上影响了FISH技术的推广。本研究中利用“鲤鱼基因组计划”的基因组测序组装结果,获得了鲤45S rDNA全长序列,依据该序列设计引物,通过PCR方法制备杂交探针,不同实验室之间只需交流引物信息即可共享探针,有效提高了FISH技术的通用性。
曾有部分物种rDNA旁系同源现象存在的报道[16],即在同一物种基因组的不同区域,存在具有同性源但不完全相同的rDNA序列,有的能够转录[17-19],有的则彻底失去功能成为假基因[16,20-24]。虽然在鲤基因组序列草图数据库中进行Blast比对的过程中尚未发现45S rDNA旁系同源序列,但为了确保覆盖可能存在的旁系同源序列,本研究中设计制备位于45S rDNA不同区域的多条探针分别用于FISH定位。
探针 CAAG1191长度为 116 bp,位于 5.8S rDNA上,是一段物种间高度保守的序列,其引物序列与人类5.8S rDNA(NR_003285.2)仅相差1个碱基,与斑马鱼 (CT583728.24)、岩原鲤(DQ994157.1)、四川白甲鱼 (DQ994153.1)、重口裂腹鱼 (DQ994148.1)等多种鲤科鱼类的同源序列则完全相同,该探针可以用作鲤科鱼类的通用探针。
探针CAAG1845长度为1 052 bp,位于18S rDNA上,序列相对保守,其引物序列与斑马鱼(BX296557.35)、银鲫 (EF189737.1)两个物种能够通用。
探针CAAG3602长度为574 bp,位于非转录IGS区域,是一段保守程度较低的序列,在NCBI中进行Blast比对,未在其他物种中检索到同源序列。与另外两条探针相比,探针CAAG3602为非转录序列,不会受到未被RNA酶降解的残余核糖体RNA的干扰,因而能够降低假阳性检出率,显微镜下观察时视野也较为洁净,易于杂交信号的辨识。
3.2 45S rDNA的染色体定位
多数鲤科鱼类的染色体二倍数为2n=50[13,25],这是鲤科鱼类的原始核型和进化基点。而鲤具有100条染色体 (2n=100),对其染色体数目和DNA含量的分析都显示,鲤是鲤科鱼类原始类群在进化过程中经历了全基因组复制事件而形成的四倍体物种[26-28]。David等[29]通过对鲤基因组非编码区59个微卫星位点的分析,证实其中有约60%的位点存在复制现象,并推测鲤为通过异种杂交实现基因组复制的异源四倍体,其基因组四倍化发生时间大约距今1 200万年前。而对基因组编码序列的研究表明,与同属于鲤科的模式生物斑马鱼 (2n=50)相比,鲤大多数功能基因都存在比斑马鱼多一倍的旁系同源序列[27,29-33],这些同源序列正是起源于基因组加倍过程中,来源于两套染色体并行使同一功能的基因组序列。
本研究中3条位于45S rDNA序列不同区域的探针在两品种鲤染色体上的定位结果均一致,仅能定位于一对同源染色体的唯一位点。作为生命活动不可或缺的重要基因,45S rDNA这一定位结果在功能基因分布整体且呈现四倍化特点的鲤基因组内是非常显著的特例,暗示着鲤基因组在经历了染色体整倍化复制事件后,又呈现出重新二倍化的现象。

图2 DAPI染色后的散鳞镜鲤和松浦鲤有丝分裂中期染色体显微图Fig.2 Chromosome images of mitotic metaphase in scattered mirror carp and Songpu carp,stained with DAPI
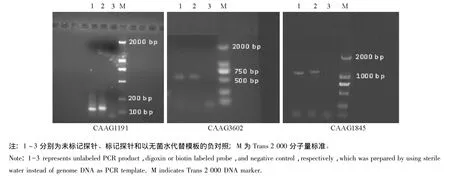
图3 标记探针的电泳图Fig.3 Electrophoresis images of labeled probes

图4 探针CAAG1191(A、B)和CAAG3602(C、D)分别在散鳞镜鲤和松浦鲤染色体上的FISH定位Fig.4 FISH localization of probe CAAG1191(A,B)and CAAG3602(C,D)on chromosomes of scattered mirror carp and Songpu carp,respectively

图5 探针CAAG1845(红色)、CAAG1191(绿色)、CAAG3602(绿色)在散鳞镜鲤和松浦鲤染色体上的FISH共定位Fig.5 FISH co-localization of probe CAAG1845(red),CAAG1191(green),and CAAG3602(green)on chromosomes of scattered mirror carp and Songpu carp
通过生物信息学比对,在鲤基因组序列草图数据库中未发现45S rDNA旁系同源序列。在45S rDNA序列的不同位置上设计探针进行FISH定位,各条探针的定位结果也均一致,表明45S rDNA位点的重新二倍化并不是由旁系同源序列的逐渐分化所带来的,而更可能是因染色体水平的断裂重排所导致。
3.3 45S rDNA作为染色体特异性标识的意义
与其他物种相比较,鲤染色体呈现出数目众多、形态较为微小的特征。以人类为例,基因组大小为3.0 Gb,具有46条染色体,有经验的观察者在光学显微镜下即可逐一辨认各条染色体,并判断某些染色体异常及其与表型性状的关系。而鲤基因组大小约为1.7 Gb[1],分散成100条染色体,每条染色体长度都较短,同组的各条染色体之间差异更小,彼此之间很难凭借光学显微镜下的形态特征进行准确分辨,这给以染色体遗传规律为研究对象的细胞遗传学研究带来了很大困难。因此,寻找染色体特异性标识探针在鲤细胞遗传学研究中具有重要意义,它能够使基因组序列与细胞学观察联系起来,实现染色体的准确识别。
45S rDNA序列则符合这种需求,它在鲤基因组中仅定位于一对同源染色体上,可以用作该染色体的特异性标识,并将其与已有遗传连锁图谱中特定的连锁群相对应。孙效文等[2]报道了鲤的第一个遗传连锁图谱,45S rDNA位于该图谱1号连锁群上,连锁群长度为227 cM,包含15个标记。本研究中所报道的3条探针均可用作1号连锁群的特异性标记,可应用于鲤细胞遗传学图谱的构建、鲤科鱼类比较基因组学的分析以及染色体结构变异与性状关联分析等的研究。
[1]Xu P,Li J,Li Y,et al.Genomic insight into the common carp(Cyprinus carpio)genome by sequencing analysis of BAC-end sequences[J].BMC Genomics,2011,12:188.
[2]Sun X,Liang L.A genetic linkage map of common carp(Cyprinus carpio L.)and mapping of a locus associated with cold tolerance[J].Aquaculture,2004,238(1/4):165-172.
[3]Xu P,Wang J,Wang J,et al.Generation of the first BAC-based physical map of the common carp genome[J].BMC Genomics,2011,12:537.
[4]Inafuku J,Nabeyama M,Kikuma Y,et al.Chromosomal location and nucleotide sequences of 5S ribosomal DNA of two cyprinid species(Osteichthyes,Pisces)[J].Chromosome Res,2000,8(3):193-199.
[5]王泽群,朱作言.鲤鱼基因组重复顺序CR1的染色体原位杂交定位[J].科学通报,1999,44(20):2182-2186.
[6]耿波,梁利群,孙效文,等.应用荧光原位杂交技术检测大麻哈鱼生长激素基因在超级鲤染色体上的插入位点[J].农业生物技术学报,2005,13(1):127-128.
[7]Mallatt J,Winchell C J.Ribosomal RNA genes and deuterostome phylogeny revisited:more cyclostomes,elasmobranchs,reptiles,and a brittle star[J].Mol Phylogenet Evol,2007,43(3):1005-1022.
[8]龚志云,吴信淦,程祝宽,等.水稻45S rDNA和5S rDNA的染色体定位研究[J].遗传学报,2002,29(3):241-244.
[9]Pisano E,Ghigliotti L.Ribosomal genes in notothenioid fishes:Focus on the chromosomal organisation[J].Mar Genom,2009,2(1):75-80.
[10]郇聘.利用荧光原位杂交技术(FISH)对中国明对虾和栉孔扇贝若干重要基因定位的研究[D].青岛:中国科学院海洋研究所,2009.
[11]贾光风,李雅娟,钱聪,等.大鳞副泥鳅(2n♀)×泥鳅(4n♂)杂种后代染色体带型及FISH分析[J].东北农业大学学报,2011,42(9):72-79.
[12]Ocalewicz K,Woznicki P,Jankun M.Mapping of rRNA genes and telomeric sequences in Danube salmon(Hucho hucho)chromosomes using primed in situ labeling technique(PRINS)[J].Genetica,2008,134(2):199-203.
[13]余先觉.中国淡水鱼类染色体[M].北京:科学出版社,1989.
[14]张克俭,高健,张景龙,等.杂交鲫(白鲫♀×散鳞镜鲤♂)及其双亲染色体组型的研究[J].遗传学报,1995,19(4):305-309.
[15]尹洪滨.四种鲤鱼染色体核型比较研究[J].水产学杂志,2001,14(1):7-10.
[16]Xu J,Zhang Q,Xu X,et al.Intragenomic variability and pseudogenes of ribosomal DNA in stone flounder Kareius bicoloratus[J].Mol Phylogenet Evol,2009,52(1):157-166.
[17]Copenhaver G P,Pikaard C S.Two-dimensional RFLP analyses reveal megabase-sized clusters of rRNA gene variants in Arabidopsis thaliana,suggesting local spreading of variants as the mode for gene homogenization during concerted evolution[J].Plant J,1996,9(2):273-282.
[18]Reed K M,Hackett J D,Phillips R B.Comparative analysis of intraindividual and inter-species DNA sequence variation in salmonid ribosomal DNA cistrons[J].Gene,2000,249(1-2):115-125.
[19]Harpke D,Peterson A.Non-concerted ITS evolution in Mammillaria(Cactaceae)[J].Mol Phylogenet Evol,2006,41(3):579-593.
[20]Childs G,Maxson R,Cohn R H,et al.Orphons:dispersed genetic elements derived from tandem repetitive genes of eukaryotes[J].Cell,1981,23(3):651-663.
[21]Hughes C E,Bailey C D,Harris S A.Divergent and reticulate species relationships in Leucaena(Fabaceae)inferred from multiple data sources:Insights into polyploid origins and nr DNA polymorphism[J].Am J Bot,2002,89(7):1057-1073.
[22]Márquez L M,Miller D J,Mackenzie J B,et al.Pseudogenes contribute to extreme diversity of nuclear ribosomal DNA in the hard coral Acropora[J].Mol Biol Evol,2003,20(7):1077-1086.
[23]Ruggiero M V,Procaccini G.The rDNA ITS region in the Lessepsian marine angiosperm Halophila stipulacea(Forssk.)Aschers.(Hydrocharitaceae):intragenomic variability and putative pseudogenic sequences[J].J Mol Evol,2004,58(1):115-121.
[24]Keller I,Chintauan-Marquier I C,Veltsos P,et al.Ribosomal DNA in the grasshopper Podisma pedestris:escape from concerted evolution[J].Genetics,2006,174(2):863-874.
[25]Arai R.A chromosome study on two cyprinid fishes,Acrossocheilus labiatus and Pseudorasbora pumila pumila,with notes on Eurasian Cyprinids and their karyotypes[J].Bull Natn Sci Mus Tokyo Ser A,1982,8(3):131-152.
[26]Ohno S,Muramoto J,Christian,et al.Diploid-tetraploid relationship among Old World members of the fish family Cyprinidae[J].Chromosoma(Berl.),1967,23(1):1-9.
[27]Zhang H,Okamoto N,Ikeda Y.Two c-myc genes from a tetraploid fish,the common carp(Cyprinus carpio)[J].Gene,1995,153(2):231-236.
[28]Larhammar D,Risinger C.Molecular genetic aspects of tetraploidy in the common carp Cyprinus carpio[J].Mol Phylogenet Evol,1994,3(1):59-68.
[29]David L,Shula B,Marcus W F,et al.Recent duplication of the common carp(Cyprinus carpio L.):Genome as revealed by analyses of microsatellite loci[J].Mol Biol Evol,2003,20(9):1425-1434.
[30]McGuigan K,Phillips P C,Postlethwait J H.Evolution of sarcomeric myosin heavy chain genes:evidence from fish[J].Mol Biol Evol,2004,21(6):1042-1056.
[31]Kongchum P,Hallerman E M,Hulata G,et al.Molecular cloning,characterization and expression analysis of TLR9,MyD88 and TRAF6 genes in common carp(Cyprinus carpio)[J].Fish &Shellfish Immun,2011,30(1):361-371.
[32]Fraser J,de Mello L V,Ward D,et al.Hypoxia-inducible myoglobin expression in nonmuscle tissues[J].Proc Natl Acad Sci USA,2006,103(8):2977-2981.
[33]Evans H,De Tomaso T,Quail M,et al.Ancient and modern duplication events and the evolution of stearoyl-CoA desaturases in teleost fishes[J].Physiol Genomics,2008,35(1):18-29.
