浑球红细菌Rhodobacter sphaeroides LHS-305腈水解酶基因的克隆及表达
2012-09-18王学东李桂南李明阳
王学东,李桂南,李明阳
(华东理工大学a.生物反应器工程国家重点实验室;b.鲁华生物技术研究所,上海 200237)
腈类化合物是一类非常重要的化合物。腈类物质的水解反应在氨基酸、酰胺、羧酸以及它们的衍生物的合成中有着非常广泛的应用[1-4]。
对腈类物质水解的方法有化学水解法和酶法2种。化学水解法一般需要满足强酸(或强碱)、高温、高压等比较剧烈的反应条件,效率低、成本高,同时也造成了相当的环境压力。而酶法水解则具有反应条件温和、高效、专一、成本低、环境污染小等优点[5-7]。
由于天然腈类化合物的存在,许多微生物都有转化腈类物质的能力。从20世纪60年代开始,人们从假单孢菌(Pseudomonas)、红球菌(Rhodococcus)、产碱杆菌(Alcaligenes)、克雷伯氏菌(Klebsiella)中发现并得到了腈水解酶[8]。近年来,随着宏基因组技术的快速发展,人们从自然环境中获得了大量未培养微生物的基因组,其中已测序的腈水解酶基因也大量出现,这就为腈水解酶的进一步研究提供了更多材料。
利用基因重组技术,构建具有腈水解酶活性的新型基因工程菌株,可以在不同程度上解决生产中遇到的许多问题,如提高酶的表达水平、增强酶的热稳定性、减少副反应的发生等。1987年,Stalker等[8]首次在克雷伯氏菌中克隆得到腈水解酶的基因,并在大肠杆菌中进行了表达。近年来,国外对重组腈水解酶的研究日益增多,但国内相关报道却比较少。国内王清路等[9]研究了腈水解酶基因在毕赤酵母中的表达,罗晖等[10]和刘俊峰等[11]分别研究了腈水解酶基因在大肠杆菌中的表达情况。
本研究以实验室自有腈水解酶产生菌为基础,通过简并PCR和染色体步移的方法得到该菌株的腈水解酶基因,然后进行了异源表达,为进一步在分子水平上对该腈水解酶进行研究奠定了基础。
1 材料与方法
1.1 材料
浑球红细菌Rhodobacter sphaeroides LHS-305由华东理工大学生物反应器工程国家重点实验室从土壤中筛选并保存,现保藏于武汉中国典型培养物保藏中心,保藏编号为CCTCC M209065。大肠杆菌(Escherichia coli)DH5α、Rosetta(DE3)为华东理工大学生物反应器工程国家重点实验室保存的菌株。限制性内切酶、连接酶、Taq DNA聚合酶、LA-Taq DNA聚合酶、PrimeSTAR®HS DNA 聚合酶等购自Takara公司。细菌基因组DNA提取试剂盒、质粒提取试剂盒、琼脂糖凝胶DNA回收试剂盒购自天根生物科技有限公司。引物由上海英骏公司合成。
1.2 腈水解酶部分基因序列片段的PCR扩增
1.2.1 简并引物的设计
从NCBI的Genebank数据库中搜索腈水解酶基因的相关蛋白质序列,并对其进行序列比对分析,利用CODEHOP设计简并引物,最终基于氨基酸保守区G(A/Q)L(C/A)CWEH设计出正向简并引物NIT-CWEH:5'-GGC GCC CTG TGC TGY KBI GAR CA-3'(简并度:96,Tm 值:63.9℃),基于氨基酸保守区G(H/N)YARP(D/E)(V/L)设计出反向简并引物NIT-ARPDER:5'-CGT CGG GCC GGG CRT ART KIC C-3'(简并度:32,Tm 值:61.3℃)。
1.2.2 腈水解酶基因保守区域的扩增
以Rhodobacter sphaeroides LHS-305基因组DNA为模板,利用根据腈水解酶基因保守区设计的引物NIT-VFPE和NIT-FDT以及NIT-CWEH和NIT-ARPDER进行PCR扩增。PCR反应使用Touch-Down PCR方法:94℃预变性5 min;94℃变性30 s,退火是从65℃下降到50℃,时间为1 min,每个循环下降0.5℃,72℃延伸30 s,共30个循环;然后在94℃变性30 s,50℃退火1 min,72℃延伸30 s,共15个循环;最后72℃延伸10 min。
1.3 染色体步移得到保守区域上下游序列
参照文献[12]设计2个随机简并引物AD1和 AD2,根据上一步得到的 Rhodobacter sphaeroides LHS-305腈水解酶基因的部分片段设计3个正向的嵌套步移引物(分别为SP1、SP2、SP3)和3个反向的嵌套步移引物(分别为 SP4、SP5、SP6),见表1。

表1 染色体步移使用的引物
用引物SP1/SP4和AD1/AD2按照表2中的PCR反应条件进行第1轮PCR扩增,将其产物稀释10倍作为模板。使用引物SP2/SP5和AD1/AD2进行第2轮巢式PCR扩增,再将其产物稀释10倍作为模板。使用引物SP3/SP6和AD1/AD2进行第3轮巢式PCR扩增。将第3轮巢式PCR扩增产物切胶回收,连接到pMD19-T载体,转化至E.coli DH5α感受态细胞中,验证为阳性克隆后测序。对克隆得到的序列进行拼接,得到完整腈水解酶基因序列。
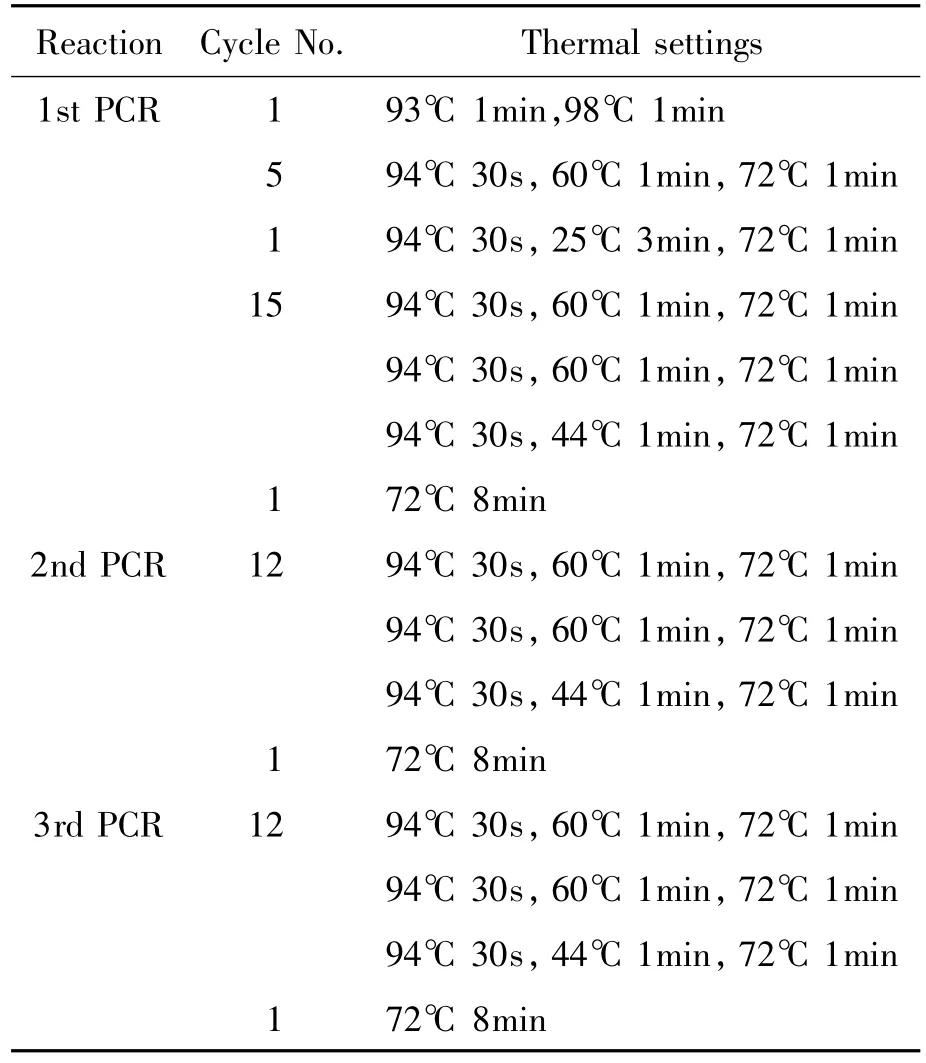
表2 染色体步移的PCR反应条件
1.4 腈水解酶基因全长的克隆和简单序列分析
根据拼接得到的腈水解酶基因全长,设计上游引物NIT-F:5'-GGAATTCCATATGCCCAAGACAGTACGTGC-3'(下划线部分为NdeI酶切位点)和下游引物NIT-R:5'-AACAAGCTTTCACGCATCCTGGGCCTC-3'(下划线部分为HindⅢ酶切位点)用于完整开放阅读框的扩增。PCR反应使用具有高保真扩增效果的PrimeSTAR®HS DNA聚合酶,反应条件为:94℃预变性5 min;94℃变性30 s,60℃退火30 s,72℃延伸1 min,共30个循环;最后72℃继续延伸10 min。将获得的全长腈水解酶基因提交Genebank数据库。
将测序得到的核苷酸序列翻译成蛋白质序列,利用NCBI数据库中的Blast程序对该蛋白质序列进行比对分析。
1.5 腈水解酶基因的表达
1.5.1 重组质粒的构建及转化
将PCR产物切胶回收后用NdeI和HindⅢ双酶切,连接到同样经过双酶切的载体pET-28a(+)上,构建重组表达载体pET28a-nit,将连接产物转化至大肠杆菌DH5α中,提取质粒DNA,用 NdeI和HindⅢ双酶切验证筛选得到阳性克隆。提取验证正确的质粒转化Rosetta(DE3),得到腈水解酶的基因工程菌株。
1.5.2 腈水解酶的诱导表达
将构建好的重组菌Rosetta/pET28a-nit和对照菌Rosetta/pET28a分别接种于5 mL含有50 μg/mL卡那霉素、34 μg/mL氯霉素的LB液体培养基中,37℃、200 r/min培养过夜,1%接种量转接至新鲜LB 液体培养基中,37℃培养至OD600为0.4 ~0.6 时加 入 IPTG(终浓度为0.2 mmol/L),20℃、200 r/min继续培养20 h左右,离心收集菌体。用10 mM磷酸钠缓冲液(pH值为7.0)清洗数次,取0.5g湿菌体催化20 mM 3-氰基吡啶反应30 min,将反应液15 kg离心10 min,取上清,用HPLC方法检测重组菌活力。检测条件:柱子为 Agilent Zorbax SB-Aq,流动相为甲醇/0.1%磷酸=20/80,流速为1 mL/min,检测波长为230 nm。
2 结果与讨论
2.1 腈水解酶基因保守区域的获得
以提取的Rhodobacter sphaeroides LHS-305基因组DNA为模板,利用引物 NIT-VFPE和NITFDT,经过PCR扩增后没有得到预期大小的目的条带。
而利用引物 NIT-CWEH和 NIT-ARPDER,经过PCR扩增后则得到了目的大小的部分腈水解酶基因的序列,保守序列的琼脂糖电泳分析如图1所示。回收400 bp左右的目的大小片段,并将其克隆至载体pMD19-T Simple中,测序结果显示PCR产物大小为402 bp。BLAST比对分析表明:该片段与库中已知的腈水解酶核苷酸序列最大相似性为83%。

图1 保守序列的琼脂糖电泳分析
2.2 保守区域上下游序列的获得
以 SP1/SP4、SP2/SP5、SP3/SP6为特异性引物,AD1、AD2为随机简并引物,利用巢式PCR方法,经过3轮PCR反应,分别克隆得到上述已知片段的上下游序列,结果如图2所示。
2.3 腈水解酶全长基因的获得和序列分析
将Rhodobacter sphaeroides LHS-305腈水解酶基因部分片段及上下游序列拼接,得到了该菌株完整的腈水解酶基因序列,长度为969 bp。将该基因序列提交Genebank数据库,序列的登录号为JN635494,推测其编码的蛋白质为322 aa,理论分子量为35426 Da,理论等电点为6.01。
将该腈水解酶基因与NCBI数据库中已知基因序列进行比对,发现Rhodobacter sp.LHS-305腈水解酶与Agrobacterium sp.H13-3(Genebank登录号:ADY67983.1)有最高的核苷酸序列相似性,为82%。此外,与同属中其他腈水解酶基因序列的相似性很低(最高只有38.4%)。
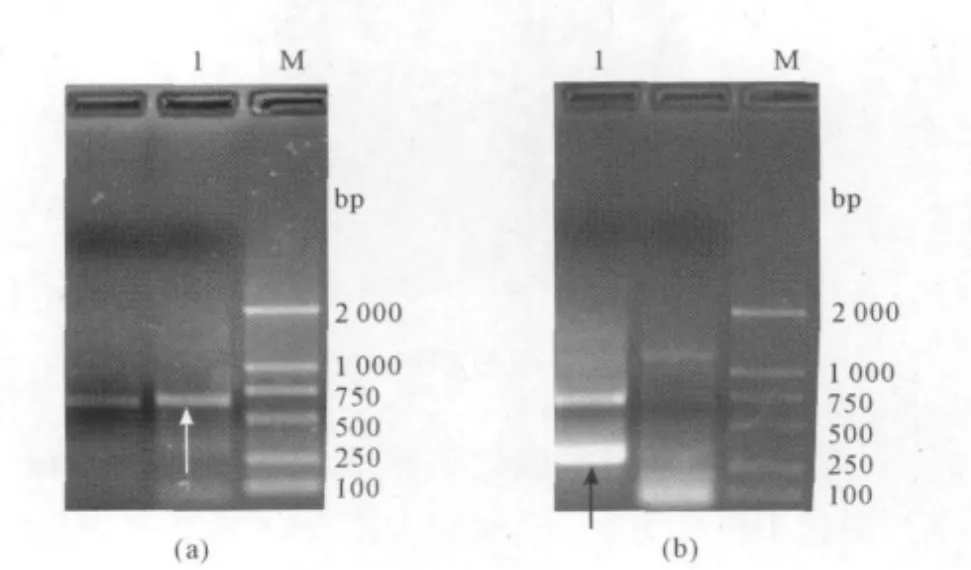
图2 保守区域上游(a)和下游(b)序列的琼脂糖电泳分析
将DNA序列翻译成对应的氨基酸序列,比对结果如图3所示。发现该腈水解酶与已经报道的Synechocystis sp.PCC 6803 的同源性为 60.2%,与Pseudomonas fluorescens Pf-5的同源性为30.7%,与同属中其他腈水解酶蛋白质序列相似性最高只有17.2%,这一结果也很好地解释了根据同菌属腈水解酶保守序列设计的简并引物没能克隆得到目的片段的原因。

图3 Rhodobacter sphaeroides LHS-305腈水解酶基因片段的氨基酸序列比对结果
2.4 腈水解酶基因的诱导表达
将构建好的重组菌株Rosetta/pET28a-nit按本文的方法诱导表达,对得到的菌液超声破碎后,取上清通过15%SDS-PAGE检测目的蛋白的表达情况,结果如图4所示。图4中可在40 kD附近看到1条清晰的蛋白表达条带,与预期的腈水解酶大小吻合。其蛋白表达量较高,约占菌体可溶蛋白总量的40%。
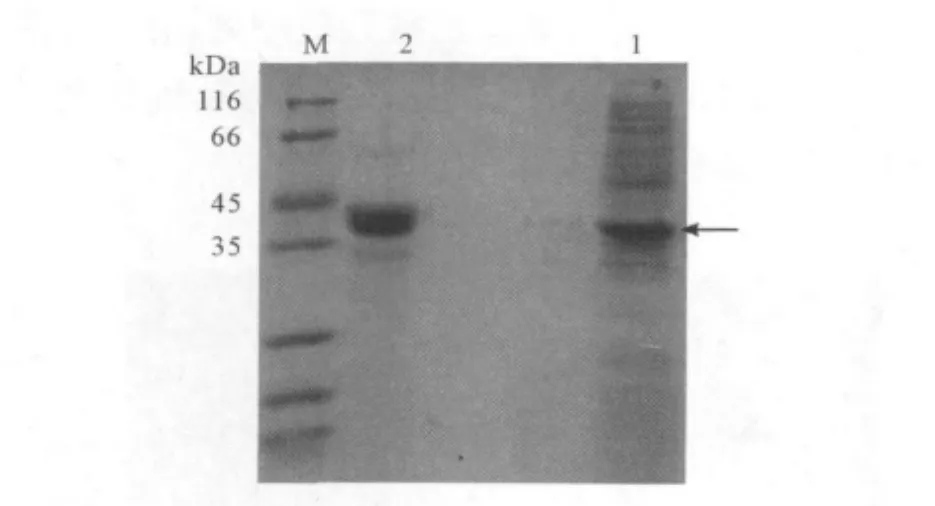
图4 重组蛋白的SDS-PAGE分析
构建好的重组菌催化3-氰基吡啶反应30 min后,通过HPLC方法,检测到大部分底物3-氰基吡啶(出峰时间为13.2 min)已经转化生成产物尼克酸(出峰时间为4.9 min),如图5所示。对构建好的重组菌利用镍柱亲和层析的方法得到了纯的腈水解酶,该酶对多种腈类底物均表现出了较高的催化活力。在pH值为7.0、40℃条件下,该酶催化3-氰基吡啶反应的速率为 15.7 μmol·min-1·mg-1。

图5 重组腈水解酶菌株对3-氰基吡啶转化结果的HPLC图谱
3 结束语
本文从华东理工大学生物反应器工程国家重点实验室自行筛选得到的菌株Rhodobacter sphaeroides LHS-305(CCTCC M209065)中克隆出1个新的腈水解酶基因,其序列与NCBI数据库中已知腈水解酶基因的最高核苷酸序列相似性为82%,与已知红细菌属中的腈水解酶基因最高核苷酸序列相似性为38.4%,但与最高氨基酸序列相似性只有17.2%。成功构建了表达载体 pET28a-nit,并在大肠杆菌Rosetta(DE3)中进行了高效表达,通过SDS-PAGE方法检测到目的蛋白得到成功表达。通过催化3-氰基吡啶反应,验证了重组菌和纯化后的重组酶的催化活力。
[1]Banerjee A,Kaul P,Banerjee U C.Purification and characterization of an enantioselective arylacetonitrilase from Pseudomonas putida[J].Arch Microbiol,2006,184:407-418.
[2]Singh R,Sharma R,Tewari N,et al.Nitrilase and its application as a‘green’catalyst[J].Chem Biodiversity,2006,3:1279 -1287.
[3]Brady D,Dube N,Pettersen R.Green chemistry:highly selective biocatalytic hydrolysis of nitrile compounds[J].S Afr J Sci,2006,102:339 -344.
[4]Martinkova L,Kren V.Nitrile-and amide-converting microbial enzymes:stereo-,regio-and chemoselectivity[J].Biocatal Biotransform,2002,20:73 -93.
[5]Martinkova L,Mylerova V.Synthetic applications of nitrile-converting enzymes[J].Current Organic Chemistry,2003,7(13):1279 -1295.
[6]Banerjee A,Sharma R,Banerjee U.The nitrile-degrading enzymes:current status and future prospects[J].Appl Microbiol Biotechnol,2002,60:33 -44.
[7]徐建妙,郑裕国,沈寅初.腈水解酶的来源、结构、作用机制及其应用[J].微生物学通报,2005,32(5):141-146.
[8]Stalker D M,Mcbride K E.Cloning and expression in Escherichia coli of a Klebsiella ozaenae plasmid-borne gene encoding a nitrilase specific for the herbicide bromoxynil[J].J BIOTECHNOL,1987,169:955 -960.
[9]王清路,张锐,倪万潮,等.透明颤菌血红蛋白基因vgb和腈水解酶基因bxn在毕赤酵母中的表达研究[J].生物工程学报,2004,20(5):730 -735.
[10]Hui L,Lu F,Yanhong C,et al.Gene cloning,overexpression,and characterization of the nitrilase from Rhodococcus rhodochrous tg1-A6 in E.coli[J].Appl Biochem Biotechnol,2010,160:393 -400.
[11]Liu J,Zhang Z,Li A,et al.Significantly enhanced production of recombinant nitrilase by optimization of culture conditions and glycerol feeding[J].Appl Microbiol Biotechnol,2011,89:665 -672.
[12]应革,武威,何朝族.TAIL-PCR方法快速分离Xcc致病相关基因序列[J].生物工程学报,2002,18(2):182-186.
