乳山湾外海溶氧低值区细菌多样性初步研究*
2012-09-06崔志松王绍良臧家业冉祥滨
崔志松,王绍良,臧家业,冉祥滨
(国家海洋局 第一海洋研究所,山东 青岛266061)
乳山湾外海溶氧低值区细菌多样性初步研究*
崔志松,王绍良,臧家业*,冉祥滨
(国家海洋局 第一海洋研究所,山东 青岛266061)
采用化学裂解法从乳山湾外海溶氧低值区不同溶解氧质量浓度(3.7~7.0mg·L-1)的6个站位海水样品中提取了环境DNA样品。以试剂盒纯化后的DNA样品为模板扩增其16SrRNA基因V3区,通过变性梯度凝胶电泳、分子文库构建及DNA测序对溶氧低值区海水中的细菌群落结构进行研究。结果表明,乳山湾外海溶氧低值区不同溶解氧质量浓度的6个站位底层海水样品中的细菌群落结构是相似的,它们均由隶属于Alteromonas(交替单胞菌属)、Salegentibacter(需盐杆菌属)等10个属的18种细菌组成。系统发育分析发现这些细菌分别属于α变形菌纲(2种)、γ变形菌纲(12种)和黄杆菌纲(3种)三个大类。在乳山湾外海溶氧低值区的海水样品中细菌多样性最高的类群是γ变形菌纲。
溶氧低值区;生物多样性;乳山湾;变性梯度凝胶电泳
海水中溶解氧质量浓度的变化受物理、化学和生物等多种过程的影响,所以它成为反映水体水质和生物生长状况的重要参数。河口和近海水域营养盐及其它有机污染物的总量剧增[1]极大地促进了相关海域的初级生产力,也造成了其底层的溶氧水平较低甚至缺氧现象,形成底层溶氧低值区(3.7~7.0mg·L-1)或低氧区(≤2.0mg·L-1)[2]。溶氧低值区和低氧区的产生、存在可能对海洋生态系统的稳定性、底栖生物的分布及生物多样性等产生重要影响。同时,底栖生物、底沉积物中的微生物种类和分布也极大地影响着底部水体溶解氧的分布。
海洋溶氧低值现象已越来越引起海洋学家的关注,河口、海湾等水体都存在溶氧低值区[2-5]。其中,关于河口区域溶氧低值现象的研究最为深入[2,6-7],而对近岸海域底层溶氧低值现象及其成因一直未进行深入的研究。关于近海溶氧低值现象对微生物群落结构变化影响的研究也很少;反之,涉及微生物在溶氧低值区或低氧区形成过程中所起到作用的研究也少有报道。微生物可以加速有机物的分解,而这一过程消耗水体中的氧气,这在一定程度上造成了近岸水体底层氧的亏损。对近海溶氧低值区中微生物群落结构、优势种的分析有利于加深对溶氧低值水域生物地球化学过程的认识,并且能够为低氧区形成的原因提供一些可参考的依据。现以乳山湾外海溶氧低值区(3.7~7.0mg·L-1)为研究对象,结合不依赖培养的分子生态学技术、DNA测序技术、序列分析技术对乳山湾外海溶氧低值区的细菌多样性进行初步研究,以期为阐明溶氧低值环境压力与微生物种群结构、优势种之间的关系提供一些依据。
1 材料与方法
1.1 站位的选择及底层海水化学参数
根据2009-09乳山湾外海不同站位表层、10m、底层三个层次海水样品各项化学参数的调查结果,选取了B7,C3,C6,E7,F3,F7共6个站位的底层海水样品进行微生物多样性研究(图1和表1)。6个站位底层海水溶解氧由高到低依次是站位C3(7.0mg·L-1),B7(6.7mg·L-1),F7(6.5mg·L-1),F3(5.9mg·L-1),E3(5.6mg·L-1)和C6(3.7mg·L-1)。由调查结果可见,不同站位之间溶解氧水平的差异显著,且均明显低于其水体溶解氧的理论饱和质量浓度,而6个站位底层海水的温度、盐度区域性差异明显小于溶解氧差异,其变化幅度也较小。这表明调查区域存在一定程度上的耗氧过程。
与温度、盐度变化规律不同的是,在溶解氧较低的C6,E3和F3站位底层海水样品中硝酸盐和磷酸盐的质量浓度也明显高于其它3个站位(表1)。这表明水体耗氧过程从一定程度上影响了营养盐的质量浓度及分布,并可能影响微生物种类及相对丰度。从目前的分析来看,乳山湾外近岸低氧区的面积较小,其成因主要是由于水体和底泥耗氧所致;人类活动(污水排放、人工养殖)的增多导致大量的有机质排入近海,并为耗氧过程提供了物质基础[8]。
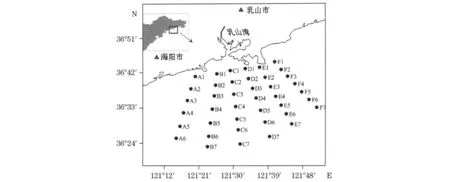
图1 乳山湾外海溶氧低值区调查站位图Fig.1 Sampling stations at the contiguous area with low value of dissolved oxygen of the Rushan Bay

表1 各站位海水样品化学参数Table 1 Chemical parameters of the sea water samples at different stations
1.2 采用化学裂解法提取环境DNA
采用化学裂解法从海水样品中提取环境总DNA,具体步骤为将500mL海水用0.22μm微孔滤膜过滤以收集细菌细胞,将滤膜转移到50mL离心管。加入10mL DNA提取液,74μL蛋白酶K,涡旋振荡使其充分混匀,37℃水浴30min。加入1 100μL 20%(质量分数)SDS,65℃水浴2h,每隔15~20min混匀一次。7 000r/min离心10min,将上清移入新的离心管。加入等体积的氯仿/异戊醇,10 000r/min离心10min,将上清移入新的离心管。加入0.6倍体积异丙醇室温沉淀1h,15 000r/min离心20min,弃上清。加入1mL 70%乙醇(体积分数)洗涤DNA沉淀,15 000r/min离心10min。弃上清,待乙醇挥发后加入200 μL无菌水溶解DNA。
1.3 采用试剂盒纯化环境DNA样品的纯化
采用化学裂解法提取到的环境DNA样品中含有抑制Taq酶活性的杂质,不利于后续的分子生物学操作,所以需要采用Promega公司的Wizard DNA Clean-Up System纯化试剂盒对环境DNA样品进行纯化。
1.4 16SrRNA V3区的PCR扩增
以纯化后的环境DNA样品为模板,采用引物V3F,V3R对细菌16SrRNA序列的V3区进行PCR扩增。在反应体系中加入引物 V3F(10μmol/L)1μL,引物 V3R(10μmol/L)1μL,dNTP(各2.5mmol/L)4 μL,10XPCR Buffer 5μL,rTaq 1U,模板DNA约50ng,最后用灭菌蒸馏水补至50μL。引物序列及采用的PCR热循环程序参照文献[9]。
1.5 变性梯度凝胶电泳(Denaturing Gradient Gel Electrophoresis,简称DGGE)
DGGE胶中聚丙烯酰胺的质量分数为8%,变性剂范围为30%~60%。将电泳缓冲液预热至60℃,然后加样。每个环境DNA样品平行做3管共150μL;上样前需要将PCR产物用无水乙醇沉淀并浓缩到15 μL,然后与Loading Buffer混匀后上样。电泳程序:30V,15min;130V,4.5h。
1.6 环境16SrRNA分子文库的构建
以纯化后的环境DNA样品为模板采用通用引物16SF,16SR[9]对细菌16SrRNA基因进行PCR扩增。在反应体系中加入引物16SF(10μmol/L)1μL,引物16SR(10μmol/L)1μL,dNTP(各2.5mmol/L)4μL,10XPCR Buffer 5μL,rTaq 1U,模板DNA约50ng,最后用灭菌蒸馏水补至50μL。采用的PCR程序:95℃4min;94℃45s,55℃1min,72℃1.5min,循环30~32次;72℃15min,4℃保持。
采用广州东盛PCR产物/DNA片段纯化试剂盒对16SrRNA的PCR扩增产物进行纯化。采用Takara pMD18-T连接试剂盒将纯化后的PCR产物连接到克隆载体pMD18-T。在反应体系中加入pMD18-T(50 ng·μL-1)1μL,SolutionⅠ(含T4DNA连接酶)5μL,PCR纯化片段约300ng,最后用灭菌蒸馏水补至10 μL。轻柔混匀,16℃温浴8h。按照分子克隆实验指南中的方法将连接产物转化Escherichiacoli(E.coli)感受态细胞DH5α,获得含有环境样品16SrRNA基因的分子文库。
1.7 分子文库的筛选、测序及系统发育分析
采用菌落PCR的方法对其中含有插入片段的阳性克隆子进行筛选、划线分离。以直接热裂解阳性克隆子得到的DNA及纯化后环境DNA样品为模板,扩增其16SrRNA V3区片段,随后进行变性梯度凝胶电泳,根据其DGGE图谱确定与环境DNA样品的各条带相对应的克隆子条带。挑取含有目标16SrRNA片段的克隆子,由上海博尚生物技术有限公司完成双向测序。测序结果提交到NCBI网站,采用BLAST软件进行比对分析。再将目标序列与模式株或参考菌株序列导入DNAMAN软件进行系统发育学分析,构建系统发育树。
2 结果与分析
2.1 从海水样品中提取环境DNA
采用化学裂解法从来自6个站位B7,C3,C6,E3,F3,F7(表层、10m、底层三个层次)的18个海水样品中均能提取到环境DNA。
2.2 环境DNA样品的纯化
环境DNA样品经试剂盒纯化后,条带亮度会略有降低,说明DNA在纯化过程中有一定的损失,但可以去除环境DNA样品中抑制Taq酶活性的杂质,便于后续的分子生物学操作。
2.3 16SrRNA V3区的PCR扩增结果
虽然以C3-表、C3-底、C6-表、C6-10m等少数几个未经纯化的环境DNA样品为模板能够直接扩增到16SrRNA V3区200bp左右的目的条带,但大多数环境DNA样品由于含有抑制Taq酶活性的杂质,未能扩增到目的条带。这表明在对环境DNA样品进行分子生物学操作前,有必要先进行纯化处理。而以纯化后的环境DNA样品为模板都可以扩增得到目的条带,这说明经过纯化处理后的环境DNA样品可以满足后续分子生物学实验的要求。
2.4 变性梯度凝胶电泳(DGGE)结果
通常DGGE谱图中的每个条带对应海水样品细菌群落中的某种细菌,并且其信号强弱在一定程度上反映菌群中该细菌的相对丰度。在图2a中泳道1~12依次为C3表,C3-10m,C3底,F3表,F3-10m,F3底,E3表,E3-10m,E3底,C6表,C6-10m,C6底;在图2b中泳道1~6依次为B7表,B7-10m,B7底,F7表,F7-10m,F7底。尽管随着站位不同、海水层次不同以及溶解氧水平不同,上述18个海水样品的DGGE谱图存在一定的差异,即条带的多寡及亮度不同,但是其总体上反映的细菌群落结构是相似的(图2),即它们是由最多18个代表不同菌种的条带组成的(图2)。值得注意的是,在所有海水样品中条带9,10与其余条带相比其信号较强且相对稳定,说明在此溶氧条件范围内它们所代表的细菌相对丰度较高。
2.5 16SrRNA分子文库的构建与筛选
根据DGGE图谱条带分析结果(图2),选取条带丰富、具有代表性的C3-10m和C6-10m两个样品扩增环境16SrRNA基因,并构建分子文库。采用菌落PCR从C3-10m分子文库中55个单菌落中筛选出21个阳性克隆子;从C6-10m分子文库中56个单菌落中筛选出29个阳性克隆子。以直接热裂解阳性克隆子得到的DNA及纯化后环境DNA样品C3-10m和C6-10m为模板,分别扩增其16SrRNA V3区片段,随后以DGGE图谱条带分析的方法确定与环境DNA样品的各条带相对应的克隆子条带。在图3和图4中,泳道上的数字表示分子文库中阳性克隆子的编号,C3-10m或C6-10m表示以这2个环境DNA样品为模板扩增到的DGGE-PCR产物。在代表乳山湾外海溶氧低值区海水样品中细菌群落的18个条带中,除1个条带未在分子文库中获得对应克隆子外,其余17个条带都能够在文库中获得对应克隆子。
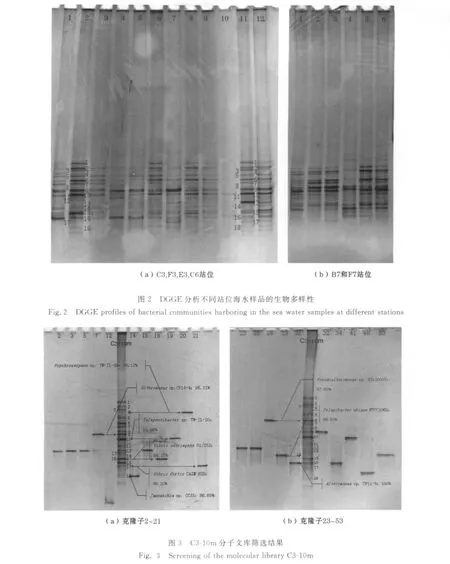

图4 C6-10m分子文库筛选结果Fig.4 Screening of the molecular library C6-10m
通过测序、BLAST比对分析,可知由DGGE图谱条带所代表的海水样品中的细菌种类(表2、图3和图4),这些细菌隶属于至少10个属。在Genbank中,条带1与海洋细菌SCRIPPS_413(AF359548)亲缘关系最近,两者的相似度达到98.66%。菌株SCRIPPS_413可能参与某些甲藻累积麻痹性贝毒的生物过程[10]。条带3与一株从东太平洋深海沉积物中分离到的、能够降解多种烷烃的模式种柴油食烷菌(Alcanivorax dieselolei NO1A;AY683531)的亲缘关系最近,但两者的相似度仅为92.05%[11]。条带4、条带10分别与中国南海分离到的产色素异养细菌冷蛇菌属细菌 TW-JL-49(Psychroserpens sp.TW-JL-49;DQ073103)、需盐杆菌属细菌TW-JL-20(Salegentibacter sp.TW-JL-20;DQ073102)亲缘关系最近,两者的相似度分别为99.12%,93.88%。这类产色素异养细菌主要存在于海洋透光层,在表层海水中的丰度最高,它们可占到可培养细菌的39.6%,或占细菌总数的1.4%[12]。条带5与从韩国黄海潮滩分离到的Kangiella koreensis DSM 16069(NR_027574)亲缘关系最近,但两者的相似度仅为84.53%[13]。Genbank中与条带6,11,13亲缘关系最近的菌株均属于假交替单胞菌属(Pseudoalteromonas)。其中条带6与从北冰洋沉积物中分离到的Pseudoalteromonas sp.BSs20007(EU365511)亲缘关系最近,两者的相似度达到97.80%。条带11与1 855m深海沉积物中分离到的一株嗜冷菌Pseudoalteromonas sp.SM9913(AY305857)亲缘关系最近,两者的相似度达到98.67%[14]。条带13与太平洋深海多金属结核区分离到的Pseudoalteromonas sp.ws17(AJ704790)亲缘关系最近,但两者的相似度仅为90.56%[15]。条带7,9,16与交替单胞菌属(Alteromonas)的3株细菌亲缘关系最近,它们之间的相似度为95.21%~100%。据报道,这个属的可培养细菌构成了南海沉积物中蛋白酶产生细菌的一个最大类群(34.6%)[16]。Genbank中与条带8,15,17亲缘关系最近的菌株均属于弧菌属(Vibrio)。条带8和条带17分别与兼性厌氧细菌Vibrio hepatarius LMG 20362(NR_025491)、富通弧菌CAIM 629(Vibrio fortis CAIM 629;NR_025575)亲缘关系最近,两者的相似度分别为98.57%,99.20%。菌株CAIM 629可发酵葡萄糖和甘露醇,而菌株LMG 20362除上述两种碳源外还可以发酵蔗糖、苦杏素[17]。条带15与病害濒死牡蛎(Crassostrea gigas)血淋巴中分离到的需钠弧菌01/252(Vibrio natriegens 01/252;AJ874353)亲缘关系最近,两者的相似度达到98.15%。注射实验证明菌株01/252是无毒的,而弧菌属的其它2个种(V.aestuarianus和V.splendidus)则会导致牡蛎死亡[18]。条带12与从美国伍兹霍尔盐沼表层沉积物中分离到的一株耐盐、Fe(Ⅲ)还原细菌海藻希瓦氏菌(Shewanella algae;X81622)亲缘关系最近,两者的相似度达到99.27%[19]。条带14与遍在远洋杆菌HTCC1062(Pelagi-bacter ubique HTCC1062;AF510191)亲缘关系最近,两者的相似度达到98.80%。菌株HTCC1062所代表的α-变形菌纲SAR11类群,是一类体积微小(0.01μm3)、难以培养的细菌,可占到大洋浮游细菌种群的26%[20]。条带18与1株好氧海洋细菌Jannaschiasp.CCS1(CP000264)亲缘关系最近,但两者的相似度仅为86.85%。菌株CCS1是一类基于细菌叶绿素α的光能自养菌的典型代表[21]。
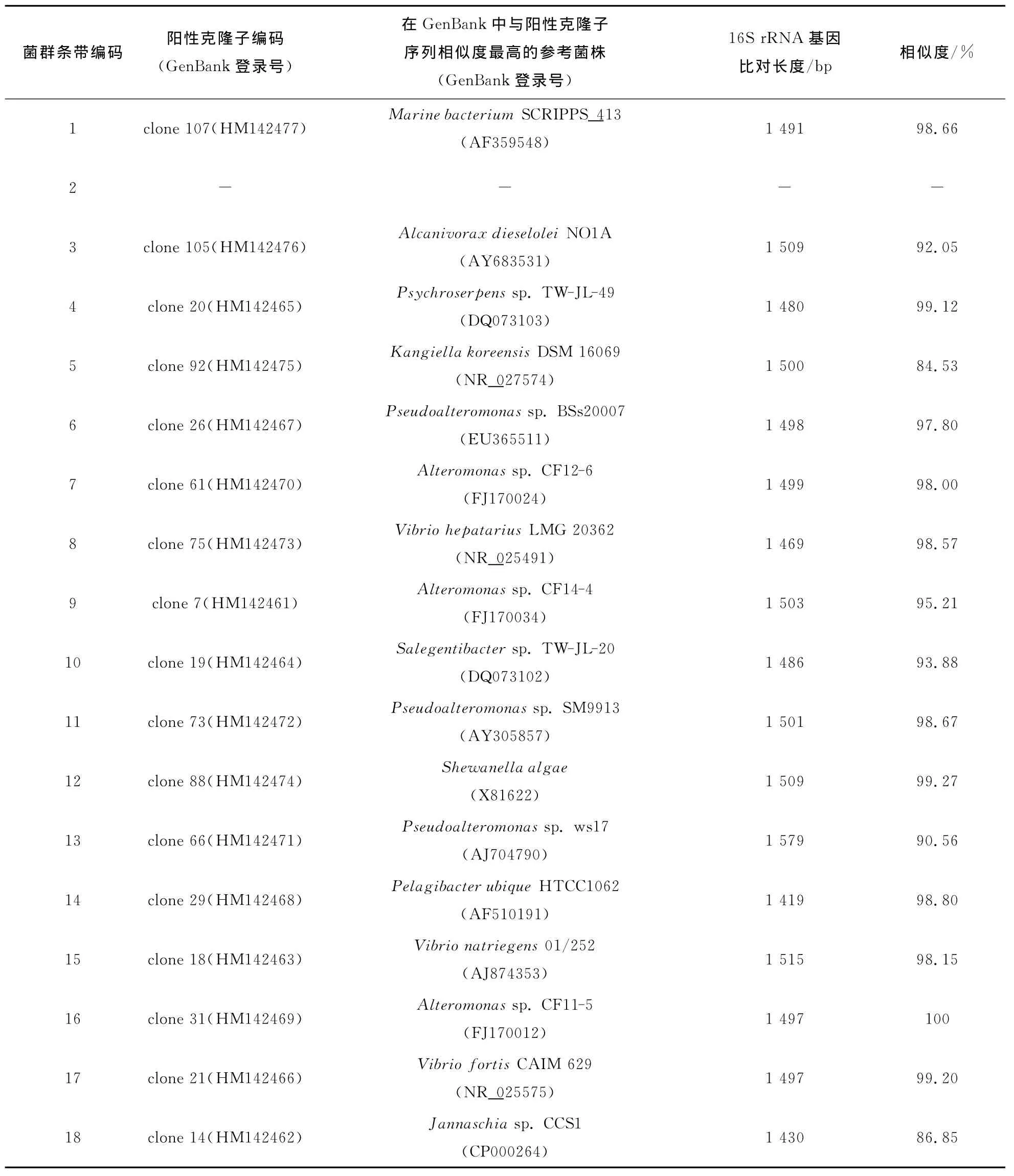
表2 海水样品C3-10m和C6-10m中细菌群落结构分析Table 2 Analysis of the structure of bacterial community harboring in sea water sample C3-10mand C6-10m
通过DNAMAN软件分析,构建了基于16SrRNA基因序列相似度的乳山湾外海低氧区海水中细菌的系统发育树(图5)。乳山湾外海低氧区细菌分别属于α变形菌纲(12种)、γ变形菌纲(2种)、黄杆菌纲(3种)三个大类。综上,在乳山湾外海溶氧低值区的海水样品中细菌多样性最高的类群是γ变形菌纲。
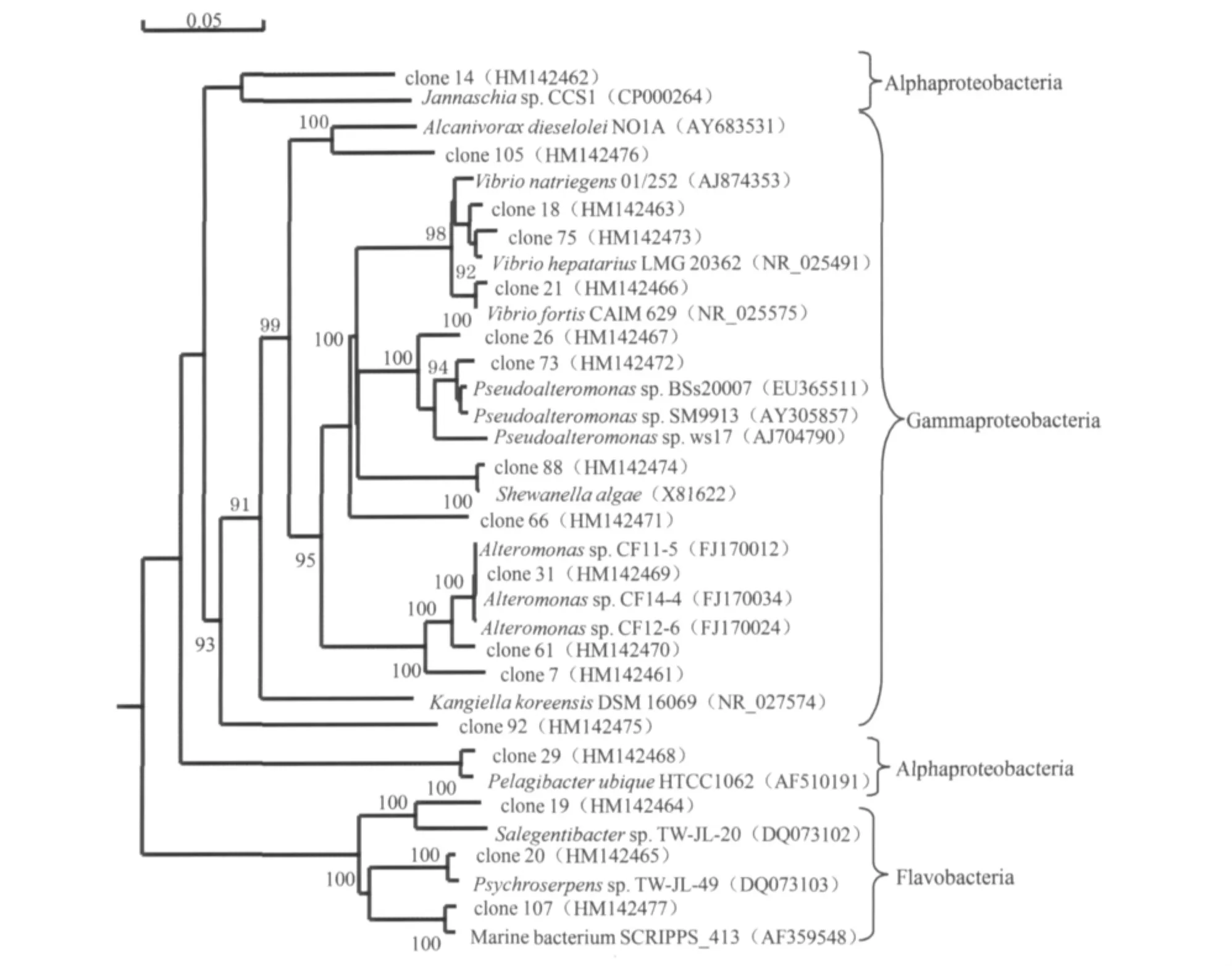
图5 乳山湾外海溶氧低值区海水样品中细菌的系统发育分析Fig.5 Phylogenetic analysis of the bacteria harboring in the sea water samples of the contiguous area with low value of dissolved oxygen of the Rushan Bay
3 讨 论
由于海洋微生物中的绝大多数是未培养或不可培养的,从沉积物、海水等海洋环境样品中直接提取总DNA然后进行分子生态学分析及核苷酸序列测定越来越受到海洋学家的重视。李友训等报道了一种改进后的化学裂解和酶消化相结合的海洋沉积物总DNA提取、纯化方法,并对东太平洋深海沉积物中的细菌多样性进行了初步分析[22]。我们采用滤膜抽滤、化学裂解、酶消化的方法成功地从乳山湾外海溶氧低值区多个站位的海水样品中提取到环境DNA,其中部分DNA的纯度较高,甚至可以不经过纯化而直接用于PCR操作。经过Promega试剂盒纯化后的所有环境DNA均可达到进行扩增、构建分子文库等分子生物学操作的要求。这种优化后的海水样品DNA提取方法耗时短、产率大、纯度高,能够为海洋微生物的多样性研究在方法学上提供一定的支持和参考。
采用DGGE技术研究环境微生物的多样性通常有2种方法。1)对环境样品的DGGE条带直接切胶回收再测序。2)先从环境DNA中扩增16SrRNA基因全序列、构建分子文库,然后再进行DGGE分析和测序。本研究采用了第2种方法,该方法的优点在于可以获得16SrRNA基因全长片段,其比对结果可信度高;缺点在于环境样品的生物多样性将被低估。在代表乳山湾外海溶氧低值区海水样品中细菌群落的18个条带中,17个条带在文库中获得对应的克隆子,这表明该文库具有较高的呈现率。实际上,DGGE图谱中的有些条带往往代表一种以上微生物(其16SrRNA基因V3区解链温度一致)。我们在文库中也挑取了与同一条带相对应的多个克隆子进行了测序、比对,但是并未测得不同的序列,即它们所含有的基因序列是一致的(例如,与条带10对应的4个克隆:克隆19、克隆97、克隆98、克隆99)。所以,在后续工作中需要提高文库的容量来达到更好呈现实际生物多样性的实验目的。
我们的研究发现,尽管所选乳山湾外海底层溶氧低值区不同站位之间存在较明显的溶解氧差异,但是它们之间的细菌种群结构差异并不显著。这说明乳山湾外海海水中的细菌群落对溶氧低值环境压力的适应能力较强。目前,关于溶氧低值海洋环境中微生物群落结构分析的报道不多。其中,刘敏等应用PCR-DGGE技术对长江口低氧区细菌群落进行分析发现,在长江口低氧水体中的细菌群落组成与非低氧水体的是不同的,在低氧区水体中的优势菌种是属于拟杆菌门(Bacteroides)黄杆菌纲(Flavobacteria)的细菌[23]。对乳山湾外海溶氧低值区不同溶解氧水平(3.7~7.0mg·L-1)的6个站位底层海水样品的细菌群落结构进行了分析和比较,并发现由条带9和条带10代表的2种优势菌种之一也是属于上述黄杆菌纲的细菌(Salegentibacter sp.TW-JL-20,93.88%)。但是,还需要进一步研究这些黄杆菌纲细菌的生理生态功能,以阐释溶氧低值区形成与微生物活动之间的关系。季倩等也对长江口外缺氧区进行了调查研究,发现在低氧区形成前后,微微型浮游生物的群落结构发生了明显变化,聚球藻和病毒的丰度明显增加,而微微型真核浮游植物的丰度则急剧下降,但异养细菌丰度的变化不明显[24]。在本研究中,乳山湾外海溶氧低值区不同溶解氧水平的6个站位底层海水样品的细菌群落结构也是相似的,它们均由隶属于10个属的18种细菌组成,其中细菌多样性最高的类群是γ变形菌纲的细菌。从我们的比对结果来看,这些细菌大多数属于化能异养细菌和光能异养细菌,并且其近缘细菌的总丰度在自然菌群中所占比例较高(条带4、条带10、条带14),它们能够在有机物分解过程中消耗氧气,这些细菌在合适条件下的大量增殖可能是导致形成溶氧低值区的因素之一。目前,乳山湾外海低氧区面积较小,且其它环境参数差异不大(表1),这也可能是导致调查区域细菌群落结构大致相似的重要原因。对溶氧低值区微生物种群结构及优势菌种的解析可以为后续的微生物分离培养、溶氧低值或缺氧现象的生物化学成因分析提供一定的参考。
致谢:中国海洋大学海洋生命学院杨官品教授对本文的细致修改。
(References):
[1] ZHANG J,ZHANG Z F,LIU S M,et al.Human impacts on the large world rivers:Would the Changjiang(Yangtze River)be an illustration?[J].Global Biogeochemical Cycles,1999,13:1099-1105.
[2] LI D J,ZHANG J,HUANG D J,et al.Oxygen depletion in the contiguous area of the Yangtze River Estuary[J].Science in China series D,2002,32(8):686-694.李道季,张经,黄大吉,等.长江口外氧的亏损 [J].中国科学:D辑,2002,32(8):686-694.
[3] DIAZ,R J,ROSENBERG R.Spreading dead zones and consequences for marine ecosystems[J].Science,2008,321:926-929.
[4] MORRISON J M,CODISPOTI L A,SMITH S L,et al.The oxygen minimum zone in the Arabian Sea during 1995[J].Deep-Sea Research II,1999,46:1903-1931.
[5] WISHNER K,LEVIN L,GOWING M,et al.Involvement of the eastern tropical Pacific oxygen minimum zone in benthic zonation on a deep seamount[J].Nature,1990,346:57-59.
[6] LIN H Y,LIU S,HAN W Y.Seasonal hypoxia phenomenon at the bottom layer of the Pearl River estuary and its potential hazard of inducing CTB[J].Journal of Zhanjiang Ocean University,2001,21(sup):25-28.林洪瑛,刘胜,韩舞鹰.珠江口底层海水季节性缺氧现象及其引发CTB的潜在威胁 [J].湛江海洋大学学报.2001,21(增刊):25-28.
[7] RABALAIS N N,TURNER R E,JUCTIC D,et al.Nutrient changes in the Mississippi River and system responses on the adjacent continental shelf[J].Estuaries,1996,19:386-407.
[8] RAN X B,ZANG J Y,WEI Q S,et al.Characteristics of dissolved oxygen and its influencing factors in the Rushan Bay mouth and its adjacent waters[J].Acta Oceanologica Sinica,2011,33(4):173-180.冉祥滨,臧家业,韦钦胜,等.乳山湾口及其邻近海域溶解氧分布特征及影响因素研究[J].海洋学报,2011,33(4):173-180.
[9] CUI Z S,LAI Q L,DONG C M,et al.Biodiversity of Polycyclic Aromatic Hydrocarbon-Degrading Bacteria from deep sea sediments of the Middle Atlantic Ridge[J].Environmental Microbiology,2008,10(8):2138-2149.
[10] HOLD G L,SMITH E A,RAPPE M S,et al.Characterisation of bacterial communities associated with toxic and non-toxic dinoflagellates:Alexandriumspp.and Scrippsiella trochoidea[J].FEMS Microbiol.Ecol.,2001,37(2):161-173.
[11] LIU C,SHAO Z Z.Alcanivorax dieselolei sp.nov.,a novel alkane-degrading bacterium isolated from sea water and deep-sea sediment[J].International Journal of Systematic and Evolutionary Microbiology,2005,55:1181-1186.
[12] DU H,JIAO N,HU Y,et al.Diversity and distribution of pigmented heterotrophic bacteria in marine environments[J].FEMS Microbiol.Ecol.,2006,57:92 105.
[13] YOON J H,OH T K,PARK Y H.Kangiella koreensis gen.nov.,sp.nov.and Kangiella aquimarinasp.nov.,isolated from a tidal flat of the Yellow Sea in Korea[J].Int.J.Syst.Evol.Microbiol.,2004,54:1829-1835.
[14] CHEN X L,ZHANG Y Z,GAO P J,et al.Two different proteases produced by a deep-sea psychrotrophic bacterial strain,Pseudoaltermonas sp.SM9913[J].Marine Biology,2003,143(5):989-993.
[15] XU M X ,WANG P,WANG F P,et al.Microbial diversity at a deep-sea station of the Pacific Nodule Province[J].Biodiversity and Conservation,2005,14(14):3363-3380.
[16] ZHOU M Y,CHEN X L,ZHAO H L,et al.Diversity of both the cultivable protease-producing bacteria and their extracellular proteases in the sediments of the South China Sea[J].Microbial Ecology,2009,58:582-590.
[17] THOMPSON F L,THOMPSON C C,HOSTE B,et al.Vibrio fortis sp.nov.and Vibrio hepatarius sp.nov.,isolated from aquatic animals and the marine environment[J].Int.J.Syst.Evol.Microbiol.,2003,53:1495-1501.
[18] GARNIER M,LABREUCHE Y,GARCIA C,et al.Evidence for the Involvement of Pathogenic Bacteria in Summer Mortalities of the Pacific Oyster Crassostrea gigas[J].Microbial Ecology,2007,53(2):187-196.
[19] ROSSELLO-MORA R A,CACCAVO F Jr,OSTERLEHER K,et al.Isolation and taxonomic characterization of a halotolerant,facultatively iron-reducing bacterium[J].Systematic and Applied Microbiology,1995,17(4):569-573.
[20] RAPPE M S,CONNON S A,VERGIN K L,et al.Cultivation of the ubiquitous SAR11marine bacterioplankton clade[J].Nature,2002,418(6898):630-633.
[21] BéjàO,SUZUKI M T,HEIDELBERG J F,et al.Unsuspected diversity among marine aerobic anoxygenic phototrophs[J].Nature,2002,415:630-633.
[22] LI Y X,LI F C,QIN S,et al.DNA extraction and DGGE analysis of the bacterial community of a deep-sea sediment sample collected at the East Pacific Ocean[J].Marine Sciences,2008,32(12):69-74.李友训,李富超,秦松,等.东太平洋深海沉积物中 DNA的提取及细菌多样性初步分析 [J].海洋科学,2008,32(12):69-74.
[23] LIU M.Analysis of the bacterial communities harboring in the hypoxia area of the Yangtze River Estuary and Huanghai Cold Water Mass by PCR-DGGE[D].Qingdao:Institute of Oceanology,Chinese Academy of Sciences,2007.刘敏.应用PCR-DGGE技术分析长江口低氧区和黄海冷水团的细菌群落组成 [D].青岛:中国科学院研究生院(海洋研究所),2007.
[24] JI Q.Study of the picoplankton harboring in the water body and bottom layer at the contiguous area of the Yangtze River Estuary[D].Shanghai:East China Normal University,2008.季倩.长江口邻近海域水体及底栖微微型浮游生物研究 [D].上海:华东师范大学,2008.
(高 峻 编辑)
Bacterial Diversity in an Offshore Low Dissolved Oxygen Content Area Contiguous to Rushan Bay
CUI Zhi-song,WANG Shao-liang,ZANG Jia-ye,RAN Xiang-bin
(The First Institute of Oceanography,SOA,Qingdao 266061,China)
Environmental DNA was extracted from sea waters collected at 6sampling stations with different dissolved oxygen level located at an offshore low dissolved oxygen content(3.7~7.0mg·L-1)area contiguous to Rushan Bay with a chemical lysis method.The bacterial community structures of these stations were determined according to the V3region of bacterial 16SrRNA genes amplified with purified environmental DNA as templates and analyzed with combination of denaturing gradient gel electrophoresis,library construction and sequencing.Results showed that the bacterial communities were similar in structure with each other,although the dissolved oxygen contents at the bottom layers of these stations were different.In total,18bacterial species were identified belonging to 10genera ofα-Proteobacteria,γ-Proteobacteria and Flavobacter as were determined with phylogenetic analysis.Species ofγ-proteobacteria were the most diverse in this area.
low dissolved oxygen content area;biodiversity;Rushan Bay;denaturing gradient gel electrophoresis
January 24,2011
Q939
A
1671-6647(2012)03-0369-11
2011-01-24
中国近海海洋综合调查与评价专项课题——浮山湾外海低氧区调查与研究(908-01-BC14);国家海洋局第一海洋研究所基本科研业务费专项资金——海洋专性解烃菌Cycloclasticus spp.的代谢特性及协同降解高分子量多环芳烃的研究(2010G23)
崔志松(1981-),男,山东青岛人,硕士,助理研究员,主要从事环境微生物学方面研究.E-mail:czs@fio.org.cn
*通讯作者
