毛白杨MSAP体系优化及DNA甲基化的初步分析1)
2012-08-09马开峰张志毅王斯琪宋跃朋张德强
马开峰 张志毅 王斯琪 宋跃朋 张德强
(林木育种国家工程实验室(北京林业大学),北京,100083)
DNA甲基化是基因组的一种主要的表观遗传修饰形式,指由Dnmt催化并以S-腺苷甲硫氨酸(SAM)作为甲基供体发生催化反应,产生N-甲基腺嘌呤、N-甲基胞嘧啶和C-甲基胞嘧啶的现象[1]。在真核生物中,DNA甲基化主要以5-甲基胞嘧啶的形式存在[2-3]。正常情况下,植物基因组DNA中约20%~30%的胞嘧啶残基发生甲基化,其中绝大部分的甲基化位于CpG二核苷酸和CpNpG三核苷酸对中[4-7],并在植物基因表达调控、外源基因防御、个别基因表达模式的遗传等途径中起着重要的作用[8-9]。 Methylation-sensitive Amplified Polymorphism(MSAP)是在AFLP(Amplified Fragment Length Polymorphism)基础上发展起来的一种分子标记技术,操作相对简便,同时不需知道被检测DNA的序列信息,即可在全基因组范围检测5’-CCGG位点的胞嘧啶甲基化变化,在不同生物上具有通用性[10-22]。该技术作为检测胞嘧啶甲基化的手段始见于Reyna-López等[10]检测二形真菌基因组 DNA甲基化,之后被广泛用于玉米[11-12]、水稻[13-14]、棉花[15-16]、油菜[17]等作物中。在林木中,研究者利用该技术对落叶松[18]、苹果[19]、甜桔栽培种[20]、红树[21]、杉木[22]等树种基因组 DNA 甲基化进行了研究,并认为基因组甲基化与林木体胚发生、生物量及杂种优势密切相关。
毛白杨(Populus tomentosa Carr.)是我国特有白杨派树种,它分布广,速生,适应性和抗性强,材质优良,是华北平原营造人工丰产林和农田防护林的重要树种[23]。因此,在表观遗传学领域对其进行研究具有重要意义,但目前研究毛白杨基因组DNA甲基化的实例尚未见报道。本实验首次将MSAP技术应用于毛白杨,通过优化选择性扩增体系,对毛白杨及其种内杂交子代的CCGG甲基化相对水平、甲基化模式的遗传变异进行分析,为进一步研究毛白杨基因组DNA甲基化与表型性状的相关性及杂种优势形成机理奠定基础。
1 材料与方法
1.1 材料来源
2008年1月分别从陕西杨凌职业技术学院、山东省国营冠县苗圃采集毛白杨P.tomentosa-1(Pt-1,♀)、P.tomentosa-2(Pt-2,♂)花枝运至北京林业大学温室进行水培杂交,获得毛白杨种内杂交子代P×tomentosa-151幼苗植株栽植于北京林业大学温室。2010年1月份采集Pt-1、Pt-2、P×tomentosa-151根段进行沙埋,获取幼化[24-25]后的植株,并将幼株种植于盛满营养丰富的基质土壤(V沙土∶V珍珠岩∶V蛭石∶V草炭=2 ∶1 ∶1 ∶1)的圆形花盆(内径38 cm,高30 cm)中,放置于北京林业大学温室使其生长。
1.2 试验方法
1.2.1 DNA 提取
2010年8月20 日同时采集上述幼化方法所得亲本 Pt-1(株高0.58 m,地径7.08 mm)、Pt-2(株高0.76 m,地径7.93 mm)及子代 P×tomentosa-151(株高0.84 m,地径8.23 mm)幼株茎端第3~4片功能叶,迅速放入液氮中冷冻处理,采用CTAB法提取全基因组DNA并利用NanoVueTM紫外/可见光分光光度计(GE Healthcare Company)检测,后置于冰箱中-20℃保存。
1.2.2 基因组酶切—连接与预扩增
利用对甲基化敏感的同裂酶Hpa II/Msp I(Promega公司生产)分别与限制性内切酶Eco RI(Promega公司生产)组合对材料基因组DNA进行双酶切和连接。Eco RI接头:5’-CTCGTAGACTGCGTACC-3’,5’-AATTGGTACGCAGTC-3’;Hpa II/Msp I接头:5’-GATCATGAGTCCTGCT-3’,5’-CGAGCAGGACTCATGA-3’。预扩增引物:Eco RI+A 5’-GACTGCGTACCAATTCA-3’;Hpa II/Msp I+0 5’-ATCATGAGTCCTGCTCGG-3’。
20μL预扩增反应体系(1×buffer)包括去离子水,3μL 酶切—连接产物,0.3 mmol/L 各 dNTP,E+A引物和H/M+0引物(序列见表1)各0.03 nmol,5 U Taq酶。94℃变性2 min,按以下参数扩增30个循环,94℃30 s,56 ℃30 s,72 ℃80 s,最后72 ℃延伸5 min。

表1 接头及扩增引物序列
1.2.3 选择性扩增
反应体系(1×buffer)总体积为20μL,其中引物E+A+2和H/M+3(序列见表1)、dNTP、Taq酶加入的量、预扩增产物的稀释倍数5因素按照L16(45)正交试验设计(表2)完成,稀释后的预扩增产物加入量为4μL。94℃变性2 min,按下列参数进行PCR扩增:第一个循环扩增参数为94℃30 s,65℃30 s,72℃80 s,以后将每个循环的复性温度递减0.7℃,共扩增12个循环;最后按下列参数扩增23个循环,94℃30 s,55 ℃30 s,72 ℃80 s。
1.2.4 变性聚丙烯酰胺凝胶电泳与银染
选择性扩增产物在6%的变性聚丙烯酰胺凝胶中进行电泳,至电泳指示剂(约100 bp)接近凝胶底部时停止,银染显影[13]。
1.2.5 谱带品质的主观评价与统计分析
将凝胶上显示的各泳道谱带品质分为5等级,第1等级为谱带粗细均匀,清晰可辨,无明显拖尾现象,记为“4”;第2等级为谱带粗细均匀,清晰可辨,有拖尾,记为“3”;第3等级为谱带粗细均匀,但稍有模糊,记为“2”;第4等级为谱带粗细相对均匀,但时有时无,模糊不可完全辨别,记为“1”;第5等级为谱带不可辨认或无带,记为“0”。以上记录数据经平方根转换后用于计算。
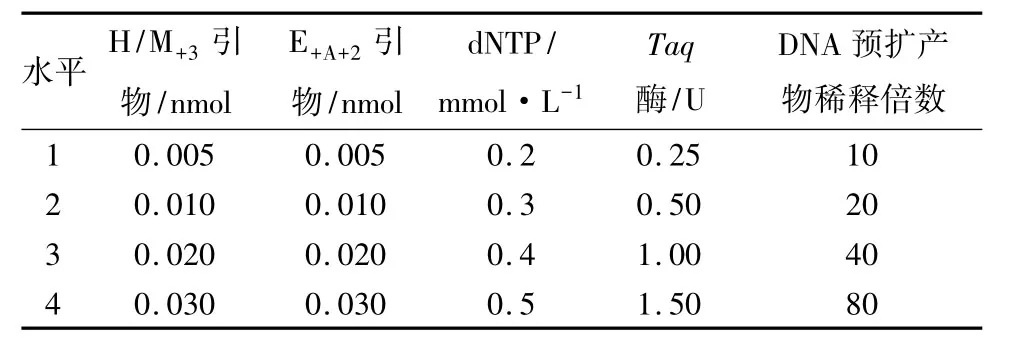
表2 选择性扩增体系因素及水平数
统计银染谱带时,对于不同材料或不同酶切处理在同一位点有带时记为“1”,无带时记为“0”。
数据统计运算在 Excel2003、SPSS13.0软件环境下进行。
2 结果与分析
2.1 MSAP 体系优化
2.1.1 基因组酶切—连接与预扩增
利用亲本基因组进行体系优化。MSAP是基于AFLP技术基础上发展起来的标记方法,在PCR过程中需要高质量的DNA模板。因此实验对提取的基因组DNA质量进行了检测,NanoVueTM紫外/可见光分光光度计(GE Healthcare Company)检测显示,OD260nm/OD280nm值为 1.82~1.84,质量浓度为230~246 ng/μL。琼脂糖凝胶电泳检测见图1A。利用Eco RI+Hpa II、Eco RI+Msp I组合分别对基因组DNA进行双酶切,为减少酶切、接头连接操作过程中DNA的损失,将酶切—连接在一步实验操作中完成,琼脂糖检测见图1B。
预扩增是MSAP标记的关键性环节,扩增产物质量的优劣直接关系到选择性扩增的成功与否。结果表明,300~500 ng范围的基因组DNA经酶切-连接后取3μL用于PCR,可得到质量较好的预扩增产物(图1C)。
2.1.2 选择性扩增
用引物 III-c、引物 IV-d、引物 V-e、引物 VI-f、引物VII-g5对引物对Pt-1、Pt-2的预扩增产扩物进行选择性扩增,银染谱带(图2)品质的主观评价是利用两次独立实验数据之和进行的(表3)。方差分析(表4)表明不同引物、dNTP、Tag酶浓度及预扩增产物稀释倍数对变性聚丙烯酰胺凝胶电泳后银染谱带品质的影响具有显著性差异,其中H/M+3引物浓度的影响最大,其次为E+A+2引物浓度。多重比较A4B4C1D3(4)E3为最优组合,即引物H/M+3、引物E+A+2、dNTP、Taq酶、预扩增产物稀释倍数分别为0.03 nmol、0.03 nmol、0.2 mmol/L、1 U(或 1.5 U)、40倍时,20μL反应体系(1×buffer)经PCR扩增、变性聚丙烯酰胺凝胶电泳后产生的银染谱带品质最佳。
2.2 毛白杨亲子代间DNA甲基化分析
2.2.1 毛白杨CCGG胞嘧啶甲基化模式及亲子代间甲基化状态
将引物H/M+3、E+A+2配对组合而成的80对随机选择性扩增引物用于优化后的MSAP反应体系,对毛白杨Pt-1、Pt-2及其种内杂交子代P×tomentosa-151的CCGG位点进行标记,参照 Zhao X X[12]、Xiong L Z[13]、Fang J G[20]等的方法将毛白杨的CCGG胞嘧啶状态分为4种:非甲基化状态(带型(1,1))、外侧胞嘧啶半甲基化模式(CNG甲基化,带型为(1,0))、内侧胞嘧啶完全甲基化模式(CG甲基化,带型为(0,1))、不可确定CCGG甲基化模式或不可确定是否为CCGG位点(CG/CNG甲基化,带型为(0,0))。
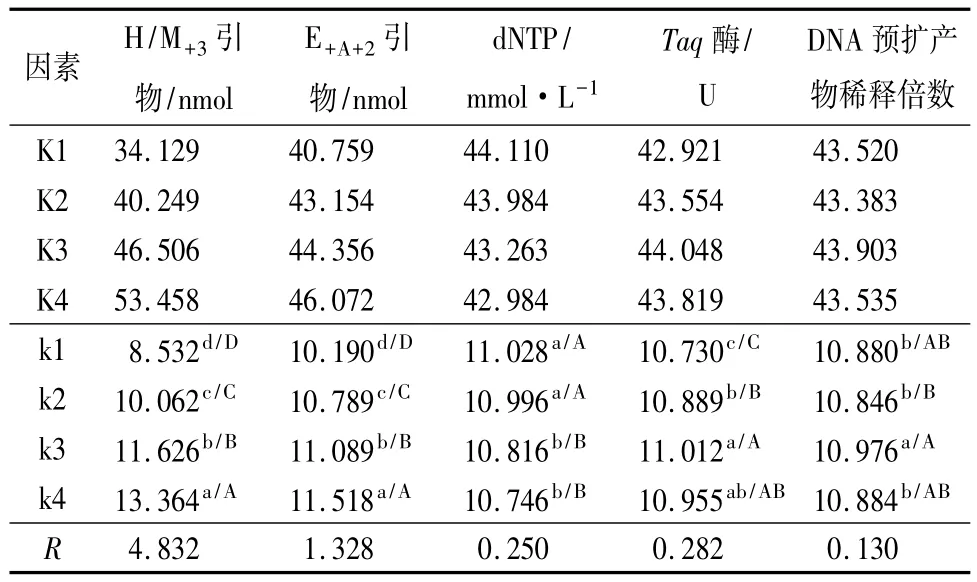
表3 L16(45)正交试验设计谱带品质多重比较
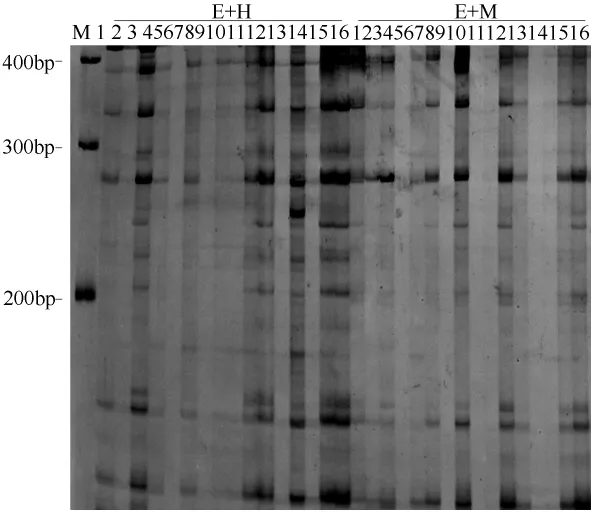
图2 选择性扩增产物变性聚丙烯酰胺凝胶电泳图
实验共筛选出32对具有良好扩增多态性的引物,检测到6种类型63种状态2314条谱带(表5),其中:A型,亲子代间CCGG甲基化状态保持完全一致的谱带为191条(占8.25%);B型,子代的CCGG胞嘧啶甲基化程度比父母本均高的谱带为100条(占4.32%);C型,子代的CCGG胞嘧啶甲基化程度比亲本均低,或低于亲本之一的谱带为328条(占14.17%);D型,子代的CCGG位点胞嘧啶发生超甲基化的谱带为308条(占13.31%);E型,亲本中有甲基化发生,子代的CCGG胞嘧啶完全去甲基化的谱带共397条(占17.16%);F型,亲本与子代CCGG位点胞嘧啶均非甲基化的谱带990条(占42.78%)。
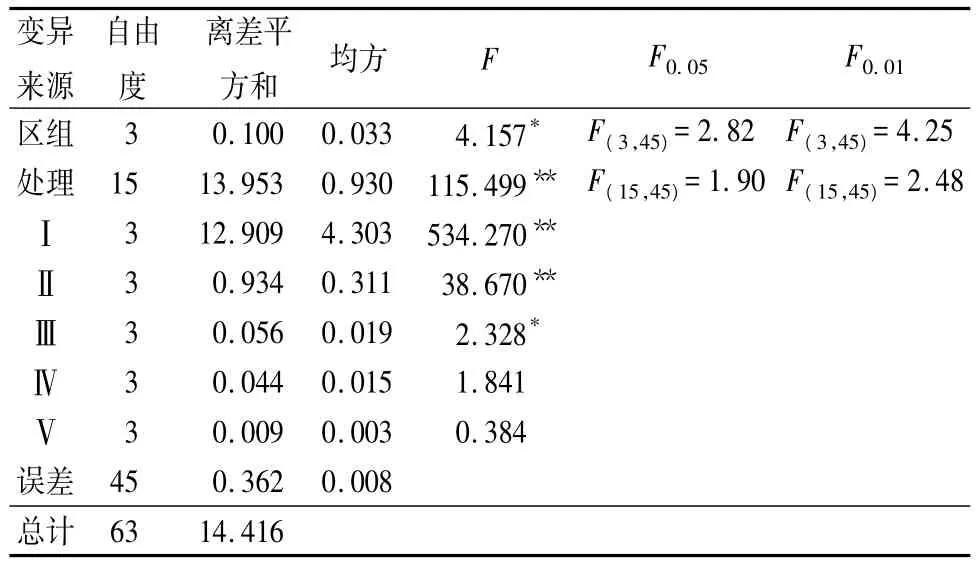
表4 L16(45)正交试验设计谱带品质方差分析
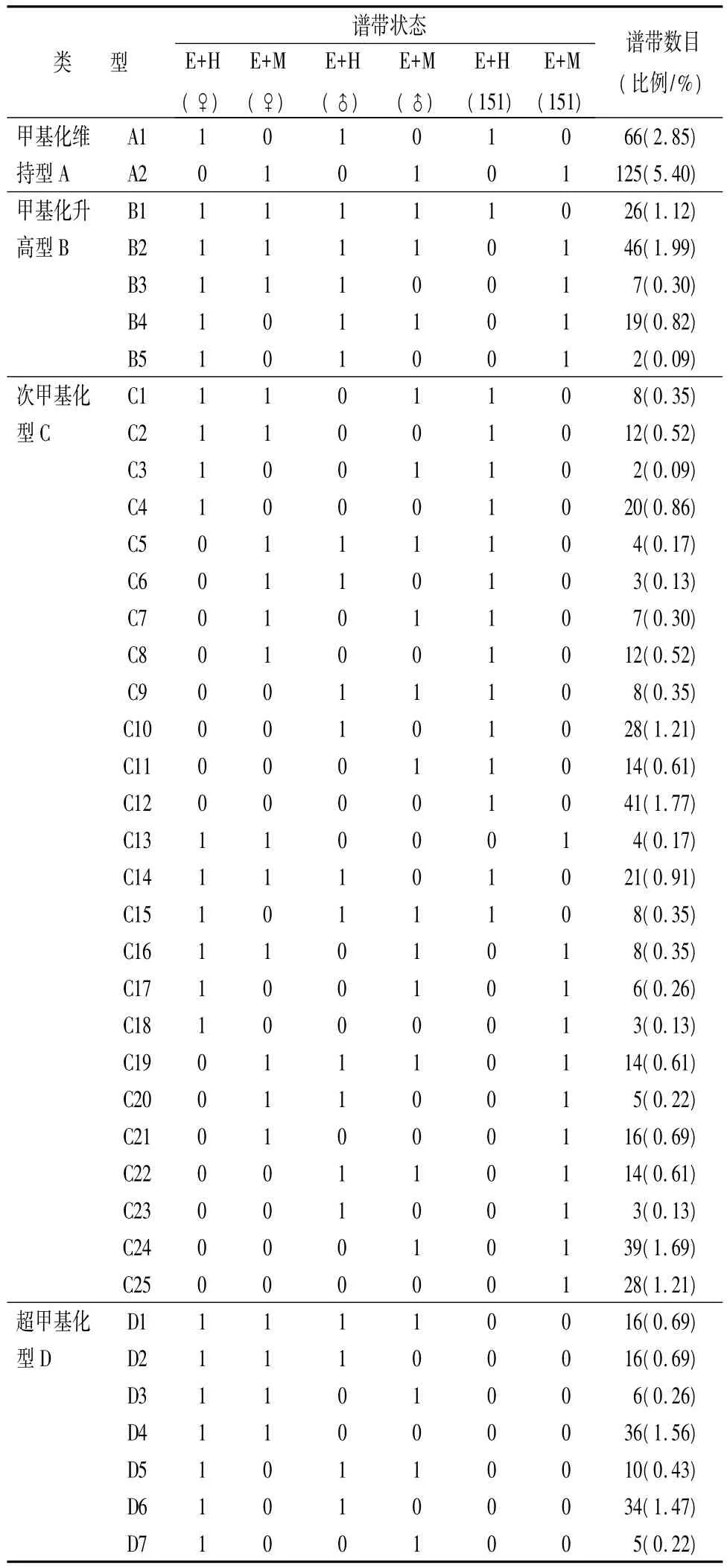
表5 毛白杨亲子代间CCGG胞嘧啶甲基化谱带类型及位点数
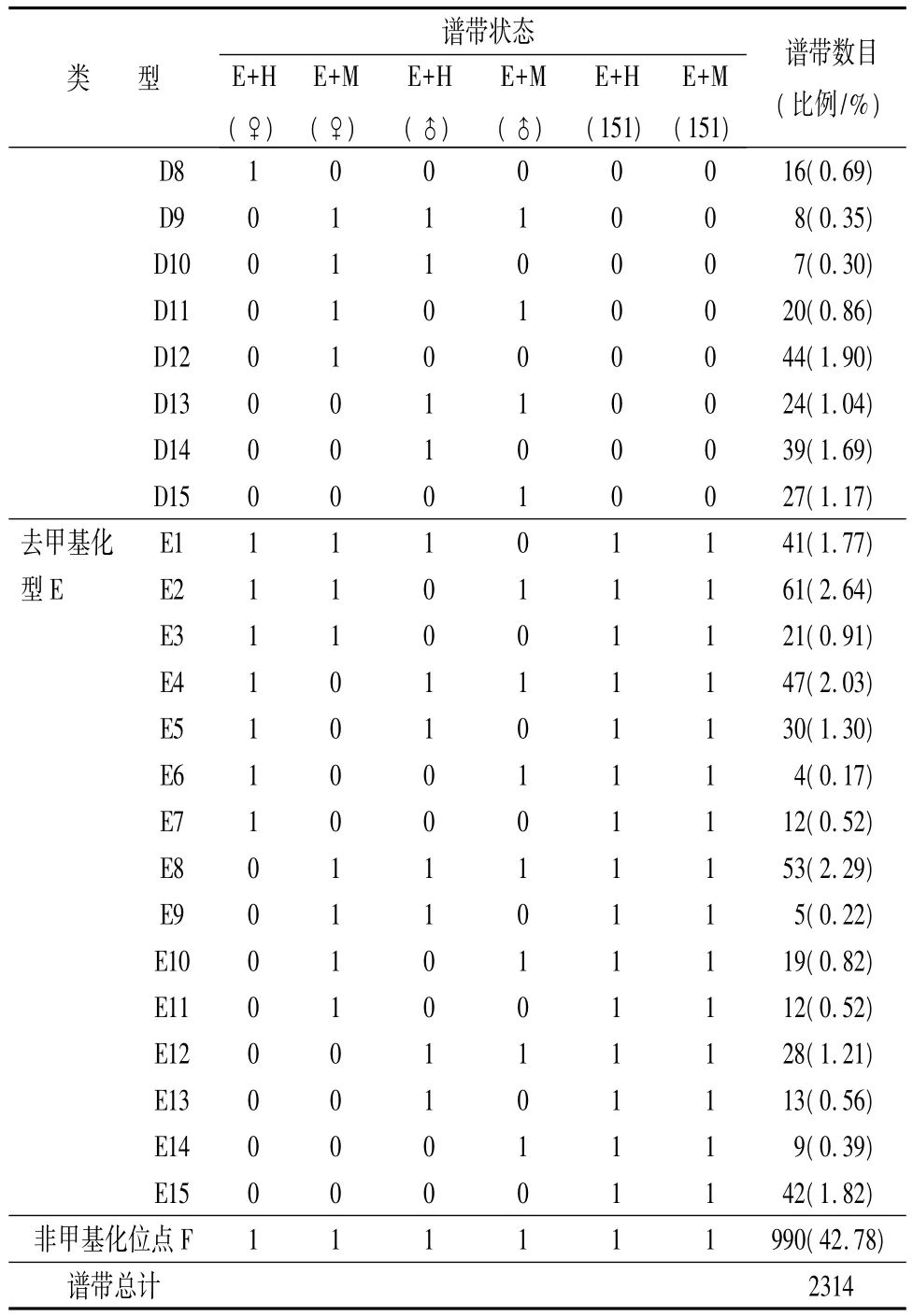
表5 (续)
2.2.2 毛白杨亲本及子代的CCGG甲基化相对水平
毛白杨Pt-1、Pt-2及其子代P×tomentosa-151的CCGG位点非甲基化相对水平约为56.83%~59.94%,总甲基化相对水平约为26.75%~29.39%,其中CG甲基化相对水平为14.65%~15.56%,高于CNG甲基化 12.10%~13.83%的相对水平。CG/CNG甲基化相对水平为13.31%~15.43%,介于CG甲基化、CNG甲基化相对水平之间(图3)。可以看出,毛白杨的CCGG甲基化相对水平低于非甲基化相对水平,CG甲基化相对水平高于CNG甲基化相对水平。由图3可知,母本的CCGG非甲基化相对水平高于父本的非甲基化相对水平,而母本的CCGG甲基化相对水平低于父本的CCGG甲基化相对水平。子代P×tomentosa-151的CCGG非甲基化相对水平高于亲本的非甲基化相对水平,而其甲基化相对水平则低于亲本的甲基化相对水平。其中子代CNG甲基化相对水平为12.10%,低于亲本12.27%、13.83%的相对水平;子代CG甲基化相对水平为14.65%,低于亲本15.30%、15.56%的相对水平;子代CG/CNG甲基化相对水平为13.31%,低于亲本15.43%、13.79%的相对水平。
2.2.3 毛白杨亲子代间CCGG胞嘧啶甲基化模式的遗传变异
CCGG非甲基化位点的遗传可分为3种(表6):ID1指子代非甲基化位点来源于母本,非父本;ID2指子代非甲基化位点来源于父本,非母本;IE指非甲基化的CCGG位点来源于父本或母本,与亲本保持完全一致。ID1与ID2两种遗传模式位点数分别为 123(占5.32%)、128(占 5.53%),两者之比为1 ∶1(χ2=0.100(1)=3.841)。
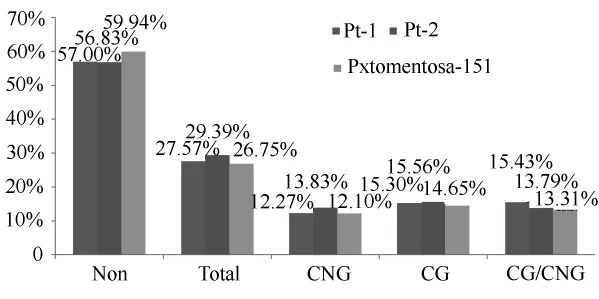
图3 毛白杨亲本与子代CCGG甲基化相对水平
CCGG胞嘧啶甲基化模式的遗传可表现为8种(表6):IA1指CG甲基化位点来自于母本,非父本;IA2指CNG甲基化来源于母本,非父本;IA3指CG/CNG甲基化位点来源于母本,非父本;IB1指CG甲基化位点来自于父本,非母本;IB2指CNG甲基化位点来源于父本,非母本;IB3指CG/CNG甲基化位点来源于父本,非母本;IC1指CG甲基化位点来源于父本或母本;IC2指CNG甲基化位点来源于父本或母本。结果显示,子代中共有547个CCGG甲基化位点(占23.64%)遗传自亲本,其中遗传自亲本的CG甲基化位点数为213(占9.20%),高于遗传自亲本的CNG甲基化位点数148(占6.40%),而遗传自亲本的CG/CNG甲基化位点数为186(占8.04%),介于CG、CNG甲基化位点数之间。子代中来源于母本的CCGG甲基化位点总数为155(占6.70%),来自于父本的CCGG甲基化位点数为201(占8.69%),可见子代中遗传自父本的甲基化位点数高于遗传自母本的甲基化位点数。
CCGG胞嘧啶甲基化模式的变异可分为7种,总数为2314(表6):VA1为亲本或亲本之一发生甲基化,子代中产生新的CG甲基化位点;VA2为亲本均无甲基化,子代中产生全新的CG甲基化位点;VB1指亲本或亲本之一发生甲基化,子代中产生新的CNG甲基化位点;VB2为亲本均无甲基化,子代中产生全新的CNG甲基化位点;VC1为亲本或亲本之一发生甲基化,子代中产生新的CG/CNG甲基化位点;VC2为亲本均无甲基化,子代中产生全新的CG/CNG甲基化位点;VD指亲本或亲本之一发生甲基化,而子代完全去甲基化,出现(1,1)带型。经检测,子代中有526个CCGG位点的胞嘧啶甲基化模式发生了变异,所占比例为22.73%。子代CCGG胞嘧啶甲基化模式变异产生的全新甲基化位点数(CG、CNG、CG/CNG甲基化位点数之和)与完全去甲基化位点数分别为88(占3.80%)、146(占6.31%),两者之比接近 1 ∶2(χ2=1.923(1)=3.841)。子代中产生的CG甲基化变异位点数与CNG甲基化变异位点数分别为126(占5.45%)、122(占5.27%),两者之比为 1 ∶1(χ2=0.065(1)=3.841)。
实验统计结果同时表明:子代P×tomentosa-151的CCGG胞嘧啶甲基化模式发生遗传变异的位点数分别为547(占 23.64%)、526(占22.73%),两者之比为 1 ∶1(χ2=0.411(1)=3.841)。
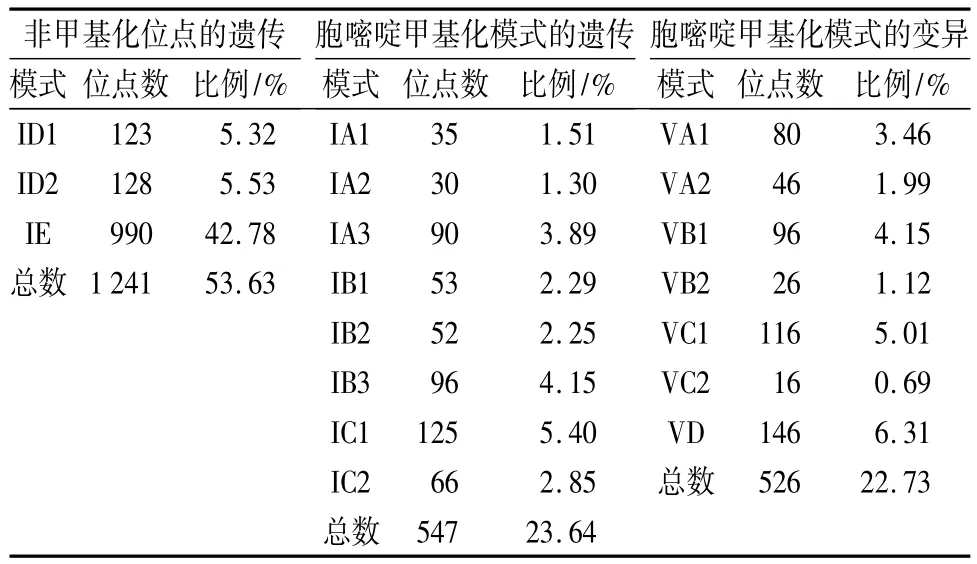
表6 毛白杨亲子代间CCGG胞嘧啶甲基化模式的遗传变异
3 讨论
3.1 毛白杨亲本与子代甲基化相对水平
本实验中由于基因组 DNA经 Eco RI+Hpa II、Eco RI+Msp I酶切后产生不同的带型,若产生(1,1)谱带,标记为非甲基化位点,若亲子代同时产生(0,0)类型,实验则无法识别,可能会造成甲基化位点统计量比实际甲基化位点数低的现象,有研究者认为该种状况发生频率较低[14-15]。因此,本文所涉及的甲基化水平均为可检测的甲基化位点的相对水平。实验表明,毛白杨亲本及子代的CCGG甲基化相对水平均低于非甲基化相对水平,CNG甲基化相对水平均低于CG甲基化相对水平。同时本文将亲本、子代甲基化相对水平进行了比较,认为子代的非甲基化CCGG位点的相对水平高于亲本的相对水平,CG、CNG、CG/CNG甲基化相对水平均低于亲本的相对水平。
DNA甲基化是已知的最早被发现的与基因抑制相关的表观遗传机制[26],那么不同的甲基化模式或水平则可能影响某些基因的表达调控。Kovarik研究基因CG和CNG的甲基化水平的结果表明所有物种的CG甲基化水平都较高,CNG甲基化水平则因物种而异[27],即甲基化模式与基因型可能具有相关性[20]。玉米基因组DNA甲基化过程中CNG甲基化相对水平低于CG甲基化相对水平[12],杂种DNA甲基化程度低于亲本,并且其甲基化程度与基因表达活性之间存在负相关[11]。Hauben等研究表明,油菜产量与 DNA甲基化水平呈负相关[17]。Zhao Y等则认为棉花子代甲基化水平比亲本低,但也有个别略微升高现象的存在[16]。在林木中,泡桐丛枝病的发生被认为与DNA甲基化水平的降低有关[28]。杉木杂交组合植株的CNG甲基化以及CG甲基化程度均低于自交组合,基因组甲基化程度越低杂种优势越明显,尤其是CNG甲基化与杂种优势呈显著性负相关[22]。然而黑杨中的研究则显示,在水分充足条件下,黑杨生长量与甲基化水平成正相关;在干旱胁迫下,DNA甲基化有可能参与生物体的生理调控[29]。本文试验材料,亲本株高与地径均小于子代,而其甲基化水平则高于子代。由于试验材料数量限制,该结论需要在群体中进行检验。
3.2 毛白杨子代甲基化模式的遗传变异
Holliday和Pogh于1975年做出CpG的甲基化和非甲基化模式能够在细胞分裂中可以像DNA序列本身一样被保留复制,也即在DNA复制完成时来自亲本的DNA链继续维持其被修饰的胞嘧啶模式的推论[30]。Bird 和 Southern[31]利用可以切割胞嘧啶非甲基化的CpG位点而不能切割胞嘧啶甲基化的CpG位点的酶,切割非洲爪蟾(Xenopus laevis)核糖体RNA基因非甲基化和甲基化位点并绘制出其模式图,认为在一条特定的DNA链上,大部分CpG是甲基化的,非甲基化位点随机出现,重要的是两条链对应的CpG位点要么全部甲基化,要么全部非甲基化,验证了Holliday等的推论。植物中大量研究表明,甲基化状态常常可以通过减数分裂稳定的遗传给后代[32-33],另有一部分发生变异,同时存在动态变化和组织特异性表达,即植物不同发育时期、不同组织、甚至在不同环境中DNA甲基化模式也不尽一致[12-14,16-17,20,28]。如天然分布于河畔与盐水沼泽的红树林,DNA甲基化的变异远大于基因型的差异[21]。本文以幼化[24-25]处理后的木本植物毛白杨亲本及子代同一时期、相同部位的叶片为材料,最大程度地消除亲本与子代间年龄效应、组织特异性等带来的误差,利用MSAP标记检测并分析了CCGG位点胞嘧啶甲基化模式的遗传变异,认为子代中发生遗传变异的位点数之比为1∶1。遗传自父本的甲基化位点数高于遗传自母本的位点数,可能与父本甲基化相对水平高有关,遗传自亲本的CG甲基化位点数高于遗传自亲本的CNG甲基化位点数。子代甲基化位点变异中产生的全新甲基化位点数与完全去甲基化位点数之比为1∶2,发生CG甲基化变异的位点数与发生CNG甲基化变异的位点数比例为1∶1。可见子代P×tomentosa-151的CCGG甲基化状态存在较大的变异,并且以去甲基化为主。Xiong L Z等[13]在研究亲本与其杂种子代的DNA甲基化差异对水稻杂种优势的影响时认为,特异位点上的甲基化的改变对杂种优势有显著效应。杉木子代中CCGG甲基化变异具有父母本效应,并且多发生在CG甲基化位点,可能与其杂种优势相关[22]。基于上述结论,本文初步推测毛白杨杂种优势的形成可能与DNA甲基化相对水平的降低或特异性位点的去甲基化相关,并认为研究毛白杨亲本与子代群体间DNA甲基化水平、遗传变异模式可能会进一步揭示毛白杨杂种优势形成的分子机制。
[1]Santi D V,Garrentt C E,Barr P J.On the mechanism of inhibition of DNA-cytosine methyltransferases by cytosine analogs[J].Cell,1983,33(1):9-10.
[2]Bender J.DNA methylation and epigenetics[J].Annual Review of Plant Biology,2004,55:41-68.
[3]Rassoulzadegan M,Grandjean V,Gounon P,et al.RNA-mediated non-mendelian inheritance of an epigenetic change in the mouse[J].Nature,2006,441:469-474.
[4]Gruenbaum Y,Naveh Many T,Cedar H,et al.Sequence specificity of methylation in higher plant DNA[J].Nature,1981,292:860-862.
[5]Richards E J.DNA methylation and plant development[J].Trends in Genetics,1997,13:319-323.
[6]Finnegan E J,Genger R K,Peacock WJ,et al.DNA methylation in plants[J].Annual Review of Plant Physiology and Plant Molecular Biology,1998,49:223-247.
[7]洪柳,邓秀新.应用MSAP技术对脐橙品种进行DNA甲基化分析[J].中国农业科学,2005,38(11):2301-2307.
[8]Henikoff S,Matzke M A.Exploring and explaining epigenetic effects[J].Trends Genet,1997,13(8):293-295.
[9]Santos K F,Mazzola T N,Carvalho H F,et al.The prima donna of epigenetics:the regulation of gene expression by DNA methylation[J].Brazilian JMedical Biol Res,2005,38:1531-1541.
[10]Reyna-López G E,Simpson J,Ruiz-Herrera J.Differences in DNA methylation patterns are detectable during the dimorphic transition of fungi by amplification of restriction polymorphisms[J].Mol Gen Genet,1997,253:703-710.
[11]Tsaftaris A S,Kafka M,Polidoros A,et al.Epigenetic changes in maize DNA and heterosis[C]//The genetics and exploitation of heterosis in crop.Mexico:Abst Int Symp,1997:112-113.
[12]Zhao X X,Chai Y,Liu B.Epigenetic inheritance and variation of DNA methylation level and pattern in maize intra-specific hybrids[J].Plant Science,2007,172(5):930-938.
[13]Xiong L Z,Xu C G,Maroof M A,et al.Patterns of cytosine methylation in an elite hybrid and its parental lines,detected by a methylation-sensitive amplification polymorphism technique[J].Mol Gen Gent,1999,261(3):439-446.
[14]Shikawa I.Surveying CpG methylation at 5’-CCGG in the genomes of rice cultivars[J].Plant Mol Biol,2001,45:31-39.
[15]Liu B,Brubaker C L,Mergeai G,et al.Polyploid formation in cotton is not accompanied by rapid genomic changes[J].Genome,2001,44:321-330.
[16]Zhao Y,Yu S,Xing C,et al.Analysis of DNA methylation in cotton hybrids and their parents[J].Molecular Biology,2008,42(2):169-178.
[17]Hauben M,Haesendonckx B,Standaert E,et al.Energy use efficiency is characterized by an epigenetic component that can be directed through artificial selection to increase yield[J].Proc Natl Acad Sci USA,2009,106(47):20109-20114.
[18]魏华丽,吴涛,杨文华,等.落叶松体细胞胚胎发生过程中DNA甲基化模式变化分析[J].东北林业大学学报,2011,39(2):33-37.
[19]Xu M L,Li X Q,Schuyler S.AFLP-based detection of DNA methylation plant[J].Molecular Biology Reporter,2000,18:361-368.
[20]Fang JG,Song CN,Qian JL,et al.Variation of cytosine methylation in 57 sweet orange cultivars[J].Acta Physiol Plant,2010,32:1023-1030.
[21]Lira Medeiros CF,Parisod C,Fernandes R A,et al.Epigenetic variation in mangrove plants occurring in contrasting natural environment[J].Plos One,2010,5(4):10326.
[22]洪舟,施季森,郑仁华,等.杉木亲本自交系及其杂交种DNA甲基化和表观遗传变异[J].分子植物育种,2009,7(3):591-598.
[23]张志毅,朱之悌,Müller-Starck G,等.毛白杨无性系同工酶基因标记的研究[J].北京林业大学学报,1992,14(3):9-18.
[24]Schier G A,Campbell R B.Differences among Populus species in ability to form adventitious shoots and roots[J].Can For Res,1976,6:253-261.
[25]朱之悌,张志毅,赵勇刚.毛白杨优树快速繁殖方法的研究[J].北京林业大学学报,1986,4:1-7.
[26]Razin A,Riggs A D.DNA methylation and gene function[J].Science,1980,210:604-610.
[27]Kovarik A,Matyasek R,Leitch A,et al.Variability in CpNpG methylation in higher plant genomes[J].Gene,1997,204(1/2):25-33.
[28]黎明,翟晓巧,范国强,等.土霉素对豫杂一号泡桐丛枝病幼苗形态和DNA甲基化水平的影响[J].林业科学,2008,44(9):152-156.
[29]Gourcilleau D,Bogeat Triboulot M B,Thiec D L,et al.DNA methylation and histone acetylation:genotypic variations in hybrid poplars,impact of water deficit and relationships with productivity[J].Ann For Sci,2010,67:208.
[30]Holliday R,Pugh J E.DNA modification mechanisms and gene activity during development[J].Science,1975,186:226-232.
[31]Bird A P,Southern E M.Use of restriction enzymes to study eukaryotic DNA methylation.I.the methylation pattern in ribosomal DNA from Xenopus laevis[J].JMol Boil,1978,118:27-47.
[32]Cubas P,Vincent C,Coen E.An epigenetic mutation responsible for natural variation in floral symmetry[J].Nature,1999,401:157-161.
[33]Kiddle N C,Richards E J.The control of natural variation in cytosine methylation in Arabidopsis[J].Genetics,2002,162:355-363.
