猪巨细胞病毒gB基因的克隆与序列分析
2012-08-08刘兴彩尹燕博徐守振李吉达郭妍妍
刘兴彩,尹燕博,徐守振,李吉达,张 毅,郭妍妍
(1.青岛农业大学动物科技学院,山东 青岛 266109;2.中国农业大学动物医学院,北京 海淀 100193;3.青岛澳兰百特生物工程有限公司,山东 青岛 266101)
猪巨细胞病毒(porcine cytomegalovirus,PCMV)属于疱疹病毒科,β疱疹病毒亚科、巨细胞病毒(cytomegalovirus,CMV)属的成员;近来有人认为将本病毒划在玫瑰疹病毒属中更为恰当[1]。其gB蛋白是CMV重要跨膜糖蛋白,在病毒与机体细胞膜的粘附、融合及病毒进入细胞和在细胞间的转移起作用[2];gB蛋白具有较好的免疫原性[3],可诱导机体产生抗CMV的特异性免疫应答[4-5]。我们应用PCR诊断方法对全国11个省市地区猪场的100份临床样品进行了检测[6],筛选出4份PCR阳性病料,进行了gB基因的克隆与序列分析,为PCMV的分类地位和进化关系提供更多的信息。
1 材料与方法
1.1 病料 来自郑州、福建、浙江金华和宁波病猪的肺脏。
1.2 菌株和载体 感受态细胞DH5α和pMD 18-T载体。
1.3 主要仪器和试剂 PCR扩增仪(LongGene MG96G),琼脂糖(美国Promega公司),Taq DNA聚合酶(5U/μL)、dNTPs(25μmol/L)为宝生物工程(大连)有限公司产品,其他化学试剂均为国产分析纯级。
1.4 引物 根据GenBank发表的PCMV AF268041序列,应用Primer Premier 5.0设计了PCMV gB全基因引物 g1:5′-ATGACAGTGAGCAGTCGGAA-3′;g2:5′-TCACACGTCCTCGGTGGATAG-3′,并由宝生物工程(大连)有限公司合成。
1.5 DNA的提取 按照文献[6]中的提取方法进行提取。
1.6 gB基因PCR扩增 取出已分装有PCR反应体系的离心管,加入2μL上述模板,混匀后稍离心,置PCR 仪中扩增:95℃ 5 min,94℃ 30s,62℃30s,72℃2min,30个循环,72℃ 10min.取 PCR产物5μL在10g/L琼脂糖凝胶上电泳,置凝胶成像系统中观察结果。
1.7 阳性克隆的鉴定与测序 回收PCR产物大小为2580bp,按照连接试剂盒说明书进行连接,然后将连接产物转化到感受态细胞DH5α中,涂平板蓝白斑筛选,将白色菌落接种到LB液体培养基中,置37℃剧烈震荡培养12h后,进行PCR鉴定,将阳性菌株送宝生物工程(大连)有限公司测序。
1.8 序列分析 应用DNAStar软件将测序结果与其他PCMV核苷酸和推测的氨基酸序列进行同源性分析,与疱疹病毒各亚科gB糖蛋白进行氨基酸序列分析,并与人疱疹病毒6型和7型(HHV-6和HHV-7)gB糖蛋白进行氨基酸序列比对,并构建进化树;应用在线分析软件TMHMM,ProtParam,Cysteine Connectivity Prediction Server和Prosite分别对gB糖蛋白进行跨膜预测,疏水性分析,半胱氨酸分析和潜在的N-糖基化位点分析。
2 结果
2.1 gB基因的PCR扩增结果 从4个不同来源的PCMV PCR阳性病料中,用PCMV gB全基因的特异性引物分别扩增出4个长度约为2580bp的条带,与设计的长度一致(图1)。
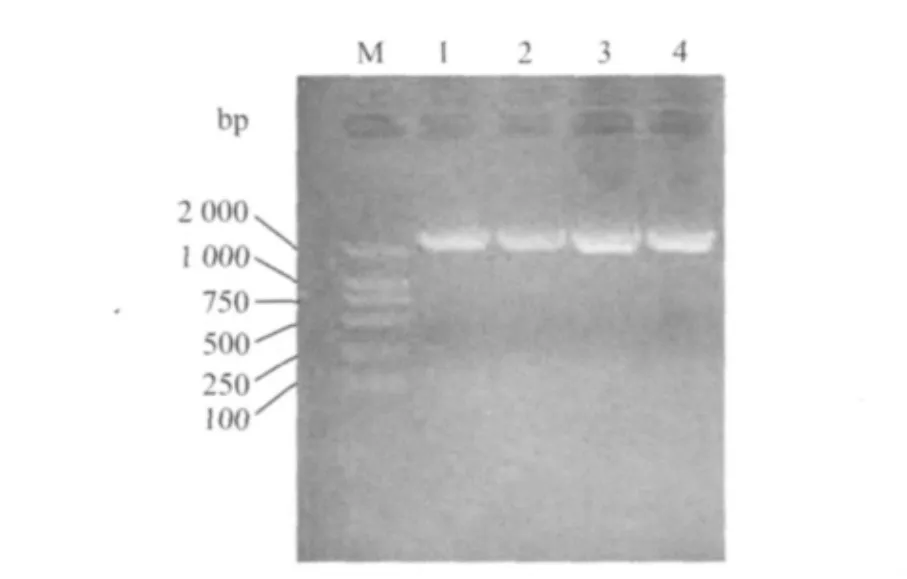
图1 不同地区gB基因的PCR产物
2.2 PCMV gB基因序列同源性分析 对上述PCR产物进行了PCMV gB基因序列测定,获得4个 PCMV gB 基 因 序 列 (NB200803,FJ200809,JH200810和ZZ200811),序列分析发现他们的同源性在98.1%~99.6%,差异性在0.3%~2.0%,与其他PCMV gB基因序列同源性在96.1%~99.7%,差异性在0.3%~4.1%,结果如图2。

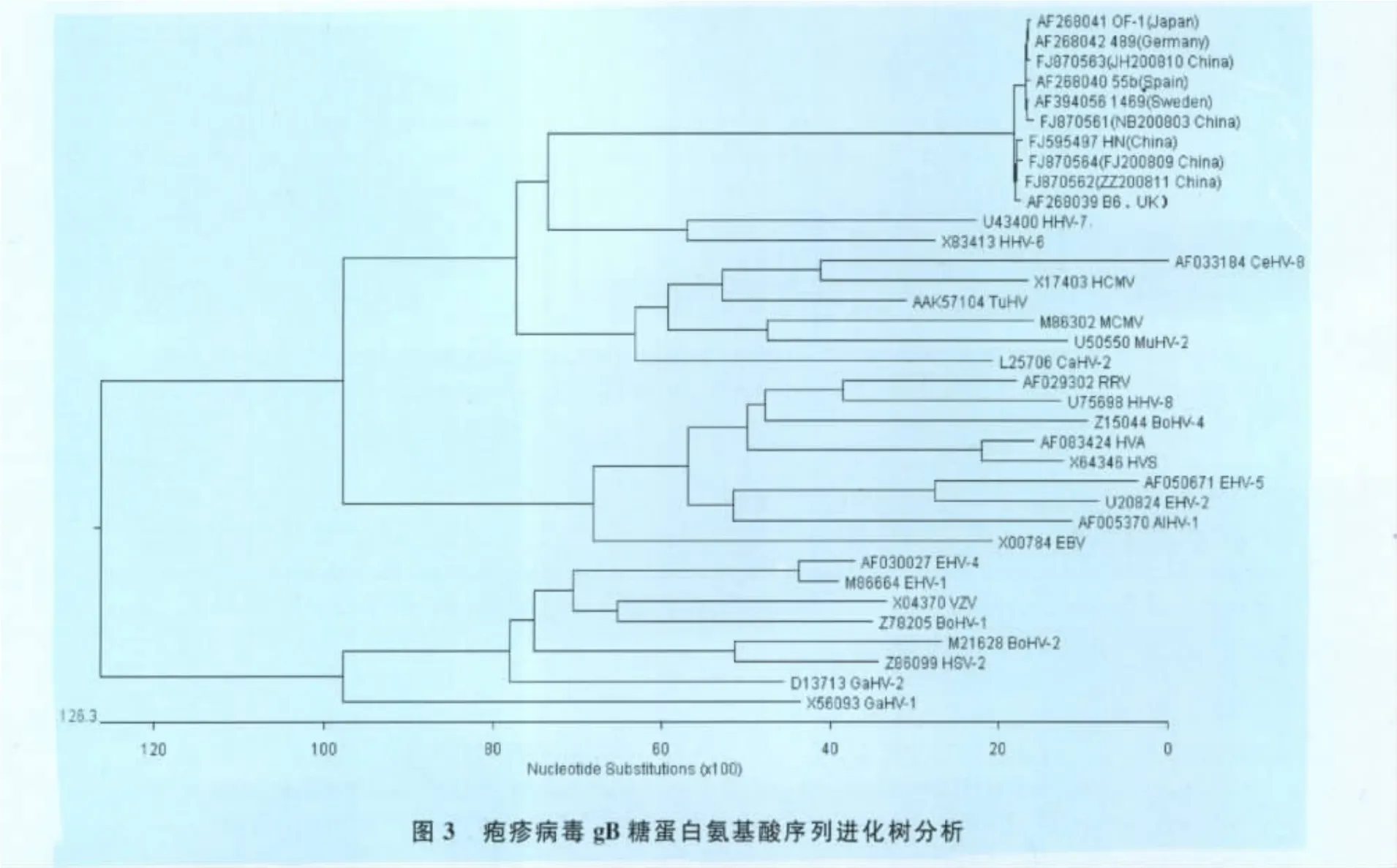
2.3 系统进化树分析 遗传进化分析发现,整个疱疹病毒属明显的分为3个亚科,而我们所获得的来自4个不同地区的PCMV gB序列(图中●所示)均属于β疱疹病毒亚科,在β疱疹病毒亚科中,HHV-6和HHV-7与PCMV各株位于同一分支。另外,我们还发现PCMV推导的gB基因氨基酸序列出现分支,如图3。
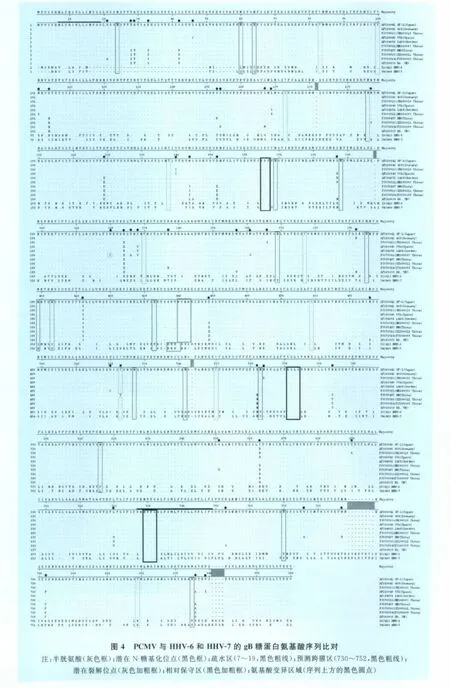
2.4 PCMV gB糖蛋白氨基酸序列与其他PCMV以及人疱疹病毒6型和7型gB糖蛋白氨基酸序列比较 推导的gB糖蛋白氨基酸序列有11个与二硫键形成有关的半胱氨酸(除NB200803在第320位:W→C和HN,湖南序列在第522位:C→Y),其中与HHV-6和HHV-7相比,共有9个潜在保守的半胱氨酸;潜在 N-糖基化位点(Asn-X-Ser or Thr)有17个,其中ZZ200811在196~198氨基酸处出现NNT→NYT,JH200810在563~565氨基酸处出现NNS→NDS以及ZZ200811、FJ200809、HN和55b(AF268040,Spain)在835~837氨基酸处出现NTT→NDS糖基化位点变异;在其氨基端包含一个疏水区(7~19),预测其为一个信号肽区域,应用Prot Param分析蛋白的亲水性,得到的亲水性最大平均数均为负值,说明具有很强的疏水性,蛋白跨膜预测发现跨膜区序列(730~752),膜外区(1~729)和膜内区(753~860),其跨膜区序列可能为跨膜锚定序列,与gB糖蛋白在膜上的锚定作用有关;潜在裂解位点是RYKR(439~442),无连续性碱性氨基酸插入,具有低致病性病毒分子特征。HHV-6和HHV-7的裂解位点分别是RRRR(396~399)和RKRR(393~396),它们均符合共有基序R-X-R/K-R规律。
从图4可以很明显的看出,黑色圆点比较密集有3个区域:第21~46位、第241~253位和第322~339位,这3个区域,变异比较集中,我们将其定为高变区。
3 讨论
本试验对来自4个不同地区的PCMV PCR阳性病料gB基因进行了克隆与序列分析,对推导的氨基酸进行了半胱氨酸、潜在N-糖基化位点、裂解位点及疏水性和跨膜分析,发现gB基因所编码的gB糖蛋白这些生物特性类似于其他疱疹病毒gB糖蛋白[7]。此外,发现在疱疹病毒属中,PCMV gB基因序列与HHV-6和HHV-7的ORFs 36~40序列同源性最高,如图3所示,包括我们测序的4个样本在内的PCMV毒株,与HHV-6和HHV-7同属一个分支,说明它们之间具有较近的生物进化关系,但两者又处在不同的两个群,说明两者存在差异。有报道称,由于它们的细胞嗜性和细胞内DNA新陈代谢的显著差异,而导致其生物进化关系复杂而又矛盾[8-9],PCMV与HHV-6和HHV-7具有许多相似的分子生物学特性,它们的不同之处可能源于宿主细胞的选择压力[10]。
序列分析表明,我们测得的4个PCMV gB基因序列同源性在98.1%~99.6%,差异性在0.3%~2.0%,推导的氨基酸序列同源性在97.0%~99.3%,差异性在0.7%~3.1%,相对而言其序列变异不大。与其他PCMV gB基因序列同源性在96.1%~99.7%,差异性在0.3%~4.1%,差异性偏大的原因主要来源于湖南病料的PCMV gB基因序列。推导的氨基酸同源性在96.5%~99.7%,差异性在0.3%~3.1%,与β疱疹病毒亚科中其他常见病毒的同源性在25.9%~38.1%,由此说明CMV不同种属间同源性较低。另外,我们发现在第21~46位这个高变区,HN(湖南)、ZZ200811、FJ200809和B6株(英国)的第33位由P→S,第42位由S→P,这说明它们之间发生了置换;第28位和29位由TA→ST,这是导致PCMV出现两个分支的原因之一。该分支的出现说明其gB基因变异越来越明显,并朝着两个方向变异,而且该变异呈地域化发展的趋势。
[1]陆承平.兽医微生物学[M].3版.北京:中国农业出版社,2001:472-477.
[2]McGregor A,Liu F,Schleiss M R.Molecular,biological,and In vivo characterization of the guinea pig cytomegalovirus(CMV)homologs of the human CMV matrix proteins pp71(UL82)and pp65 (UL83)[J].J Virol,2004,78(9):9872-9889.
[3]Mitchell D K,Holmes S J,Burke R L,et al.Immunogenicity of a recombinant human cytomegalovirus gB vaccine in seronegative todders[J].Pediatr Infect Dis J,2002,21:133-138.
[4]Mocareki E S Jr,Kemble G W.Recombinant cytomegalovirus for study of replication and pathogenisis[J].Interviology,1996,39(5-6):320-330.
[5]Britt W J,Mach M.Human cytomegalovirus glycoproteins[J].Intervirology,1996,39(5-6):401-412.
[6]刘兴彩,尹燕博,张毅,等.猪巨细胞病毒PCR诊断技术的建立[J].中国兽医杂志,2009,45(12):34-35.
[7]Goltz M,Widen F,Banks M,et al.Characterization of the DNA polymerase loci of porcine cytomegaloviruses from diverse geographic origins[J].Virus Genes,2000,21:249-255.
[8]Frenkel N,Roffman E.Fields Virology[M].3rd ed.Philadelphia:Lippincott-Raven Publishers,1996:2609-2622.
[9]Pellett P E,Black J B.Fields Virology[M].3rd ed.Philadelphia:Lippincott-Raven Publishers,1996:2587-2608.
[10]Frederik Widen,Michael Goltz,Wittenbrink N,et al.Identification and sequence anaktsus of the glycoprotein B gene of porcine cytomegalovirus[J].Virus Gene,2001,23(3):339-346.
