基于Repast的细菌趋化性仿真模型研究
2012-07-13郭雪清宋莉莉
郭雪清,宋莉莉
(广州军区武汉总医院 信息科,湖北 武汉 430070)
近年来,实验研究显示单细胞生物分子的随机交互与其行为变化存在关联,因此即使是基因完全相同的细胞也可能表现出不同行为[1]。这些研究表明,通过信号进行传递的细胞分子之间的随机事件对单个细胞的行为具有重要意义。而现有大部分计算模型一般仅刻画了生物系统某个方面的特性,如分子组成,分子间相互作用,以及细胞行为等,细胞的群体特性往往采用微分方程进行描述[2-3]。这些方法都不不足以反应细菌随机运动方式和行为随外界情况变化而产生的适应性改变,而基于Agent的建模仿真方法能够比较有效地解决这一问题。
文中对如何采用基于Agent的建模仿真方法来研究这一特性进行了初步探讨。基于Repast仿真平台,建立Agent仿真模型,对细胞内部的生物化学过程进行建模,同时将其运动过程(行为)在三维环境中进行显示。为了对该方法进行演示,文中以细菌趋化仿真模型为例进行详细说明。
1 Agent建模仿真平台Repast
Agent建模方法起源于复杂适应系统 (Complex Adaptive System,CAS)理论,其基本思想是:CAS的复杂性起源于主体的适应性,主体与环境以及与其它主体之间的相互作用,不断改变着它们的自身,同时也改变着环境,各个主体通过相互间交互,可以在整体层次上突现出新的结构、现象和更复杂的行为。基于Agent的建模与仿真方法已成为研究CAS最具活力和影响力的方法之一。
Repast是目前应用最广泛的、免费、开源的Agent建模与仿真工具包。Repast最早由美国芝加哥大学以及Argonne国家重点实验室的研究人员开发,现在由非盈利性组织ROAD(Repast Organization for Architecture and Development)负责维护。其最初的设计目标是提供一个易于使用和扩展且功能强大的社会系统仿真工具包,随着不断的发展和完善,现在已经成为一个成熟通用的多Agent仿真平台[4-5]。Repast系统体系结构如图1所示[6]。
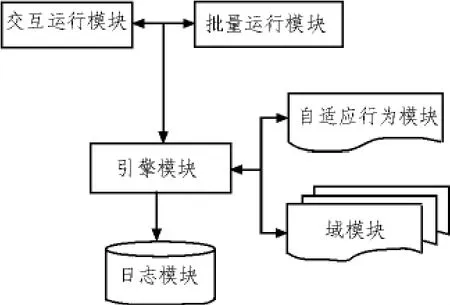
图1 Repast系统体系结构图Fig.1 Architecture of Repast
Repast系统体系结构功能非常强大,主要由一系列模块组成,包括引擎模块 (Engine Module)、日志模块(Logging Module)、交互运行模块(Interactive Module)、批量运行模块(Batch Run Module)、 自适应行为模块 (Adaptive Behaviors Module)和域模块(Domains Module)6个部分。这些模块可分为固定模块和可变模块两类。
引擎模块主要用于控制仿真中的活动状态,它包括引擎控制器、程序调度器、发生器和Agent组件。引擎控制器与交互运行模块和批量运行模块一起工作,实现仿真运行中的初始化、开始、暂停、步长、停止和重新启动功能。引擎模块是一个固定模块。
记录模块负责记录仿真结果,根据输入的复杂性又分为数据记录器和对象记录器两种类型的记录组件。记录模块是一个固定模块。
交互运行模块是一个固定模块,主要协助用户对仿真运行过程进行控制和管理,在用户和引擎模块之间起中介作用,主要包括接口控制器和可视化环境等组件。
批量运行模块是一个固定模块,主要实现在不需要用户干涉的情况下就能批量完成一系列仿真运行计算,由参数扫描框架和批量控制器两个组件构成,模型的参数组合通过参数扫描框架传递给批量控制器,每个批量控制器在仿真运行过程中管理一个引擎控制器。
自适应行为模块是一个可变模块,如果Agent需要完成相应的行为(如学习和适应行为),就可以为此提供相应的组件,如遗传算法、神经网络等。
域模块也是一个可变模块,主要提供一些特殊功能,有助于完善模型功能,如普通网络、社会系统、地理信息系统等工具。
目前,Repast已经发展到Repast3,提供了3种语言版本:Repast for Java (Repast J),Repast for Microsoft.NET(Repast.NET)和 Repast for Python Scripting(Repast Py)。 2007 又发布了新型的可视化仿真平台 Repat Simphony(Repast S),文中将采用Repast S来设计和开发细菌趋化仿真模型。
2 细菌趋化模型
考虑到生物学中细菌趋化性的典型信息处理过程[7],文中以埃希氏大肠杆菌为例,仅考虑细菌与其所处的化学环境进行交互,对其趋化行为进行仿真。该细菌趋化模型采用多Agent方法建立,每个细菌都被视为一个具有独立的化学向性网络、发动机和鞭毛的Agent,它可以在一个三维环境中自由移动。通过该模型就可以获得单个细菌和细菌群的趋化性试验数据。在该模型中,外部环境的信号通过一个趋化性网络被转换为细胞内部的信号。通过该信号就可以影响细胞发动机的状态在顺时针和逆时针之间发生转换。发动机逆时针旋转时,鞭毛呈集中状态,细胞平稳运动,当发动机顺时针旋转时,鞭毛呈分离状态,细胞则不规律的翻滚从而进行随机运动。具体算法如下:
1)趋化性网络算法
趋化性网络模型采用Morton-Firth[7]和 Korobkova[1]等人提出的模型。该模型以细胞占有的感知器作为输入,以细胞内做出反应调整的分子数目作为输出。
2)感知器绑定算法
假定感知器感知到化学配合基的概率与感知器的状态有关。非激活感知器感知到化学配合基的概率为q=[L]/([L]+KD),其中[L]为化学配合基的浓度,KD是分裂常量。激活的感知器感知到化学配合基的概率与非激活感知器相同,但是常量则相对较大。
3)发动机状态转换算法
发动机的行为是非线性的,其旋转状态由细胞内部的分子数目决定。仿真初始时会定义一个标准分子数目水平,当过去0.3 s内的平均分子数目值超过标准值时,发动机顺时针旋转,否则发动机逆时针旋转,发动机的状态必须保持一个最小时间才能发生改变。
4)鞭毛状态转换算法
这里定义了两类鞭毛状态转换算法。一种是精确的根据发动机的状态来改变自身状态。另外一种算法中,鞭毛的状态从集中转换到分离时有20%的几率可能失败。
5)运动算法
按照 Berg和 Brown(1972)的假定[3],在一次运行中,细胞的速度是不发生变化的,而仅仅是方向发生变化,从而影响位置的变化,每一时间片都通过速度、方向与上一时刻的位置来更新当前位置。当细胞进行翻滚时,细胞的位置是不发生变化的,新方向与原方向的夹角α服从形状参数为4、范围参数为18.32以及位置参数为-4.6的伽玛分布。
3 细菌趋化仿真模型的Repast实现
3.1 模型分析
细菌趋化模型主要考虑细胞在环境中对化学物质的一种趋向性运动现象,在该问题中只存在细胞这一种Agent。而其他细胞的组成部分如发动机、鞭毛等都作为细胞Agent的组成类而不是作为单独的Agent实现。
由于细胞可以在三维空间中自由的移动,所以可以采用连续的三维空间来表示细胞Agent的空间环境。
根据细菌趋化模型的描述和假设可以知道,细胞除了自身的位置外,其属性还包括速度、运动方向、细胞内做出反应调整的分子数目、发动机的状态以及鞭毛的状态。细胞Agent根据上一时刻的位置、速度以及运动方向运动到新的位置,然后使用感知器感知当前位置的化学物质浓度,根据细胞内对环境做出反应调整的分子数目来影响改变发动机的状态和鞭毛的状态从而影响细胞的运动方向,然后再次改变位置,周而复始。细菌趋化模型中,细胞只由环境中的化学物质浓度而改变状态,所以不与其他Agent进行交互。
3.2 模型设计
根据前面细菌趋化问题的模型分析,可以确定相应的Agent类型和行为。基于RepastS开发细菌趋化模型的具体组成包、类以及它们之间的依赖关系如图2所示。主要的类和接口包括:
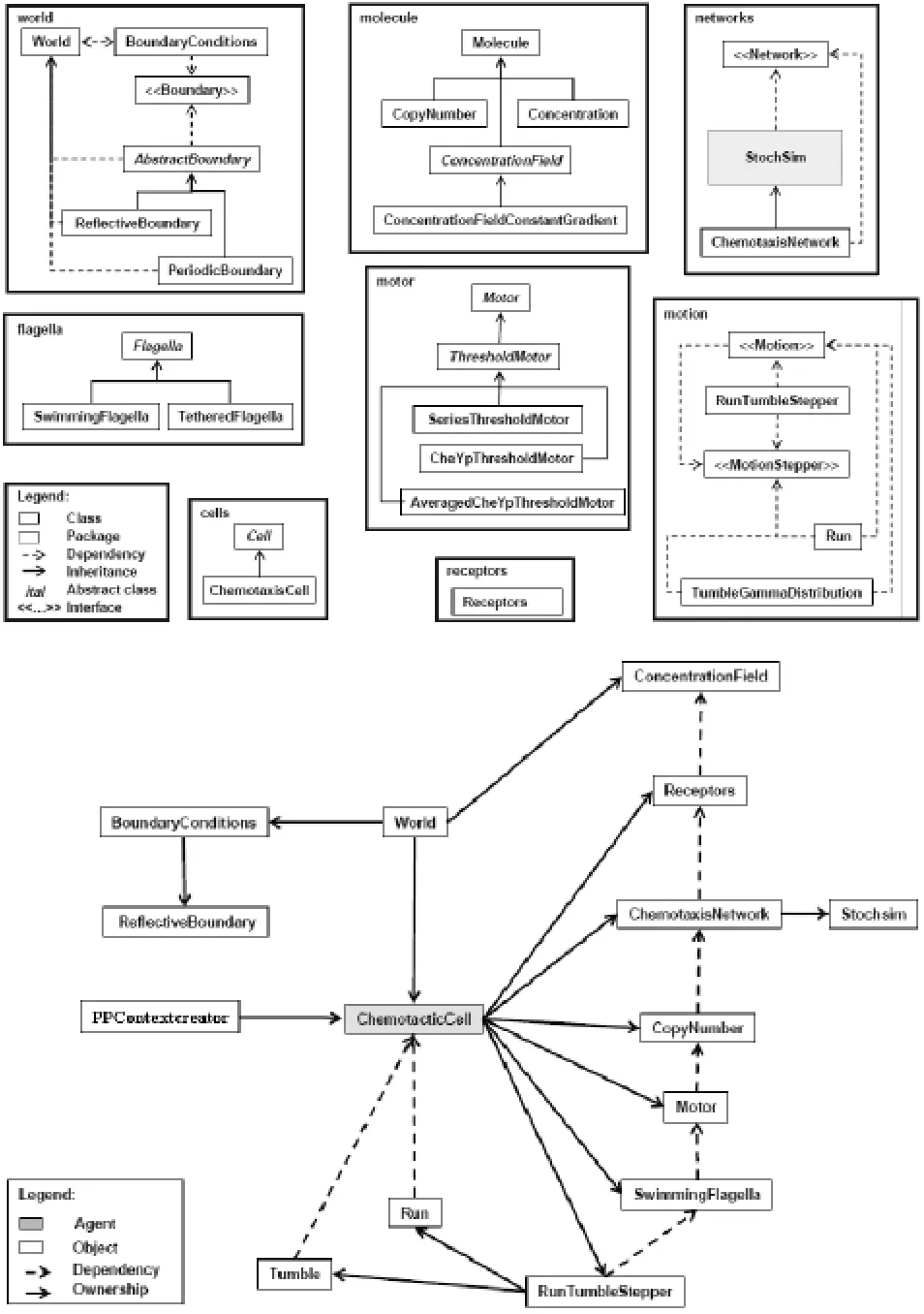
图2 细菌趋化模型类结构及关系图Fig.2 Bacterial chemotaxis class structure and its relationship
1)World类 代表了Agent所处的环境。World类包含了一系列的细胞Cell类和边界BoundaryConditions类。
2)Cell类 Cell类是一个抽象的Agent对象类,缺省提供了位置、方向、体积。ChemotaxisCell类继承了Cell类并加入了新的属性:Receptors、Network、Motor和 Flagella 类的对象。
3)Receptor类 该类提供了从环境中读取化学配合基浓度的方法。
4)NetWork接口 所有的趋化网络都要使用该接口。该接口提供了方法来设置和获取分子个数,并且也提供了step函数来推进整个网络的状态更新。
5)Motor类 该类是一个抽象类,它保存了两个状态:顺时针和逆时针,并提供了方法来在这两个状态之间进行转换。它也提供了一个抽象的step方法用于随时间更新发动机的旋转状态。有多个类继承了该抽象类,它们采用了几个不同的转换算法。
6)Flagella类 该类也是抽象类,保存了两个状态:集中和发散。TetheredFlagellum类继承了Flagella类,它实现的转换算法是完全根据发动机的状态来改变自身状态。SwimmingFlagella也继承了Flagella类,它实现的状态转换算法有20%的几率由发散状态转为集中状态时可能失败。
7)Motion类 主要定义了细胞的运动,包括平稳运动和翻滚运动。
8)Molecules类 主要定义了分子类型,浓度等属性。
9)PPContextCreator类 负责根据 Repast S的接口规范创建Agent和网格对象。
3.3 仿真过程
Repast平台运行仿真程序的流程如图3所示。
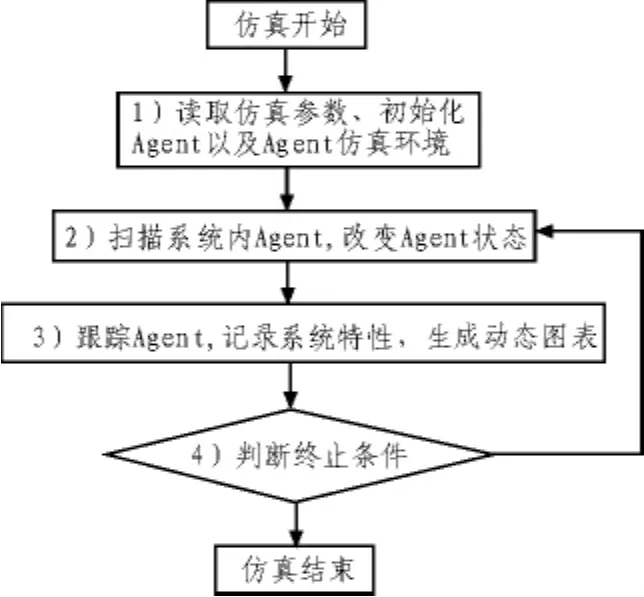
图3 Repast仿真过程的流程图Fig.3 Flow chart of simulation process
其中步骤1由PPContextCreator负责完成,具体过程如下:
1)读取仿真参数,包括细胞的个数,化学环境空间大小,细胞体积等;
2)根据步骤1)所获取的参数初始化连续的三维空间和Agent环境;
3)根据细胞个数创建细胞Agent实例,具体步骤如下:
①创建细胞Agent实例并初始化位置、运动方向和体积;②创建并初始化细胞的趋化网络实例,然后赋予细胞Agent实例;③创建并初始化细胞感知器实例,然后赋予细胞Agent实例;④创建并初始化细胞发动机实例,然后赋予细胞Agent实例;⑤创建并初始化鞭毛实例,然后赋予细胞Agent实例;⑥创建并初始化运动驱动器实例,然后赋予细胞Agent实例。
4)将所有的细胞Agent实例加入三维空间并移动到初始位置。
仿真初始化完成后,用户运行仿真,由Repast仿真引擎获取三维空间内的所有细胞Agent并依次调用细胞Agent的step()方法从而更新细胞Agent的状态。
在细胞Agent的step()方法中主要完成的工作包括:更新环境化学物质浓度、细胞移动、更新发动机以及鞭毛的状态,具体过程如图4所示。
在一个仿真时间片内,Repast仿真引擎会更新所有细胞Agent的状态而Repast平台的记录模块则收集用户事先定义的感兴趣的系统特性,并由绘图模块动态生成图表 (即图3中的步骤3),然后由仿真引擎判断仿真终止条件(即图3中的步骤4),若仿真不结束则推进仿真时间片并再次更新所有细胞Agent的状态(即重复图3中的步骤2),否则仿真结束。
3.4 仿真结果
为了验证本文方法的有效性,根据Morton-Firth和Korobkova[7]使用的参数设计了两个仿真试验:单个细胞趋化行为和群体细胞趋化行为,并将其趋化过程表现在3D环境中,如图5所示。这些试验仅考虑单个细胞或多个细胞在化学物质浓度梯度变化的环境中的趋化行为。
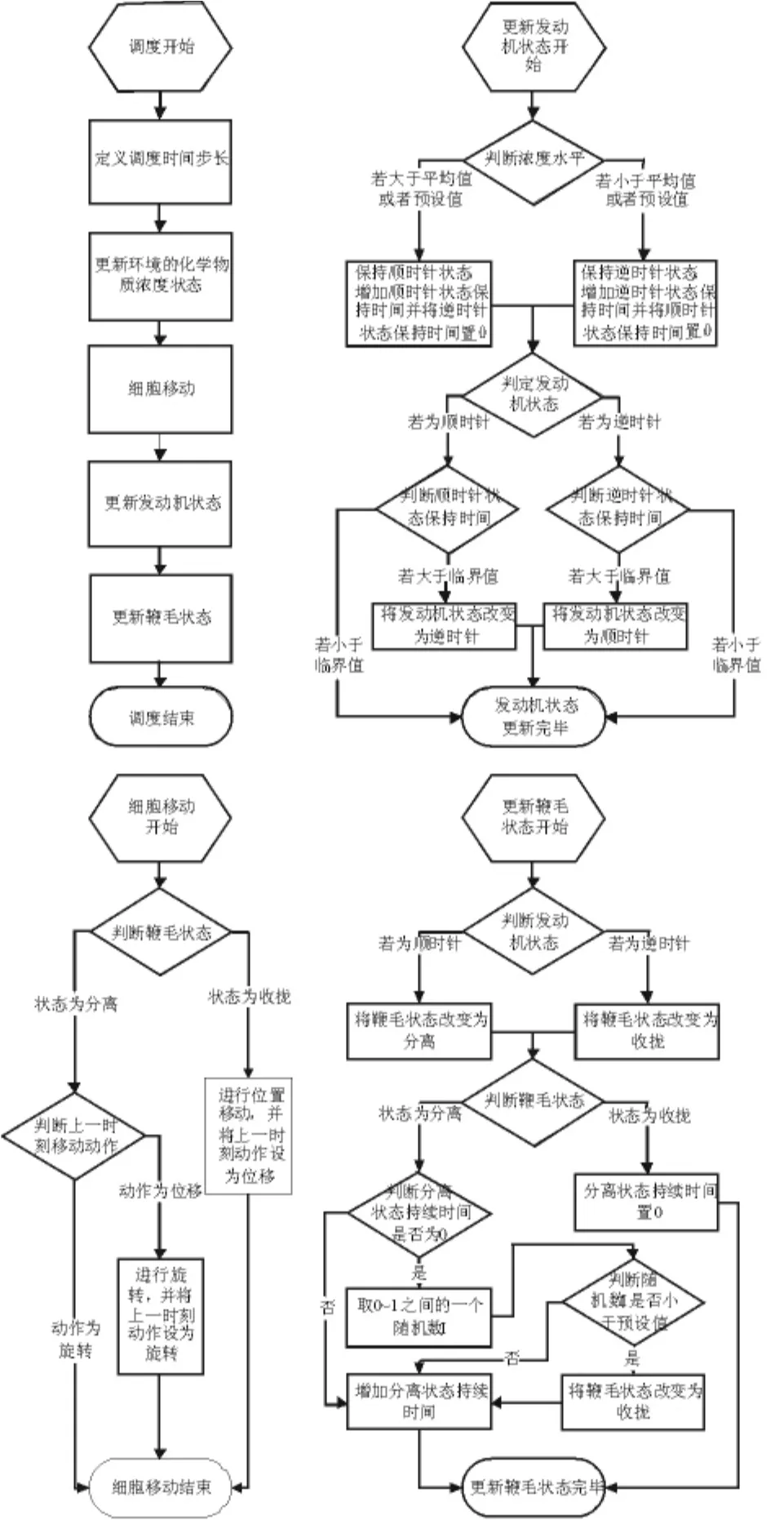
图4 细胞Agent的状态更新过程Fig.4 State updating process for cell Agent
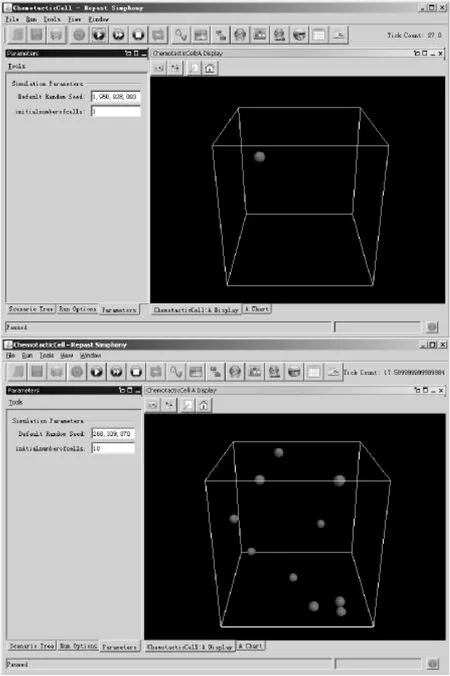
图5 三维显示效果Fig.5 Visual 3D effects of simulation
4 结 论
文中以Agent建模仿真方法为指导,以Repast建模仿真平台为手段,探索细胞在化学环境中的运动特性,为开展细菌趋化问题研究提供了良好的可重用仿真试验平台。随着研究的不断深入,还需要进一步对基于Agent的细菌趋化模型进行扩展完善,最终为细菌趋化性研究提供一个通用的仿真试验平台。
[1]Korobkova E,Emonet T,Vilar J M,et al.From molecular noise to behavioural variability in a single bacterium[J].Nature,2004(428):574-578.
[2]Solis F J,Corts J C,Cardenas O J.The stability of the keller-Segel model[J].Math Comp Model,2004(39):973-979.
[3]Berg H C.Motile behavior of bacterial[J].Physics Today,2000(53):24-29.
[4]姚灿中,杨建梅.基于Multi-Agent的复杂网络疾病传播仿真模型[J].计算机工程与应用,2010,46(20):12-15.
YAO Can-zhong,YANG Jian-mei. Complex network simulation model of disease spread based on multi-agent model[J].Computer Engineering and Applications,2010,46(20):12-15.
[5]瞿毅臻,李琦,甘杰夫.基于Repast平台的SARS传播仿真建模研究[J].计算机科学,2008,35(2):286-288.
QU Yi-zhen,LI Qi,GAN Jie-fu SARS transmission simulation and modeling based on repast platform[J].Computer Science,2008,35(2):286-288.
[6]Visual Agent—based Model Development with Repast Symphony[EB/OL].[2012-02]http://www.dis.anl.gov/publicatiom/articles/North_et_al_Repast_Simphony_Tutorial.pdf.
[7]Morton-Firth,Shimizu T,Bray D.A free-energy-based stochastic simulation of the rat receptor complex[J].J.Mole.Bio,1999(287):1059-1074.
