基于SSR标记的茶树新品种遗传多样性分析及指纹图谱构建
2012-06-08章志芳马建强
章志芳,马建强
(中国农业科学院茶叶研究所,国家茶树改良中心,浙江 杭州 310008)
DNA是生物的基本遗传物质,DNA水平上的遗传变异是物种差异最直接的体现。随着分子生物学技术的发展而形成的DNA分子标记技术,因其多态性好、易操作和标准化、不受环境及作物生育期等因素影响等优点,已成为作物品种鉴定最有效的检测手段。在一系列的分子标记体系中,SSR标记以其共显性、操作简单、通用性好、重现性高等优点,成为最广泛应用的分子标记。目前我国建立的DNA指纹数据库大多采用SSR标记体系[1]。
茶树为多年生木本植物,从幼苗期到成熟投产需要几年时间,而在茶树幼苗期通过表型性状进行品种鉴定十分困难,如若品种混杂,势必影响生产效益。因此,开发高效准确的品种鉴定方法对于茶树新品种保护、良种推广等具有重要意义。茶树SSR标记的开发和应用较晚,已发表的茶树SSR标记约300余个[2-4],且多应用于遗传多样性、遗传结构等研究[5-6],而在茶树DNA指纹图谱方面的报道较少[7]。笔者利用SSR标记对14个茶树新品种进行遗传多样性分析和分子鉴定,并尝试构建DNA指纹图谱,以期为今后开展茶树分子指纹图谱库构建、新品种鉴定和评价等研究提供参考。
1 材料与方法
1.1 材料
供试的14个茶树样品(表1)采自中国农业科学院茶叶研究所“国家种质杭州茶树圃”,选取完整、无病虫害感染的一芽二叶新梢,经液氮迅速冷冻后保存于-70℃冰箱中备用。

表1 供试的14个茶树品种
1.2 方法
1.2.1 基因组DNA提取 采用改进的CTAB法[8]提取基因组DNA,1%琼脂糖凝胶电泳检测DNA质量,ND-1000 UV-Vis Spectrophotometer检测 DNA浓度,稀释到10 ng/μL用于PCR扩增。
1.2.2 EST-SSR标记 参照乔婷婷[9]、周炎花[10]报道的EST-SSR标记,挑选其中10个多态性较高的标记由上海生物工程技术服务有限公司合成。
1.2.3 PCR扩增及产物检测 扩增反应在Bio-Rad PTC-200 PCR仪上进行,反应体系和扩增程序参照乔婷婷[19]的方法。PCR产物用10%的聚丙烯酰胺凝胶在CBS垂直电泳系统上检测,电泳后参照Charters和Wilkinson的方法进行银染显色[11]。
1.2.4 数据统计与分析 每个SSR标记只统计目的片段范围内的一个等位位点,其中每一条多态性条带视为一个等位变异,将电泳图上清晰的条带记为1,同一位置无带或不易分辨的弱带记为0。建立0-1原始数据库,然后将相同的带型组合记为一种基因型;并根据分子质量大小,将等位基因从大到小依次记作 1、2、3等,EST-SSR为共显性标记,对于二倍体茶树而言,一条带表示纯合,两条则为杂合,记录为 11、22、33、12、13、23 等基因型。
使用POPGENE[12]统计每个标记在14个茶树品种中扩增的等位基因数(Observed number ofalleles,Na),计算有效等位位点(Effective number of alleles,Ne)、等位基因频率(Allele frequency),观测杂合度(Observed heterozygosity,HO)、期望杂合度(Expected heterozygosity,HE)、Shannon 信息指数(Shannon’s information index,I)。采用 PowerMarker[13]计算基因型数(Genotype)、多态性信息量(polymorphism information content,PIC);SSR 位点的多态性信息量PIC=1-∑Pi2,式中Pi表示第i个等位位点出现的频率;并根据Nei’s遗传距离[14]构建茶树品种的无根Neighbor-Joining(NJ)聚类图。
2 结果与分析
2.1 14个茶树品种的SSR多态性
10个EST-SSR标记在供试的茶树品种中均表现出多态性(表2)。其中PIC>0.5的有3个,变异范围在 0.173(TM283)~0.618(TM209),平均为 0.387,表明这些标记在参试材料中多态性适中;10个标记共检测到29个等位基因,等位基因数变异范围为2~4个,平均2.9个;有效等位基因数变异范围为 1.237(TM283)~3.143(TM209),平均 1.993 个;基因型最多的为 7个(TM209),最少的为 2个(TM136,TM283),平均 3.9 个;遗传多样性指数最高的为 TM209(1.213),最低的为 TM283(0.341)。
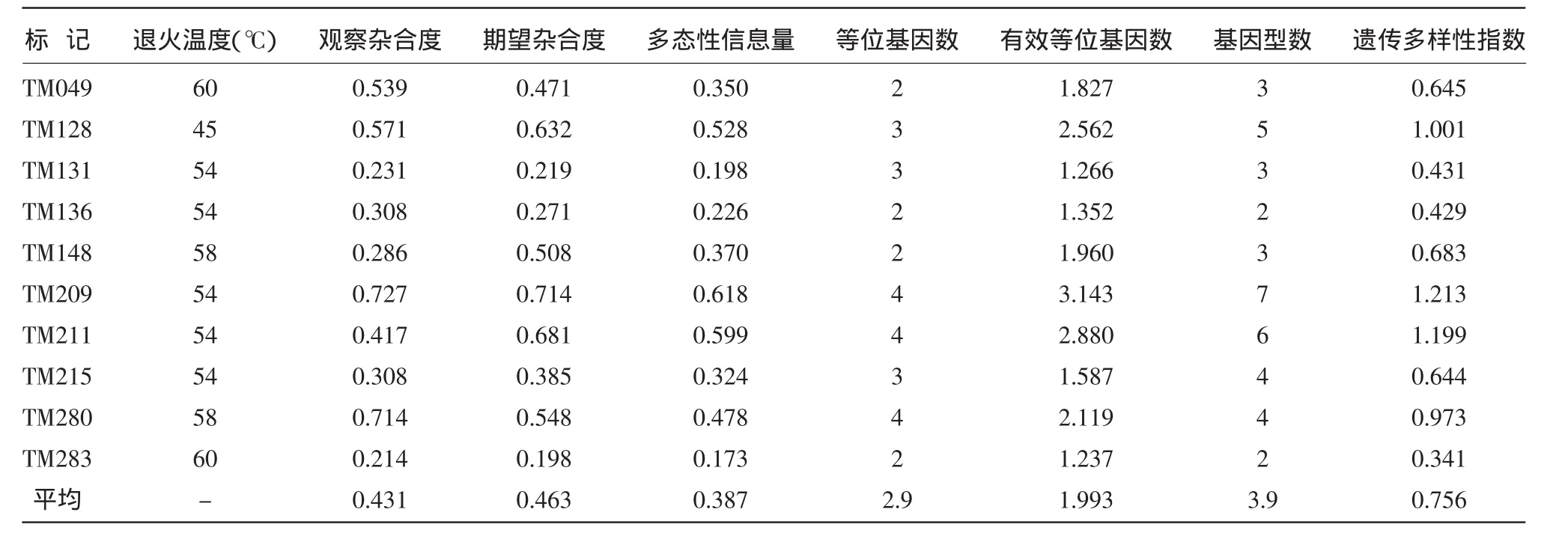
表2 10个茶树品种SSR标记的相关特征
2.2 茶树品种SSR标记等位基因的出现频率
29个等位基因在试验品种中的出现频率差异很大(表3)。TM283的等位基因2出现频率最高(89.29%);其次为TM136的等位基因2(84.62%)和TM131的等位基因1(88.46%)。TM280的等位基因1出现频率最低(3.57%);其次为TM131的等位基因2和TM215的等位基因3(均为3.85%)。对比表2和表3,可见出现频率较高的等位基因所对应的SSR标记其遗传多样性指数都较低。出现频率较低的等位基因所在的SSR标记分为两类:一类是标记的等位基因数较多的标记(TM209,TM211,TM280),另一类是等位基因数较少的标记(TM283)。
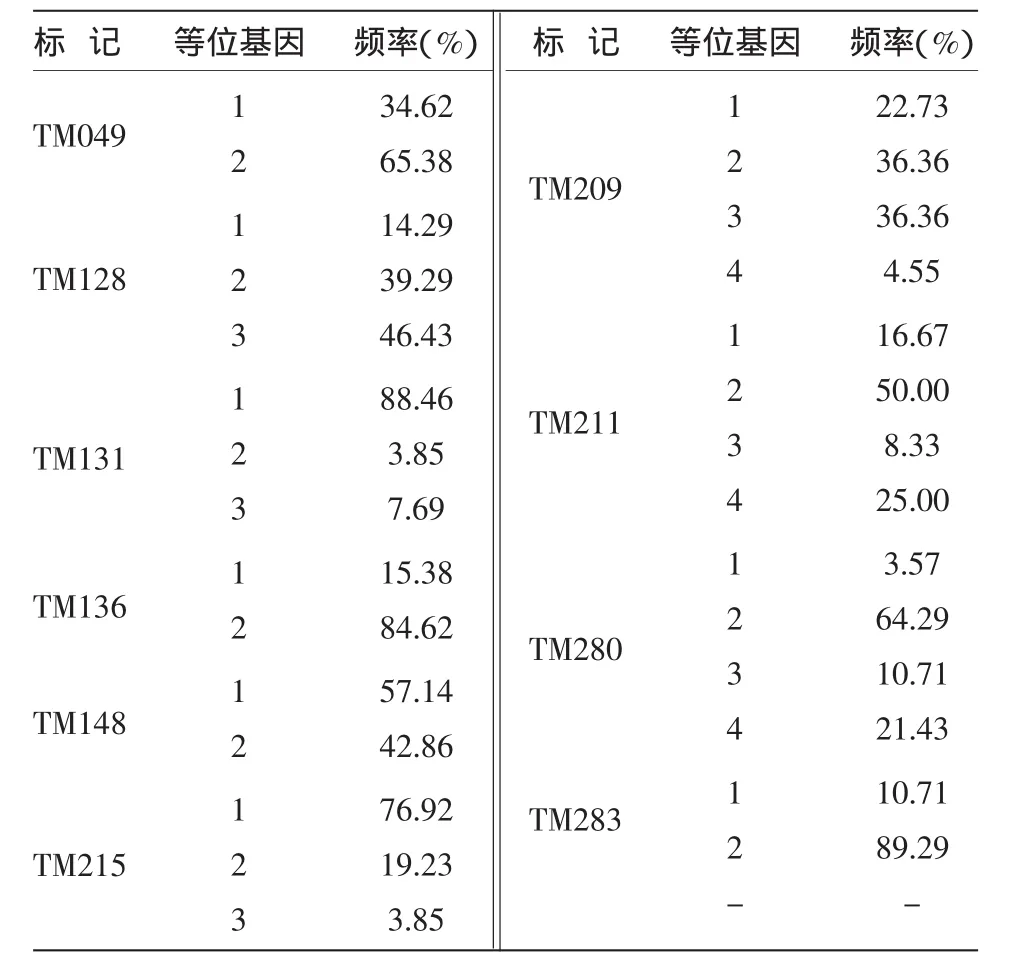
表3 SSR标记不同等位基因的出现频率
2.3 14个茶树品种的亲缘关系
根据10个SSR标记等位基因出现频率计算Nei’s遗传距离(D),结果显示14个茶树品种的遗传距离在0.036~0.472之间,其中南江1号和901的遗传距离最小,表明这两个品种间亲缘关系较近,而鄂茶5号和农抗早的遗传距离最大,说明这两品种亲缘关系最远。基于Nei’s遗传距离,采用NJ法构建品种聚类图(图1)。当D=0.12时,14个茶树品种可聚为三类;第一类包括4个品种,2个来自湖北(五峰212,五峰310),2个来自安徽(石佛翠,农抗早);第二类包括2个安徽品种(901,石佛香)和1个重庆品种(南江1号);第三类包括7个品种,其中2个湖北品种(鄂茶5号,鄂茶6号),3个福建品种(茗科3号,茗科4号,早玫瑰),1个四川品种(名山特早芽)和1个江西品种(大叶龙)。

表4 基于SSR标记的14个茶树品种的DNA身份证编码
2.4 14个茶树品种的DNA指纹图谱
将每个品种经不同标记扩增获得的等位基因带型,按照固定标记顺序排列,串联组合成特异的带型编码,再转化为计算机可识别的DNA身份证编码,即该品种的分子指纹图谱。为了减少编码序号长度,通常在构建指纹图谱时只选用几个鉴别能力较强的标记。该研究中只需4个SSR标记(TM128,TM280,TM148,TM211)即可鉴定 14 个茶树品种。因此,将这4个标记作为构建指纹图谱的核心标记。按照上述标记编号对每个茶树品种的等位基因带型组合进行编码,获得13位数的DNA身份证编码(表4),依此构建14个茶品种的指纹图谱(图1)。结果显示,每个茶树品种都对应一个惟一的指纹图谱,可较为快速地辨别各个品种。

图1 基于SSR标记的14个茶树品种的NJ聚类图及DNA指纹图谱
3 讨论与结论
3.1 茶树品种SSR标记等位基因变异
理论上参试品种越多,标记检测到的等位基因数就越多。乔婷婷[9]研究表明,其试验选用的10个SSR标记在308份茶树资源中检测到的等位基因数变异为 3(TM131,TM283)~8(TM211),平均为4.6,PIC只有 TM283小于 0.5,最高为 0.870(TM128),平均为0.677。而在该研究的14个茶树品种中检测到的等位基因数变异为2~4个,平均2.9个,其中9个标记(除TM131)检测到的等位基因数都少于之前的研究,只有3个标记(TM128,TM209,TM211)PIC 值 大 于 0.5, 最 高 为 0.618(TM209),最低为 0.173(TM283),平均为 0.387。这说明参试材料的不同,可能会造成标记检测效率差别较大。因此,在构建SSR指纹图谱库时应尽可能多地包含核心种植资源,以便获得更多的特异等位基因。
3.2 茶树品种SSR标记等位基因出现频率
该研究中等位基因出现频率变异范围为3.57%(TM280)~89.29%(TM283)。其中,出现频率较高的等位基因所在标记其遗传多样性指数都较低。而出现频率较低的等位基因所在的标记分为两类:一类是标记的等位基因数较多(TM209,TM211,TM280),但各等位基因出现频率较分散,使得其中某一个等位基因频率较低;另一类是等位基因数较少的标记(TM283),但其中某一个等位基因出现频率极高,而其他等位基因频率很低。对于这些在试验材料中出现频率很低的等位基因,可将其视为特异性等位基因(如TM131等位基因2,TM 209等位基因4,TM215等位基因3,TM280等位基因1),依此便可高效地鉴别具有该特异等位基因的材料,因此也适用于指纹图谱构建。
3.3 14个茶树品种的亲缘关系
研究结果表明,来自不同省份的14个茶树品种的Nei’s遗传距离在0.036~0.472之间,说明这些区试品种的亲缘关系较近。这可能是由于试验所用的14个品种均为选育的绿茶适制性品种,在遗传背景上可能较相近。基于遗传距离的NJ聚类图显示,当D=0.12时,14个茶树品种可聚为三类,来源相同的品种基本聚在一起。
3.4 茶树品种DNA指纹图谱的构建
标记的多态位点数对SSR分子标记的鉴别力有着重要的正向影响[15]。因此,在构建图谱时大多选择多态性较高的标记。该研究中利用4个SSR标记即可将14个茶树品种全部分开,并根据扩增的等位基因谱带组合类型将DNA多态性信息直接转化为计算机化的数字编码,然后绘制成可视化的条形码,结果显示每个品种都有唯一的指纹图谱,可以快速地对各品种进行鉴定。理论上,在利用SSR标记区分不同品种(二倍体)时,某一基因型的出现频率为2/(n+1)n(n为等位基因数),那么10个标记则可获得[(n+1)n/2]10个基因型组合类型。研究中10个SSR标记平均检测到2.9个等位基因,即理论上可区分3.3×107个品种。由此可见,利用SSR标记构建茶树指纹图谱进行品种鉴定具有极大的潜能和应用价值。随着茶树基因组测序的进行,可利用的SSR标记将大幅增加,构建覆盖整个茶树基因组的茶树指纹图谱库将成为可能,这对于茶树新品种的选育、鉴定、保护等具有重要意义。
[1] 李根英,Dreisigacker S,Warburton M L,等.小麦指纹图谱数据库的建立及SSR分子标记试剂盒的研发[J].作物学报,2006,32(12):1771-1778.
[2] Zhao L P,Liu Z,Chen L,et al.Generation and characterization of 24 novel EST derived microsatellites from tea plant(Camellia sinensis)and cross-species amplification in its closely related species and varieties[J].Conserv.Genet,2008,9:1327-1331.
[3] Ma JQ,Zhou Y H,Ma C L,et al.Identification and characterization of 74 novel polymorphic EST-SSR markers in the tea plant,Camellia sinensis(Theaceae)[J].Am.J.Bot,2010,97(12):153-156.
[4] Sharma H,Kumar R,Sharma V,et al.Identification and crossspecies transferability of 112 novel unigene-derived microsatellite markers in tea (Camellia sinensis)[J].Am.J.Bot,2011,98(6):e133-138.
[5] Yao M Z,Ma C L,Qiao T T,et al.Diversity distribution and population structure of tea germplasms in China revealed by ESTSSRmarkers[J].Tree Genet.Genomes,2012,8(1):205-220.
[6] FangW P,Cheng H,Duan Y S,et al.Genetic diversity and relationship of clonal tea(Camellia sinensis)cultivars in China as revealed by SSRmarkers[J].Plant Syst.Evol,2012,298(2):469-483.
[7] 杨 阳,刘 振,赵 洋,等.湖南省主要茶树品种分子指纹图谱的构建[J].茶叶科学,2010,30(5):367-373.
[8] Dellaporta SL,Wood J,Hicks JB.A plant DNA minipreparation:version II[J].Plant Mol.Biol.Rep,1983,1:19-21.
[9] 乔婷婷.茶树资源遗传多样性及其表型性状关联EST-SSR位点的初步鉴定[D].北京:中国农业科学院,2010.
[10] 周炎花.基于叶片形态学和EST-SSR茶树遗传多样性和遗传演化研究[D].福州:福建农林大学,2010.
[11] Charters Y M,Wilkinson M J.The use of self-pollinated progenies as‘in-groups’for the genetic characterization of cocoa germplasm[J].Theor.Appl.Genet.,2000,100:160-166.
[12] Yeh F C,Yang R C,Boyle T B J,et al.POPGENE,the userfriendly shareware for population genetic analysis[M].Edmonton:Molecular Biology and Biotechnology Centre University of Alberta,1997.
[13] Liu K,Muse S V.PowerMarker:an integrated analysis environment for genetic marker analysis [J].Bioinformatics,2005,21:2128-2129.
[14] Takezaki N,NeiM.Genetic distances and reconstruction of phylogenetic trees from microsatellite DNA [J].Genetics,1996,144:389-399.
[15] 韩凤龙,李正玲,胡 琳,等.用于河南省小麦品种特异性和一致性鉴定的SSR分子标记研究[J].中国农业科学,2010,43(18):3698-3704.
