山核桃SSR标记的初步开发
2012-05-30黄银芝董雷鸣黄有军曾燕如吴智敏
宋 双,黄银芝,董雷鸣,黄有军,曾燕如,吴智敏
(1.浙江农林大学 亚热带森林培育国家重点实验室培育基地,浙江 临安 311300;2.浙江省龙泉市林业局,浙江 龙泉 323700)
简单重复序列(simple sequence repeat,SSR)是一类一般由1~6个碱基组成的基元重复串联而成的脱氧核糖核酸(DNA)序列[1],基元重复次数一般为10~50,同一类微卫星DNA可分布在基因组的不同位置上;基因组中SSR多态性较丰富,呈共显性遗传,所以可提供的信息量较高,结果稳定可靠[2]。SSR标记分为两大类,即基因组SSR(genomic SSR)和表达序列标签SSR(expression sequence tag-SSR,ESTSSR)。基因组SSR是基于基因组序列开发的,采用构建基因组文库的方法,开发耗时费力,而且因为探针的缘故,种类有一定的局限性;EST-SSR是指存在于基因表达序列内的SSR,不包括存在于内含子及非表达调控区之中的SSR[3-4],它基于cDNA文库的构建及cDNA测序。Scott等[5]从5000条葡萄属Vitis EST中开发了124个SSR。EST-SSR标记不仅具有基因组SSR标记多态性高、共显性、重复性好的特点,而且开发成本相对低廉,在种属间具有良好的通用性[6]。另外,由于部分EST-SSR标记来自于基因的编码区,所以更容易获得基因表达的信息,可为功能基因的直接鉴定提供重要依据[7]。目前,该分子标记已被广泛应用于品种指纹鉴定[8-9]、遗传连锁图谱构建[10-11]、遗传多样性分析[12-13]和分子标记辅助育种[2,14-15], 在诸如小麦 Triticum aestivum[16-17], 大豆 Glycine max[18-19], 葡萄 Vitis vinifera[5], 胡椒 Lindera sp.[20], 白菜 Brassica campestris[21], 水稻 Oryza sativa[22], 猕猴桃 Actinidia chinensis[23]等植物中已成功开发了EST-SSR标记,但尚无山核桃Carya cathayensis EST-SSR标记的报道。山核桃是浙江省重要的经济树种。近年来随着研究的深入,已陆续在山核桃中建立了扩增片段长度多态性(amplified fragment length polymorphism,AFLP)[24], 随机扩增多态 DNA (random amplified polymorphic DNA, RAPD)[25], 微卫星间隔片段多态性(inter-simple sequence repeat, ISSR)[26], 序列相关扩增多态性 SRAP(sequence-related amplified polymorphism,SRAP)[27]标记分析体系,并将它们应用于山核桃遗传研究。研究发现:将这些标记应用于天然群体山核桃样品的分析,多态性总是不丰富[26-29]。这一方面与山核桃本身的遗传特性有关,另一方面与分子标记本身的局限性有关。上述分子标记均为显性标记,不能区分显性纯合位点与显性杂合位点,尽管SRAP相对而言可显示部分共显性[30-31]。因此,有必要开发诸如SSR的山核桃共显性标记,以深化山核桃的遗传研究。
1 材料与方法
1.1 试验材料
1.1.1 EST-SSR引物设计的数据源 黄银芝等[32]以山核桃果实油脂形成、转化、积累关键时期的果实为材料,构建了一个滴度为5.0×109空斑形成单位(pfu)·mL-1的山核桃脂肪代谢相关cDNA文库,并对部分cDNA片段进行了克隆测序。以序列分析中发现的含有SSR碱基重复特点的序列作为EST-SSR引物设计的数据源。
1.1.2 供试山核桃 5月,山核桃幼叶充分展开后,在山核桃产区采集40个单株的叶片,各单株在同一地点彼此间至少相距100 m。采样地点主要分布在山核桃主产区浙江省临安市的岛石、马啸、颊口、昌化、龙岗、横路、顺溪、湍口等地,所有单株均为野生成年大树。
1.2 方法
1.2.1 EST-SSR引物的设计 对含有SSR碱基重复特点的cDNA序列,利用Primer Premier 5(http://www.premierbiosoft.com/crm/jsp/com/pbi/crm/clientside/ProductList.jsp)进行引物设计。参照软件说明书设置引物设计参数:产物长度为100~300 bp;引物长度为18~25 bp;退火温度为45~65℃,且上下游引物的退火温度相差不大于5℃;引物碱基(GC)为40%~60%;尽量避免引物二级结构的出现。
1.2.2 DNA的提取 借鉴郭金剑[29]建立的适用于山核桃叶片DNA的提取方法,即改良十六烷基三甲基溴化铵(CTAB)法提取基因组DNA。DNA经吸光度值D(λ)测定及电泳检测,质量达到要求的稀释到100 mg·L-1用于 SSR 分析。
1.2.3 SSR反应体系的优化 本研究在郭金剑[29]建立的适用于山核桃的SSR反应体系的基础上,根据实验中出现的现象,对DNA模板用量及聚合酶链式反应(PCR)循环数进行优化,得到山核桃SSR最佳反应体系。
1.2.4 扩增产物检测及SSR引物筛选 EST-SSR引物由上海生工生物工程技术有限公司合成。先取3个样品的DNA对引物进行粗筛,即用优化的SSR反应与扩增体系扩增DNA后,通过10.0 g·L-1的琼脂糖凝胶电泳分离,检测引物是否有扩增产物。对有扩增产物的引物对,再用来自天然群体的40个山核桃单株样品进行扩增,扩增产物采用100.0 g·L-1非变性聚丙烯酰胺凝胶,在1×三羟基氨基甲烷-硼酸(TBE)电泳缓冲液中150 V恒压电泳1.5~2.0 h;电泳结束后,凝胶用蒸馏水清洗1 min;随后2.0 g·L-1的硝酸银和体积分数为10%乙醇溶液固定银染10 min;再用蒸馏水漂洗2次,2 min·次-1,后在20.0 g·L-1氢氧化钠和体积分数为0.4%甲醛混合溶液中显色,最后清水漂洗凝胶,并在BIO-RAD GS800成像系统中拍照保存。
1.2.5 数据分析 由于SSR为共显性标记,可以区分显性纯合子和显性杂合子,因此电泳结果按照共显性标记的读带方法:若条带间距离在20 bp以内,则视为1个杂合位点,2条带分别读为A,B带;若20 bp以内只有1个条带,则视为纯合位点,读为AA或BB。
1.2.6 山核桃SSR标记与其它标记分析结果的比较 本课题组已建立了山核桃AFLP,RAPD,ISSR,SRAP标记分析体系。本研究将SSR开发的初步实验结果与其余4种标记体系分析的结果进行了比较。
2 结果与分析
2.1 EST-SSR引物的设计
从1010个测序的cDNA克隆中得到了188条非冗余序列,发现43个cDNA序列具有SSR碱基重复特点。利用Primer Premier 5进行引物设计,其中基于9个cDNA序列设计的引物达不到设置的引物参数要求。因此,最后共设计SSR引物34对(表1)。

表1 开发的山核桃SSR引物序列及在供试样品中检测到的总位点数Table1 Sequences of the SSR primers developed and the total loci detected in Carya cathayensis

表1 (续)
2.2 DNA的提取
从供试样品中提取的基因组DNA D(λ)260/D(λ)280都为1.8~2.0,且DNA电泳条带明亮、清晰,纯度较高,可用于后续SSR分子标记的分析。
2.3 SSR体系的优化
经过试验过程不断优化,最终PCR产物电泳的扩增条带清晰,无拖尾、条带很粗,确定10.0 μL的PCR反应体系为:10×缓冲液 (buffer)1.0 μL,氯化镁 (MgCl2)1.0 μL,三磷酸碱基脱氧核苷酸 (dNTP)0.3 μL, SSR 正反向引物(浓度为 10μmol·L-1)各 0.5 μL, Taq DNA 聚合酶 0.1 μL(上海申能博彩生物科技有限公司), 模板 DNA(100 mg·L-1) 0.5 μL, 加无菌双蒸水(ddH2O)至终体积 10.0 μL; PCR 扩增条件为:94℃预变性5 min;随后30个循环,每个循环94℃变性30 s,退火30 s,72℃延伸60 s;最后72℃延伸3 min,其中退火温度各引物对有所不同(表1)。与郭金剑[29]建立的山核桃SSR体系相比较,本实验反应体系中的缓冲液用量减少了0.5 μL,Taq DNA聚合酶用量减少了0.1 μL,DNA用量减少了200 ng;郭金剑在其实验中使用了30.0 g·L-1的琼脂糖凝胶,而在本实验中采用的是分辨率更佳的非变性聚丙酰胺凝胶电泳(PAGE)。因此,反应体系中一些组分的用量有所减少,而电泳结果更加清晰。
基于各SSR引物对在3个DNA样品中的扩增结果,在琼脂糖凝胶中对引物对进行了初步的筛选。34对引物中有4对引物无扩增产物(图1)。对有扩增产物的引物对,用40个样品进行PCR扩增,扩增产物采用非变性PAGE电泳,结果30对引物有扩增产物(图2),但其中3对引物的扩增产物在500 bp以上。按照引物设计的参数,预期的扩增产物为100~300 bp。因此,这3对引物的扩增属非特异性扩增。最终确认可用的SSR引物为27对(表1)。

图1 山核桃SSR引物初步筛选电泳图(部分)Figure1 Preliminary screening of SSR primers in Carya cathayensis (partial)

图2 山核桃SSR标记分析电泳图(部分;引物对:SSR34)Figure2 Electrophorogram of SSR markers in Carya cathayensis (partial; primer pair: SSR34)
此外,在可用的27对引物的扩增产物中,按照重复基元的种类划分,可将引物分为3种重复基元的引物,其中二核苷酸重复基元出现的频率最高,为66.7%;其次分别为三核苷酸和四核苷酸重复,它们分别占30%和3.3%。二碱基重复基元中出现频率较多的重复基元是(A/T)n和(T/C)n,它们各占二核苷酸重复基元引物的35%和45%(表2)。
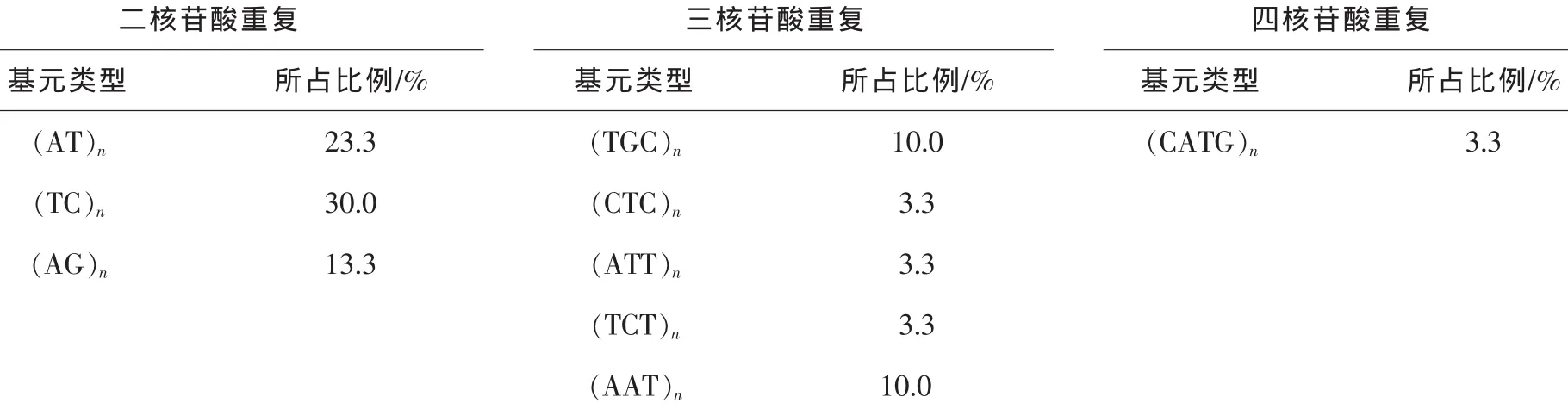
表2 山核桃EST-SSR引物扩增产物的重复基元特性Table2 Repeat contigs of SSR primer-amplified products in Carya cathayensis
2.4 SSR标记与其他标记分析结果的比较
27对SSR引物对40个山核桃样品进行分析,共扩增得到46个位点,平均每对引物产生1.7个位点,其中杂合位点28个,占总位点数的60.9%;纯合位点18个,占位点数的39.1%。27对引物中有18对在供试样品中扩增出22个多态性位点,占总位点数的47.8%。
与本课题组建立或优化的山核桃其他分子标记[28-29,33]相比较,SSR标记多态性位点的比例较高,且能够区分显性纯合位点及显性杂合位点外,但其余各指标均不如显性标记(表3)。SSR多态性产生的机制是因为DNA复制和修复过程中碱基的滑动、错配或减数分裂过程中姊妹染色单体的不均等交换[34];山核桃EST-SSR标记检测的位点数为1~3个,远不如其他显性标记。当然,多态性分析反映的是物种的遗传多样性,与该物种的本质特性有关。因此,利用SSR分析研究遗传多样性时,为获得一定数量的多态位点用于遗传分析,通常所需的SSR引物较多。对山核桃而言,还需继续开发SSR引物。
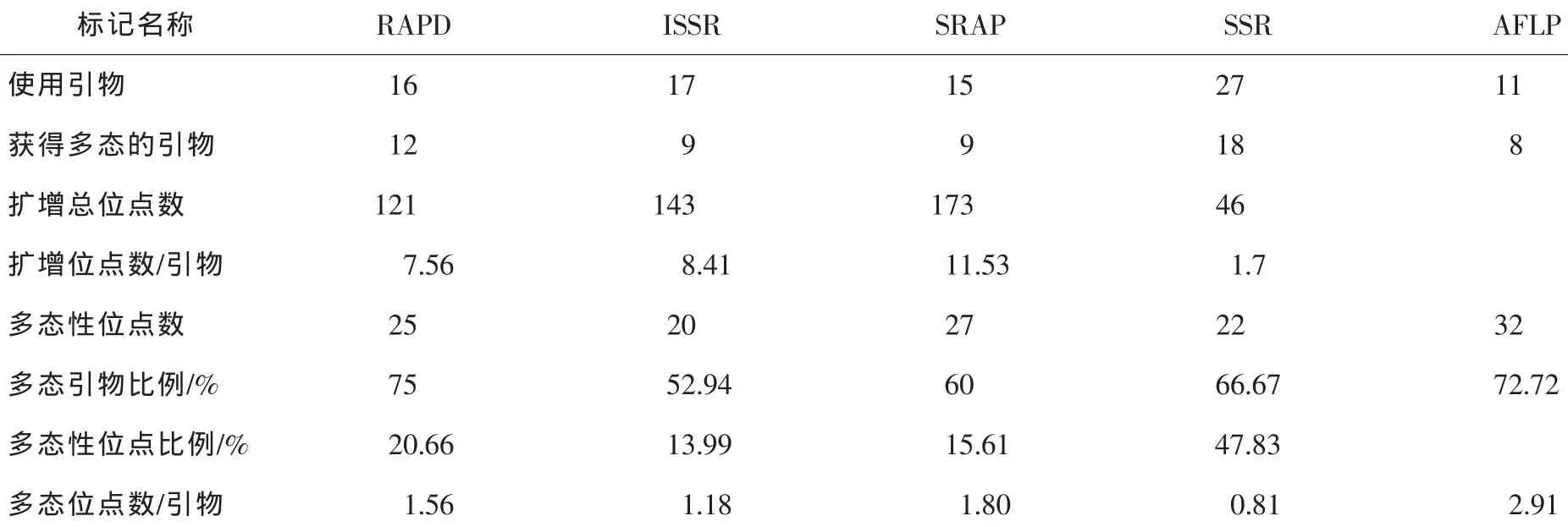
表3 山核桃各种分子标记扩增结果的比较Table3 Comparison among the amplification results of various molecular markers in Carya cathayensis
3 讨论
Silva等[35]通过研究甘蔗Saccharum sinensis EST,设计了20对EST-SSR引物,并利用这些引物研究了甘蔗栽培种和甘蔗属Saccharum内种间的多态性,结果表明EST-SSR具有较高的种间保守性。鉴于SSR标记的共显性及种属特异性,其开发的成本往往较高,因涉及文库的构建及序列的测定。本研究基于cDNA文库构建及部分测序结果,开发了27对山核桃EST-SSR引物,为后续SSR引物的进一步开发打下了实验基础。山核桃SSR标记的进一步开发,可以在现有文库cDNA测序的基础上继续进行。此外,黄有军[36]以山核桃雌花成花过程的花芽RNA为材料,结合454测序,共获得了1575个具有SSR碱基重复特点的序列。这些材料都可以作为山核桃EST-SSR标记进一步开发的数据源。
cDNA为基因表达过程产生的mRNA反转录而来,不含内含子序列。不同物种同一基因cDNA序列进行比对时或多或少具有一定的相似性。因此,理论上基于EST序列开发的SSR引物对的通用性要优于基于基因组文库开发的SSR引物对,尽管仍存在种属特异性的特点。本研究小组正在进行山核桃与美国山核桃Carya illinoinensis正反交研究,拟将从山核桃中开发的EST-SSR标记用于同属两物种正反交子代的分析,进一步验证所开发标记的通用性。
本研究对山核桃EST-SSR标记进行了开发,初步获得27对可用的SSR引物。研究的实验结果将为深入研究山核桃的遗传多样性、种质资源评价等奠定基础。
[1]HAYDEN M J, SHARP P J.Sequence-tagged microsatellite profiling (STMP): a rapid technique for developing SSR markers [J].Nucl Acids Res, 2001, 29: e43.
[2]HE C, POYSA V, YU K.Development and characterization of simple sequence repeat(SSR) markers and their use in determining relationships among Lycopersicon esculentum cultivars [J].Theor Appl Genet, 2003, 106 (2): 363-373.
[3]SHARMA V, BHARDWAJ P, KUMAR R, et al.Identification and cross-species amplification of EST derived markers in different bamboo species [J].Conserv Genet, 2009, 10: 721-724.
[4]刘贯水.毛竹SSR位点引物开发及部分竹种系统学分析[D].北京:中国林业科学研究院,2010.LIU Guanshui.Development of SSR Loci Primers in Phyllostachys heterocycla var.pubescens (Mazel) Ohwi and Phylogenetic Analysis in Bamboo Species [D].Beijing: Chinese Academy of Forestry, 2010.
[5]SCOTTKD, EGGLERP, SEATONG, etal.AnalysisofSSRsderivedfromgrapeESTs[J].TheorApplGenet, 2000, 100(5): 723-726.
[6]CORDEIRO G M, CASU R, MCLNTYRE C L, et al.Microsatellite markers from sugarcane (Saccharum spp.)ESTs cross transferable to erianthus and sorghum [J].Plant Sci, 2001, 160: 1115-1123.
[7]张智俊,管雨,杨丽,等.毛竹EST资源SSR标记分析与筛选[J].园艺学报,2011,38(5):989-996.ZHANG Zhijun, GUAN Yu, YANG Li, et al.Analysis of SSRs information and development of SSR markers from moso bamboo (Phyllostachys edulis) ESTs [J].Acta Hort Sin, 2011, 38 (5): 989-996.
[8]MARTIN H, JEROEN R V, ALBRECHT E, et al.Variation of DNA fingerprints among accessions within maize inbred lines and implications for identification of essentially derived varieties(Ⅱ)genetic and technical sources of variation in AFLP data and comparison with SSR data [J].Mol Breed, 2003, 12: 97-106.
[9]MAHMOOD A, BAENZIGER P S, BUDAK H, et al.The use of microsatellite markers for the detection of genetic similarity among winter bread wheat lines for chromosome 3A [J].Theor Appl Genet, 2004, 109 (7): 1494-1503.
[10]PIQUEMAL J, CINQUIN E, COUTON F, et al.Construction of an oilseed rape (Brassica napus L.) genetic map with SSR markers [J].Theor Appl Genet, 2005, 111 (8): 1514-1523.
[11]TAE-YOUNG H, TAKASHI S, MASAKAZU T.High-density integrated linkage map based on SSR markers in soybean [J].DNA Res, 2009, 16: 213-225.
[12]ENOKI H, SATO H, KOINUMA K.SSR analysis of genetic diversity among maize inbred lines adapted to cold regions of Japan [J].Theor Appl Genet, 2002, 104 (8): 1270-1277.
[13]ILPREET D, RIAR S, ACHIN S, et al.EST-SSR development from 5 Lactuca species and their use in studying genetic diversity among L.serriola biotypes [J].J Hered, 2011, 102 (1): 17-28.
[14]KIMURA T, SHI Yongzhong, SHODA M.Identification of Asian pear varieties by SSR analysis [J].Breed Sci,2002, 52: 115-121.
[15]EUJAYL I, SLEDGE M K, WANG L, et al.Medicago truncatula EST-SSRs reveal cross-species genetic markers for Medicago spp.[J].Theor Appl Genet, 2004, 108 (3): 414-422.
[16]陈海梅,李林志,卫宪云,等.小麦EST-SSR标记的开发、染色体定位和遗传作图[J].科学通报,2005,50(20):2208-2216.CHEN Haimei, LI Linzhi, WEI Xianyun, et al.Development, chromosome mapping and mapping of EST-SSR markers in wheat[J].Chin Sci Bull, 2005, 50 (20): 2208-2216.
[17]庄丽芳,宋立晓,冯祎,等.小麦 EST-SSR标记的开发和染色体定位及其在追踪黑麦染色体中的应用[J].作物学报, 2008, 34(6): 926-933.ZHUANG Lifang, SONG Lixiao, FENG Wei, et al.Development and chromosome mapping of 81 new wheat ESTSSR markers and application for characterizing rye chromosomes added in wheat [J].Acta Agro Sin, 2008, 34 (6):926-933.
[18]常玮,赵雪,李侠,等.大豆EST-SSR标记开发及与Genomic SSR的比较研究[J].中国油料作物学报,2009,31(2): 149-156.CHANG Wei, ZHAO Xue, LI Xia, et al.Development of soybean EST-SSR marker and comparison with genomic SSR marker [J].Chin J Oil Crop Sci, 2009, 31 (2): 149-156.
[19]洪彦彬,陈小平,刘海燕,等.源于大豆 EST的花生属Arachis同源 SSR标记的开发及利用 [J].作物学报,2010, 36 (3): 410-421.HONG Yanbin, CHEN Xiaoping, LIU Haiyan, et al.Development and utilizaiton of orthologous SSR markers in arachis through soybean (Glycine max) EST [J].Acta Agr Sin, 2010, 36 (3): 410-421.
[20]YI G, LEE J M, LEE S, et al.Exploitation of pepper EST-SSRs and an SSR-based linkage map [J].Theor Appl Genet,2006, 114 (1): 113-130.
[21]忻雅,崔海瑞,卢美贞,等.白菜EST-SSR信息分析与标记的建立[J].园艺学报,2006,33(3):549-554.XIN Ya, CUI Hairui, LU Meizhen, et al.Data mining for SSRs in ESTs and EST-SSR marker development in Chinese cabbage [J].Acta Hort Sin, 2006, 33 (3): 549-554.
[22]朱宏波,方宣钧,杨仁崔.利用水稻基因组序列数据开发SSR标记的方法[J].分子植物育种,2003,1(2):273-276.ZHU Hongbo, FANG Xuanjun, YANG Rencui.Developing SSR markers using public rice genome sequence data[J].Mol Plant Breed, 2003, 1 (2): 273-276.
[23]FRASER L G, HARVEY C F, CROWHURST R N, et al.EST-derived microsatellites from Actinidia species and their potential for mapping [J].Theor Appl Genet, 2004, 108 (6): 1010-1016.
[24]郭金剑,池伟,赵鑫珠,等.山核桃AFLP实验技术体系的建立[J].浙江林学院学报,2008,25(4):532-537.GUO Jinjian, CHI Wei, ZHAO Xinzhu, et al.An AFLP analysis system for Carya cathayensis [J].J Zhejiang For Coll, 2008, 25 (4): 532-537.
[25]王正加,黄坚钦,郭传友,等.山核桃RAPD反应体系的优化[J].浙江林学院学报,2003,20(4):429-433.WANG Zhengjia, HUANG Jianqin, GUO Chuanyou, et al.The optimal reaction system of RAPD in Carya cathayensis [J].J Zhejiang For Coll, 2003, 20 (4): 429-433.
[26]沈林.无融合生殖连锁分析模型:美国山核桃×山核桃杂种的实例研究[D].临安:浙江农林大学,2010.SHEN Lin.A Model for Linkage Analysis with Apomixis: A Case Study for Pecan × Hickory Hybrids [D].Lin’an:Zhejiang A&F University,2010.
[27]李元春.基于分子标记的山核桃生殖特性研究[D].临安:浙江农林大学,2011.LI Yuanchun.Molecular Marker Analysis of Parents and Their Semi-sib Progenies for the Reproduction in Hickory[D].Lin’an: Zhejiang A&F University, 2011.
[28]沈林,郭金剑,李阳,等.基于AFLP分析的山核桃群体结构及遗传多样性[J].福建林业科技,2009,36(4):84-86.SHEN Lin, GUO Jinjian, LI Yang, et al.Population structure and genetic diversity of Carya cathayensis revealed by AFLP analysis [J].J Fujian For Sci Technol 2009, 36 (4): 84-86.
[29]郭金剑.山核桃群体结构及多样性的研究[D].临安:浙江农林大学,2008.GUO Jinjian.Study of Population and Diversity in Carya cathayensis Sarg.[D].Lin’an: Zhejiang A&F University, 2008.
[30]张彤,屈淑平,崔崇士.工业SRAP标记在蔬菜作物遗传育种中的应用[J].东北农业大学学报,2009,40(1):119-122.ZHANG Tong, QU Shuping, CUI Chongshi.Application of SRAP marker on genetics and breeding of vegetable crops[J].J Northeast Agric Univ, 2009, 40 (1): 119-122.
[31]郭修武,张鹏翔,郭印山,等.应用 SRAP分子标记技术鉴定葡萄种间杂交后代[J/OL].分子植物育种:网络版,2011,9:1389-1393.GUO Xiuwu, ZHANG Pengxiang, GUO Yinshan, et al.Authenticity identification of progenies from interspecific cross red Globe (Vitis vinifera) × Shuangyou (Vitis amurensis) by SRAP markers [J].Fenzi Zhiwu Yuzhong, 2011, 9:1389-1393.
[32]黄银芝,周秦,黄有军,等.山核桃脂肪代谢相关cDNA文库的构建[J].浙江农林大学学报,2011,28(1):80-85.HUANG Yinzhi, ZHOU Qin, HUANG Youjun, et al.Construction of a fat metabolism-related cDNA library in Carya cathayensis [J].J Zhejiang A&F Univ, 2011, 28 (1): 80-85.
[33]李元春,沈林,曾燕如.山核桃SRAP体系的建立及与RAPD和ISSR标记的比较[J].浙江农林大学学报,2011,28(3): 505-512.LI Yuanchun, SHEN Lin, ZENG Yanru.Establishment of a SRAP analysis protocol in Carya cathayensis and a comparison among SRAP, RAPD, ISSR analysis protocols [J].J Zhejiang A&F Univ, 2011, 28 (3): 505-512.
[34]POWELL W, MORGANTE M, ANDRE C, et al.The comparison of RFLP, RAPD, AFLP and SSR (microsatellite) markers for germplasm analysis [J].Molr Breed, 1996, 2: 225-238.
[35]SILVA A G.Preliminary analysis of microsatellite markers derived from sugarcane expressed sequence tags (ESTs)[J].DNA Seq, 2004, 15 (1): 71-76.
[36]黄有军.山核桃成花机理研究[D].南京:南京林业大学,2010.HUANG Youjun.Study on Mechanism of Floral Formation in Carya cathayensis [D].Nanjing: Nanjing Forestry University,2010.
