梓树属植物基因组DNA提取及RAPD体系优化
2012-05-29王巧玲
王 念,王巧玲
梓树属植物基因组DNA提取及RAPD体系优化
王 念1,王巧玲2
(1.河南省林木良种选育与种质资源保护重点实验室,郑州 450008; 2.河南省林业调查规划院,郑州 450045)
以楸树幼嫩叶片为材料,进行楸树基因组DNA提取及RAPD扩增条件优化,试验表明,采用改良的2×CTAB法提取的DNA质量较高,适宜于RAPD分析。RAPD反应的优化体系为(25 ul反应体系):17.8 ul ddH2O;0.5 ul dNTP;2.5 ul 10×buffer;1.0 ul 随机引物;2.0 ul Mgcl2;1.0 ul模版;0.2 ul Taq 酶 。
楸树;DNA提取;RAPD
梓树属是紫葳科的关键物种。在梓树属中楸树最为名贵,是我国传统的优质珍贵用材树种,素有“木王”之称[1]。随机扩增多态DNA(Random Amplified Polymorphic DNA,简称RAPD)是J.Wi11iams和J.Welsh[2]研究小组于1990年同时提出的一种随机引物扩增、寻找多态DNA片段的遗传标记技术。RAPD以PCR技术为基础,以基因组DNA为模板,以单个人工合成的随机多态核苷酸序列(通常为10个碱基对)为引物,在热稳定的DNA聚合酶作用下,进行PCR扩增[3]。扩增产物的多态性反映了基因组片段的多态性。RAPD技术在物种亲缘关系、系统发育分子水平的鉴别,以及分子生物学、分子生态学的研究中能够起到重要的作用。我们对梓树属植物基因组DNA提取及RAPD扩增条件优化进行了初步研究,为梓树属植物分子水平的深入研究奠定了良好的基础。
1 材料与方法
1.1 材料
试验材料金丝楸(J)、灰楸(H)、梓树(Z)、美国梓树(M)、豫楸1号(Y)来自河南省林业科学研究院试验场。随机引物由大连宝生物生物工程公司合成,Taq酶,dNTP,10×PCR Buffer,Mg2+均购自上海生物工程公司。PCR扩增采用美国伯乐公司MyCycler PCR扩增仪器,凝胶成像采用伯乐Gel Doc XRTM凝胶成像分析系统。
1.2 方法
1.2.1梓树属基因组DNA提取
采用2×CTAB法提取梓树属基因组DNA:①取幼嫩叶片加液氮研磨成粉末状,转入1.5 ml离心管中;②加入600 ul 65℃预热的2×CTAB溶液混匀并置于65℃水浴中保温30~60 min;③取出冷却后,上下摇匀,加入600 ul的氯仿异戊醇(24∶1),12 000 r/min离心10 min,④取上清液于另1个1.5 ml的离心管中;加600 ul异丙醇,12 000 r/min离心10 min,倒掉上清液,用75%的酒精清洗,沉淀晾干后加100 ul无菌水溶解;⑤加10 ul(10 mg/ml)的RNA酶,37℃水浴酶解1 h,补水至500 ul;⑥加500 ul的苯酚/氯仿/异戊醇(25∶24∶1),混合均匀,12 000 r离心10 min,抽提纯化2次;⑦取上清液,加入等体积的氯仿,混合均匀,12 000 r离心10 min;⑧取上清液,加入1/10体积3 mol/L的乙酸钠和2.5倍体积的预冷无水乙醇,-20℃放置30 min以上;⑨12 000 r离心10 min后,沉淀即为提取得到的基因组DNA,用70%酒精清洗3次,晾干后加100 ul TE或无菌水溶解。
1.2.2基因组DNA浓度及质量检测
将原液稀释50倍后,用紫外分光光度计测定DNA样品在260 nm和280 nm的吸光度,计算OD260 nm/OD 280 nm的值,按1 OD260=50 ng/μl计算基因组DNA原液的浓度,即50×OD260 ×稀释倍数。
琼脂糖凝胶电泳检测基因组DNA片段的大小,将DNA样品用无菌水稀释50倍,取5 ul用0.8琼脂糖进行凝胶电泳分析,检测所提取DNA片段量。
RAPD分子标记技术的缺点就是重复性差,试验过程中容易受到反应体系中其他因素的影响,比如退火温度、模板DNA质量、扩增参数的设置等。该研究对影响扩增的主要因素Mg2+的浓度、Taq聚合酶的用量、dNTP的用量进行了优化,建立适宜梓树属反应的优化体系。
采用25 ul反应体系,包括ddH2O、dNTP、10×Buffer、引物、模板、Taq酶,设置镁离子浓度梯度为50 umol/L、100 umol/L、150 umol/L、200 umol/L、250 umol/L;dNTP浓度梯度为50 umol/L 、100 umol/L 、150 umol/L 、200 umol/L、250 umol/L;Taq酶浓度梯度0.5 U、1.0 U、1.5 U、2.0 U、2.5 U,通过对比试验找出最佳反应体系,并利用42种不同引物进行优化条件的检验。
1.3 DNA扩增反应程序
根据树种的不同和引物合成的退火温度,程序设置如下:首先进行预变性95℃ 5 min,预变性之后进入循环体系:变性:94℃ 30 s;退火:36℃ 30 s;延伸:72℃ 45 s,共45个循环,最后72℃再延伸10 min,反应结束后4℃保存。
1.4 电泳检测
实验结束后,取15 ul扩增产物用0.5倍TBE电泳缓冲液,在1.5%的琼脂糖凝胶中进行电泳,电压为100 V。电泳结束,EB染色20 min后,在伯乐凝胶成像仪下观察、拍照。
2 结果分析
2.1 梓树属基因组DNA提取质量及电泳分析
通过分光光度计分析得知提取得到的DNA浓度较高,样品色泽白色透明,OD260nm/ OD280nm为1.73,接近1.8,表明DNA浓度符合RAPD分析要求。
通过0.8%琼脂糖凝胶电泳分离检测,溴化乙锭染色,观察DNA分子的完整性,结果见图1。基因组DNA在凝胶上表现为一条规则的带,基本无降解现象,有少量的RNA,但不影响RAPD分析。

图1 楸树基因组总DNA电泳图谱
2.2 RAPD扩增条件优化结果及参数确定
2.2.1镁离子和dNTP浓度优化及结果
该实验设计了镁离子为50 um/L、100 um/L、150 um/L、200 um/L、250 um/L的浓度梯度,从实验结果分析,镁离子浓度对扩增产物的影响很明显:浓度太低时,没有扩增产物出现(图2中第1、2泳道),浓度太高时,条带模糊且多态性低(图2中的5泳道)。在浓度为150 um/L或200 um/L时条带多且较为清晰。
在扩增反应体系中,dNTP是PCR反应的原料,在实验过程中会螯合部分镁离子,减少溶液中镁离子的数量,导致扩增谱带的改变直至产物消失。因此,镁离子和dNTP的浓度要综合考虑,结合实验结果(见图2),采用镁离子最终浓度是200 umol/L,dNTP的最佳浓度是50 umol/L。
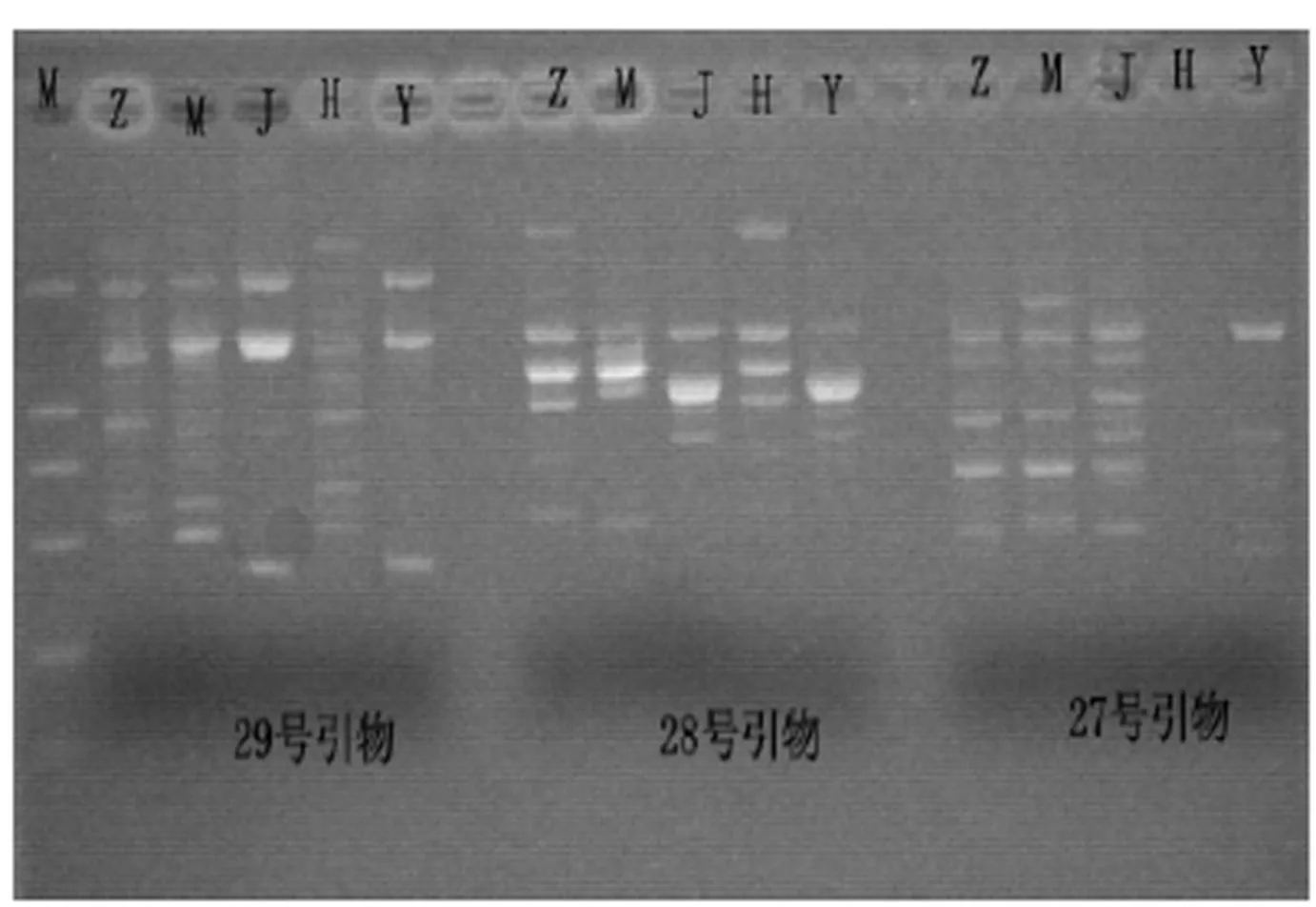
图 2 dNTP和镁离子浓度优化扩增效果
2.2.2 Taq酶浓度优化及结果
该实验设计Taq酶5个浓度梯度,分别是25 ul体系中0.5 U、1 U、1.5 U、2 U、2.5 U。由图3的电泳图可见,Taq酶以1 U为最好,条带多而且清晰,浓度减少条带清晰度下降,而浓度增加条带变粗,背景较模糊。
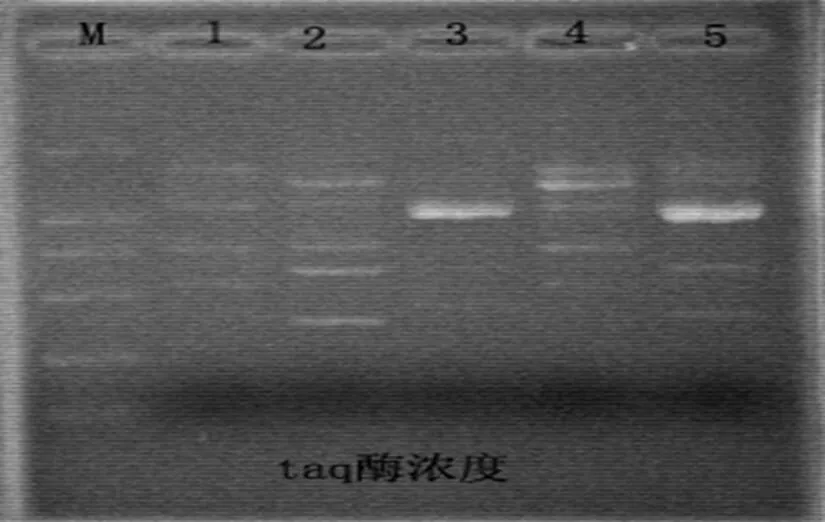
图3 Taq酶浓度优化扩增效果
2.3 RAPD扩增条件优化参数确定
经过对镁离子、dNTP、Taq酶的优化,建立如下RAPD优化体系:10×buffer:2.5 ul;Mgcl2:2.0 ul;dNTP:0.5 ul;Taq酶:0.2 ul;引物:1.0 ul;模板:1.0 ul,加双蒸水至25 ul。
PCR循环参数设定:95℃预变性5 min,94℃变性30 s、36℃退火30 s、72℃延伸45 s,进行45个循环,然后72℃延伸10 min,最后4℃保存。
2.4 梓树属基因组RAPD测定结果
用优化参数和选定的扩增条件对梓树属5种植物基因组DNA进行扩增,扩增产物用1.5%琼脂糖电泳分离,溴化乙锭染色,凝胶成像系统照相,结果见图4。由图4可见,DNA扩增图谱清晰,豫楸1号和金丝楸谱带相同,这和豫楸1号是从金丝楸中选育出来的实践结果一致。不同品种之间DNA存在明显的多态性,其中1 500 bp为5个品种所共有,在不同样本中没有变异,这表明它们是比较保守的DNA片断,其他条带在不同品种和个体中或有或无,这反映了梓树属品种的遗传多样性,同时也说明了我们建立的RAPD优化体系适合梓树属植物遗传多样性分析。
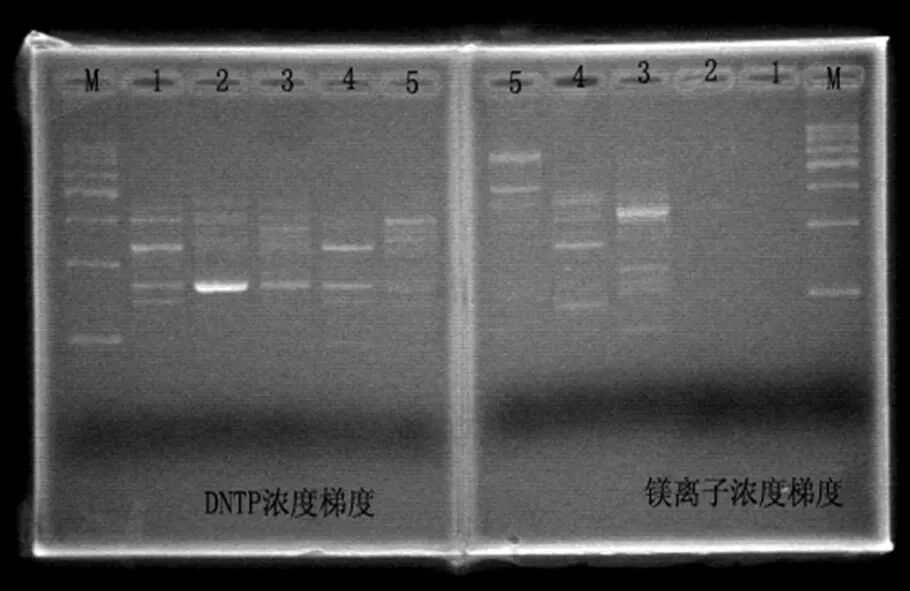
图4 不同品种在不同引物条件下的RAPD扩增效果
3 结论与讨论
通过对反应体系的优化,建立了梓树属植物的RAPD-PCR最佳反应体系,为下一步进行梓树属植物种质资源的RAPD分析及其亲缘关系鉴定、遗传多样性研究提供了可靠的试验依据。
在优化RAPD扩增条件过程中,应尽可能找出各种对RAPD 扩增产生不良影响的高低变数[4]。多次实验证明,采用梯度设置法是优化RAPD扩增条件的简便快速、经济实用方法,它使RAPD扩增条件优化过程实现了程序化和数量化,采用该方法、程序与策略,可以在短时间内获得RAPD扩增全部优化参数,节约大量的试剂、DNA样本和实验时间,应用该方法进行RAPD 扩增可以获得图谱清晰、稳定可靠的实验效果。
[1]郭从俭. 楸树栽培[M]. 北京:中国林业出版社,1988.
[2] Willamas J.G. Kubelik.A.R. Livak,K.J.etc. DNA Polymorphisms as amplified by arbitrary primers are useful as genetic marker[J]. Nucleic AciRes.1990, 1(18): 6531-6535.
[3]黄映萍. DNA分子标记研究进展[J]. 中山大学研究生学刊,2010,31(2):27-36.
[4]奥斯伯F,金斯顿RE,塞德曼JG,等. 精编分子生物学实验指南[M]. 北京:北京科学出版社,1999:37-39.
Genomic DNA Extraction and Optimization of RAPD System of Catalpa Scop
WANG Nian1, WANG Qiao-ling2
(1.Key Laboratory of Forest Germplasm Conservation、Breeding of Improved Variety of Henan-Province,Zhengzhou 450008, China; 2. Henan Forest Inventory and Planning Institute, Zhengzhou 450045, China)
The method of genomic DNA extraction from new leaves and the optimization of RAPD analytic conditions were studies in Catalpa bungei. The result showed that the high-quality genomic DNA was obtained by the revised 2×CTAB method. The optimal PCR system for RAPD analysis was as follows:17.6ul ddH2O;0.5ul dNTP;2.5ul 10×buffer; 1.0ul random Primer;2.0ul Mgcl2;1.0ul template;0.4ul Taq polymerase in 25ul reaction system.
Catalpa bungei; Genomic DNA extraction; RAPD
Q 343.1
A
1003-2630(2012)01-0001-03
2012-02-15
国家林业局“十二五”科技支撑项目(编号:2012BAD01B05)。
王念(1978-),女,河南省上蔡县人,工程师,主要从事林木遗传育种研究。
(责任编辑:王文彬)
