小鼠胰岛素样生长因子1(IGF1)基因启动子的生物信息学分析
2012-05-11王兴平罗仍卓么
李 峰, 张 宇,王兴平,罗仍卓么,李 荣
小鼠胰岛素样生长因子1(IGF1)基因启动子的生物信息学分析
李 峰, 张 宇,王兴平,罗仍卓么,李 荣
(湖南文理学院 生命科学学院, 湖南 常德, 415000)
生物信息学的发展为在线分析软件预测基因启动子的相关信息提供了众多有重要价值的参考信息. 采用进化足迹法, 结合生物信息学工具, 运用在线软件进行分析, 分析了小鼠IGF1基因的启动子结构. 分析结果表明, 在小鼠IGF1基因的启动子保守区内预测出6个转录因子结合部位, 为探讨该基因的表达调控机制提供了重要参考.
IGF1; 进化足迹法; 启动子区; 转录因子; 生物信息学分析
胰岛素样生长因子1(IGF1)由159个氨基酸残基组成, 是一种多功能细胞增殖调控因子, 其生物学功能主要是刺激有丝分裂, 诱导及促进细胞分化. 在临床上, IGF1在神经损伤、糖尿病、生长迟滞、骨质疏松等疾病的治疗中显示出了广阔的前景, 因此对IGF1基因的表达调控的研究有重要的意义.
基因表达调控机制是后基因组时代一个重要的研究内容. 基因正确的表达依赖于一个复杂的调控机制. 不同的转录因子对外界环境的各种刺激或不同发育阶段的各种信号做出反应, 结合于转录调控元件, 激活或抑制基因的转录, 从而控制不同基因的表达.
启动子(promoter)是RNA聚合酶识别、结合并开始转录所必需的一段DNA序列. 不同的启动子都存在保守序列, 包括RNA聚合酶识别位点和结合位点. 真核生物启动子是在基因转录起始位点(+1)及其5′上游近端大约100—200bp(或下游100bp)的一组具有独立功能的DNA序列, 包括核心启动子元件(TATA框)和上游启动子元件(CAAT框和GC框等). 启动子元件与相应的转录结合因子结合决定基因转录起始和频率[1-6].
进化足迹法指通过两种或两种以上基因组间同源基因序列的比较, 发现进化中保守的功能元件. 生物的基因组由于内外环境的影响产生突变, 每个突变的结果依赖于其对表型的影响. 致死的突变通常被自然选择消除, 而对表型无影响的突变(中性突变)或影响不大的突变可以在群体中随机出现. 这样造成的结果就是突变在无功能的DNA碱基对处积累的速度大大快于功能性位点[1-6]. 因此, 如果一段序列高度保守, 则很可能意味着这段序列是有功能的[3].
本文采用进化足迹法, 对该基因的启动子区进行分析. 首先获得该基因在人类中的同源基因及其启动子区. 再通过搜索TRANSFAC数据库和基因组之间的比较, 筛选出人与小鼠新基因启动子区保守区共同存在的转录因子结合部位(transcription factor binding sites), 降低了假阳性率, 使预测结果更为准确[7].
本文通过对IGF1基因启动子区的生物信息学分析, 查找出基因启动子区中存在的转录因子结合位点, 为探讨该基因表达调控机制及构建组织特异性启动子的研究奠定基础.
1 材料与方法
1.1 材料
1.1.1 IGF1基因序列和基因组结构
IGF1基因cDNA序列全长为7 121bp, 阅读框架位于第295-774位之间. IGF1基因位于小鼠10号染色体的C1区, 基因组跨越72 324 642 bp, 含5个外显子和4个内含子.
1.1.2程序和数据库
序列对比程序BLASTN: www.ncbi.nlm.nih.gov/BLAST/
启动子在线分析软件: Promoter2.0: www.cbs.dtu.dk/services/promoter
启动子在线分析软件: Neural Network Promoter Prediction: www.fruitfly.org/seq_tools/promoter.html
启动子在线分析软件: Promoter SCAN:http://bimas.dcrt.nih.gov/molbio/proscan
转录因子预测软件: Match1.0-public: www.gene-regulation.com/pub/programs.html/match
CpG island 预测软件: www.ebi.ac.uk/emboss/cpgplot/
转录因子搜索程序: transcriptional factor search, TF2 SEARCH: http://www.cbrc.jp/research/db/TFSEA- RCH.html
1.2 方法
1.2.1 建立小鼠和人类IGF1基因的同源基因对
登陆NCBI网站, 在Nucleotide数据库中搜索目标基因 “mouse IGF1 gene”, 在给出的大量相关序列中选择最适合、最符合要求的序列作为目标序列. 本文选择的是小鼠胰岛素样生长因子1 mRNA剪接变异体1序列, 即Mus musculus insulin-like growth factor 1 (IGF1), transcript variant 1, mRNA. 将该基因的cDNA序列(登录号为NM-010512)用BLAST程序先在人类nr数据库中进行比对, 得到一系列与其相匹配的核酸序列, 再从中找出与其匹配程度最高的cDNA序列, 再将该人类cDNA序列重新用BLAST程序与小鼠基因组比对, 找到小鼠中与其匹配程度最高的序列. 如果此序列与最新输入的新基因DNA序列(即小鼠IGF1基因 mRNA剪接变异体1序列)一致, 就将小鼠中的这条序列的编码基因作为该新基因的同源基因. nr数据库比对结果, 人类同源基因GenBank的登录号为NM-001111283.1.
1.2.2 IGF1基因启动子区的确定
将IGF1基因的cDNA序列用BLAST程序在小鼠nr数据库中进行比对, 得到一系列与其相匹配的核酸序列. 从中找出小鼠基因组DNA序列中与其匹配程度最高的同源基因DNA序列, 登录号为NT-039500.7. 确定该同源基因第一个外显子在基因组DNA上的定位, 然后找出第一个外显子5’端上游5 000 bp的序列, 该序列包含着潜在的启动子序列, 登录号为NT-039500.
1.2.3 IGF1基因启动子预测
利用上述各种启动子在线分析软件进行预测.
1.2.4 IGF1基因启动子区转录因子结合位点分析
选用Match1.0程序, 核心序列相似性(core similarity)设定为0.90, 矩阵相似性(matrix similarity)设定为0.95. 输入IGF1基因上游5 000 bp. 搜索TRANSFAC position weight matrix(PWM)数据库中的脊椎动物转录因子结合部位, 获得该基因启动子的转录因子结合结果. 同时使用上述两个程序分别对小鼠和人类同源基因启动子区的转录因子结合位点进行分析, 并搜索出位于两个序列保守区内相同位置的转录因子结合位点. 其余的结合部位作为假阳性被去除, 明显降低了假阳性率, 提高预测准确度.
2 结果
2.1 IGF1基因启动子预测结果
通过GenBank核酸数据库的搜索, 建立新基因在人和小鼠之间的同源基因对, 利用BLAST程序比对人和小鼠基因组数据库, 分别获得该基因第一外显子上游5 000 bp序列. 对小鼠IGF1基因序列第一个外显子上游5 000 bp序列进行3种不同的在线软件分析. 得出潜在的启动子结构(表1). 同时也以同样的方法在线分析预测人类同源基因的启动子区, 不过限于篇幅, 本文不再重复列出其结果. 同时运用3种程序分析预测启动子, 可以提高预测结果的可靠性.
从表1可以看出3种不同的启动子预测软件对同一个基因启动子区进行预测所得的结果都有差异. 这是因为不同程序分析预测启动子的算法不同. 通常确定启动子的算法有很多种, 有的是根据启动子区各种转录信号, 如TATA 盒、CCAAT 盒, 结合对这些保守信号及信号间保守的空间排列顺序的识别进行预测. 例如PROMOTER 2.0, 用神经网络方法确定TATA 盒、CCAAT盒、加帽位点(cap site) 和GC 盒(GCbox) 的位置和距离, 识别含TATA 盒的启动子. PROMOTER SCAN根据转录因子结合部位在基因组中分布的不平衡性,将转录因子结合部位分布密度与TATA 盒的权重矩阵(weight matrix) 结合起来, 从基因组DNA中识别出启动子区. 同时本文还分析预测了该基因CpG island. CpG island经常出现在管家基因或频繁表达的基因的启动子附近, 许多脊椎动物的启动子区都与CpG岛的位置重合. CpG island具有阻止序列甲基化的作用, 而DNA甲基化能够直接抑制基因转录. 从表1可以看出CpG island分布在1 000 bp到2 000 bp的范围, 分布在启动子区附近.

表1 IGF1基因启动子预测结果
2.2 小鼠IGF1基因启动子区转录因子结合部位分析
经Match1.0程序搜索TRANSFAC PWM数据库, 获得小鼠IGF1基因启动子的转录因子结合部位共16个(表2). 经Match1.0程序搜索TRANSFAC PWM数据库, 获得与小鼠IGF1基因同源的人类基因启动子的转录因子结合部位共22个(表3). 再用进化足迹法分析, 利用ConSite程序确定人和小鼠同源基因保守区域, 对比确定小鼠和人IGF1基因保守区域内共有的转录因子结合部位有6个(表4). 可以预测, 6个转录因子结合部位是表达IGF1蛋白所必需的启动子序列. 同时, 这6种人和小鼠共有的转录因子也是哺乳动物在长期进化过程中选择保留下来的IGF1基因表达所不可或缺的反式作用因子.
(1)Ttk 69K: 属于TTK蛋白激酶系列中的一员. 具有C2H2型的锌指基序, 即由2个半胱氨酸(C)和2个组氨酸(H)在一个Zn2+四周形成的的四面体结构域, 基序在锌结合位点上突出形如指状结构, 该结构同DNA螺旋的主沟结合, 识别和结合DNA序列. 该蛋白质参与蛋白质氨基酸磷酸化、有丝分裂纺锤体的组装等过程, 对细胞增殖起正调控作用.
(2)FOXD3: 叉头框D3(forkhead box D3), 属于叉头框蛋白家族的一员. 该蛋白家族是一类DNA结合区具有翼状螺旋结构的转录因子. FOX蛋白不仅能直接对基因进行转录调控, 还能协助其他转录因子参与转录调节. FOXD3对基因转录有正、负双向调控作用, 是维持胚胎上胚层细胞多潜能性和在体外建立胚胎干细胞系所必需的蛋白质.
(3)HNF-4: 肝细胞核因子4(hepatocyte nuclear factor 4), 属于细胞核受体超家族成员, 在肝脏的发育、肝细胞分化成熟过程中起重要调控作用, 同时对细胞的生长和增殖起负调控作用. 若该蛋白的编码基因发生病变, 则有可能会引起非胰岛素依赖型的糖尿病.
(4)Nkx2-5: 果蝇NK2转录因子相关基因座5(NK2 transcription factor related, lous 5). 该蛋白有324个氨基酸组成. 有TN结构域、HD结构域和NK2-SD结构域3类保守结构域, 能参与多种生命活动的调节, 尤其是在心脏的早期发育和在成熟心脏的功能维护中有非常重要的作用. 其基序为螺旋—转折—螺旋(H-T-H)结构.
(5)Elf-1: E74样因子(est域转录因子), 是Est家族的成员之一, 参与发生过程、细胞的有丝分裂原的激活、癌发生以及病毒基因激活等多种生命活动, 基序为H-T-H. Elf-1蛋白全长619个氨基酸, 是T淋巴细胞特异性转录因子, 参与负调控T细胞受体信号转导途径.
(6)Oct-1: Oct-1是POU同源域(homeo domains)蛋白家族中的代表性成员之一. 该蛋白质具有由75个氨基酸构成的N端pou区和由60个保守氨基酸序列构成的C端同源转换区, 对启动子内八聚体元件ATGCAAAT或ATTTGCAT有专一的高度亲和性. 同源转换区的基序为H-T-H结构.

表2 小鼠IGF1基因启动子的转录因子结合部位预测结果

表3 小鼠IGF1基因同源的人类基因启动子的转录因子结合部位预测结果
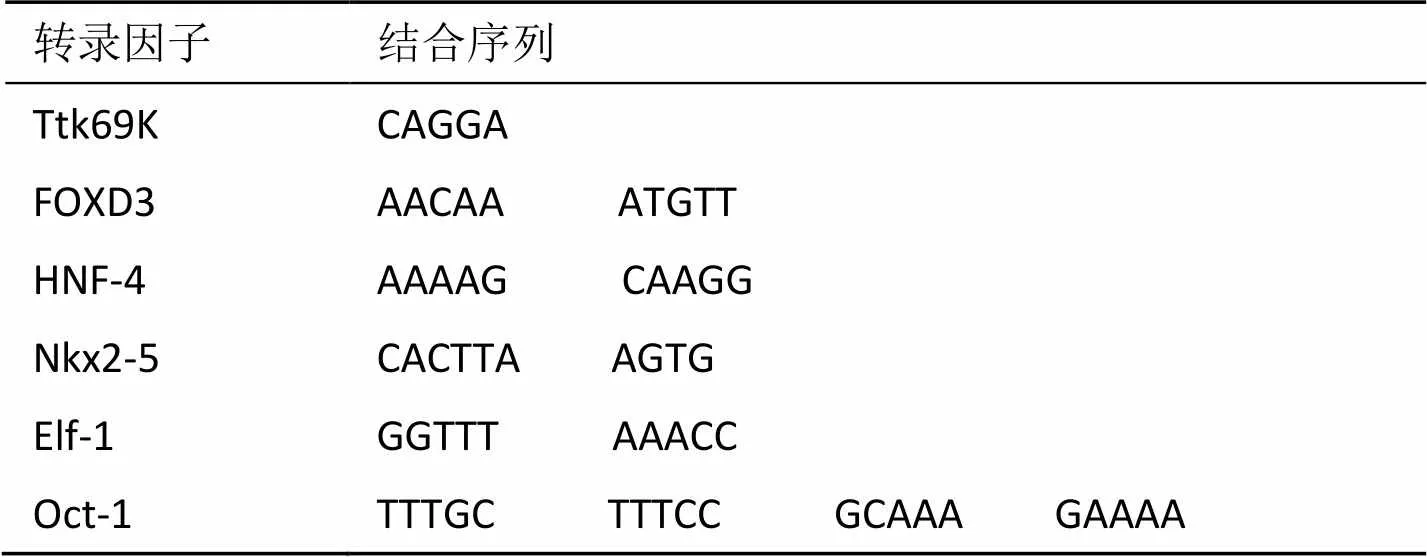
表4 小鼠和人IGF1基因保守区域内共有的转录因子结合部位
3 讨论
基因启动子特异的DNA序列与特定的转录因子的相互作用, 决定下游基因表达的时间、空间和数量等特性, 也是基因表达调控的重要组成部分. 因此进行基因表达调控的研究就需要确定基因序列中的转录因子结合部位. 基因表达的调控区仅占非编码区的一小部分, 全部用实验方法分析显然难以实现. 随着人类基因组及多种模式生物基因组测序工作的完成, 以及各种分析程序的不断完善, 生物信息学已成为基因启动子区转录因子结合部位分析必不可少的工具之一.
利用在线软件可对一段DNA序列进行启动子预测. 如TATA盒、GC盒及转录起始位点(TSS)等, 也可搜索数据库寻找转录因子结合位点.
进化足迹法是利用进化过程中有害突变与中性突变对表型的不同影响, 通过基因组之间的比较, 找到基因组进化中的保守区. 由于位于保守区内的转录因子结合部位更有可能是有功能的部位, 去除非保守区的预测结果就可以明显减少预测结果中的非功能性转录因子结合部位, 降低假阳性率, 使预测结果更为准确. 人和小鼠与他们的共同祖先只有7 500万年的进化距离, 序列间的长远既非常明显又使功能区得以保留. 根据人和小鼠基因组比较的结果, 两者调控区的保守程度低于编码区, 但远高于基因组平均水平, 适于做进化足迹分析. 利用BLAST比对完后, 程序在每个序列中分别搜索转录因子结合部位, 只有那些位于两个序列保守区内的相同位置的结合部位才作为结果输出, 其余的结合部位作为假阳性被去除, 具有较好的特异性[8-13].
本文利用生物信息学技术并结合进化足迹法对小鼠IGF1基因的启动子区进行了分析, 找出了人和小鼠保守区内共有的转录因子结合部位, 为探讨IGF1基因的调控机制和构建组织特异性启动子提供了指导方向, 具有一定的参考价值. 由于数据库搜索分析只是在序列中对那些已知的位点进行查找, 而那些未知的新的转录因子结合部位则不能通过这种方法进行分析, 尚存在不足之处. 此外对所分析的结果还需要实验验证.
利用生物信息学分析出的转录因子并不十分全面, 也存在着不足之处, 尤其是对启动子的分析, 必须将信息学分析和实验分析结合起来才能得到高可靠性的结果. 但随着基因组序列信息的日益丰富, 计算方法和数据库的不断完善, 生物信息学将会得到更加广泛的应用, 基因表达的调控机制也将逐步得到阐明.
[1] 蒋彦, 王小行.基础生物信息学及应用[M]. 北京: 清华大学出版社. 2005.
[2] 朱玉贤, 李毅, 郑晓峰. 现代分子生物学[M]. 3版. 北京: 高等教育出版社, 2007.
[3] 王金发. 细胞生物学[M]. 北京: 科学出版社, 2003
[4] 张成岗, 贺福初. 生物信息学方法与实践[M]. 北京: 科学出版社, 2002.
[5] 郝柏林, 张淑誉. 生物信息学手册[M]. 上海: 上海科学技术出版社, 2000.
[6] 刘进云, 赵广荣等. 转录因子实用技术[M]. 北京: 清华大学出版社, 2005.
[7] 邱明轩, 李峰, 张建宏, 等.NAP1基因和 ARHP基因启动子区的生物信息学分析[J]. 湖南文理学院学报: 自然科学版,2007, 19(3): 37-40.
[8] 刘凤鸣. 人PDCD4启动子的确定及其转录调控的初步研究[D]. 山东大学, 2010.
[9] 赵黎黎. 小鼠TLE4基因启动子克隆及分析[J]. 畜牧兽医学报, 2010 , 41(3): 274-278.
[10] T.K.Attwood, D.J.Pasrry-Smith著, 罗静初等译. 生物信息学概论[M]. 北京: 北京大学出版社, 2002
[11] 管晓翔, 陈巍魏, 陈龙邦, 等.p27Kip1基因启动子区的生物信息学分析[J]. 医学研究生报, 2010, 23(10): 1029-1032.
[12] 何玮玮, 管晓翔, 陈龙邦. Ⅰ、Ⅱ期乳腺癌组织中p27K ip1的表达及其临床意义[J]. 医学研究生学报, 2009, 22(3): 252-259
[13] 卜友泉, 宋方洲, 易发平, 等. 一个新的人NFBD1启动子的鉴定与分析[J]. 第四军医大学学报, 2007, 28(22): 2020-2024.
Bioinformatics analysis of Mus. IGF1 Gene gene promoter regions
LI Feng, ZHANG Yu, WANG Xing-ping, LUOREN Zhuoma , LI Rong
(Department of Biological Science, Hunan University of Arts and Science, Changde 415000, China)
Bioinformatics tools and phylogenetic foot-printing were used to predict the transcriptional factors binding sites in the promoter regions of IGF1 gene. Promoters were analyzed by on-line software. By several online soft wares, 6 transcription factor binding sites were predicted in the promoter conserved region of IGF1 gene. Analysis of IGF1 gene’s promoter regions may prove a model gene with which to study the regulatory mechanisms, and has important significance in transcriptional regulation research.
IGF1 gene; phylogenetic foot-printing; transcriptional factor binding sites; promoter regions; Bioinforma- tics analysis
10.3969/j.issn.1672-6146.2012.03.012
Q 34
1672-6146(2012)03-0040-06
2012-08-15
湖南省自然科学基金项目(08JJ3037), 湖南省高校创新平台开放基金项目(09K097).
李峰(1966-), 男, 教授, 研究方向: 蛋白质结构与功能. E-mail: youquanli@126.com
(责任编校:谭长贵)
