基于遗传优化获取微阵列最佳分类规则*
2012-03-06陈湘涛
陈湘涛,陈 东
(湖南大学信息科学与工程学院,湖南长沙 410082)
基于遗传优化获取微阵列最佳分类规则*
陈湘涛†,陈 东
(湖南大学信息科学与工程学院,湖南长沙 410082)
基于遗传编程(GP)提出一种最优规则遗传算法(BRGA)对分类规则进行优化的方法,获取最佳分类规则集,此算法可以调整分类器模型的相关参数,在适当增加迭代基础上大幅提高分类的精确度,具有相当的灵活性和可理解性.利用6个基因数据集检验了算法的性能.仿真结果表明,本文提出的算法与其他文献的方法相比,在具有较高分类精确度和稳定性前提下大幅降低了计算复杂度及冗余.
最优规则遗传算法;微阵列;遗传编程;分类规则;计算复杂度
生物医学研究表明,人类大多数疾病的发病机制,比如癌症,从根本上来说都和基因息息相关.微阵列数据是将样本实验形成的影像转为基因表达矩阵,矩阵行表示基因,列表示类别样本,矩阵中的元素描述不同基因在不同样本的表达水平.
由于微阵列芯片技术[1]获得的基因数据数量远大于样本数量,随着维数的增加,最大的障碍则是在高维特征空间运算时存在的“维数灾难”.微阵列大量基因数据仅为样本分类提供了少数有分类意义的、具有明显特征的基因.因此,在样本分类之前,选择特征基因是至关重要的,这直接影响到之后生成的分类器性能.微阵列分类作为生物指标的探索成为生物信息学一个重要的课题,事实上,由于存在更多的癌症类型和潜在的癌症子类,如果展开肿瘤分类问题到多重肿瘤类别,数据集包含更多的类别和非常少量的样本,问题将变得更具有挑战性.
一些研究报告指出,在基因选择部分使用遗传算法能改进微阵列数据的分类性能[1-2],因此,遗传算法已广泛用于解决包括数据分类的各种难题[3-4].本文提出一种最优规则遗传算法(Best Rule Genetic Algorithm,BRGA),选用一种基于遗传优化的分类算法生成分类规则,用二进制向量表示分类规则,初始化规则集,设定相应的适应度及初始种群的规模,通过变异产生一定数量的最优分类规则.通过实验,使用6个基因表达数据集来验证算法的性能.
微阵列数据分类技术通常包含2部分内容:1)基因选择;2)构建分类器模型.文献[5]在基因选择部分使用排列值计分RBS算法,很好地解释了基因之间的相关性,大幅降低基因矩阵维度,在一定程度上减少了计算复杂性;在构建分类器部分提出了LCR方法,可以用很少的基因构造形成分类规则,提高了算法的可理解性.但分类规则的形成过程仍存在很多不足,如分类器模型中规则形成框架过于缜密,容易导致过拟合,产生庞大规则集的迭代过程相当繁琐,并产生大量冗余的规则,导致计算复杂度较高且算法收敛速度较低.分类器的构建则是整个技术的核心所在,传统的微阵列分类方法有:加权投票(WV)[6],K近邻(k-NN)[7],支持向量机(SVM)[8],费舍尔线性判别分析(LDA)[9],人工神经网络(ANN)[10],遗传规划(GP)[11],最小二乘逻辑回归[12]和朴素贝叶斯方法[13]等.由于它们仅仅聚焦于分类性能,而不能进一步提供任何医学和生物学依据,导致这些分类算法往往产生僵硬的分类系统,存在稳定性弱和开销大的特征,缺乏可扩展性.决策树算法[14]和随机森林算法[15]基于决策规则产生分类器模型,此类算法获得的分类规则在某种意义上包含了生物体基因之间的相关性,但如果训练样本存在小的差异会导致决策树结构产生大的变化,致使分类器缺乏稳定性,这些分类方法仍然存在很大的局限性.
1 BRGA方法的基本思想
BRGA算法是在遗传优化的基础上,将分类规则集作为种群,使用二进制串表示其中任意一条分类规则,计算对应于基因属性的比较关系的分类规则适应度值,经过若干代的繁殖过程,包括选择、交叉和变异运算,反复迭代优化,获取具有较高适应度的最佳分类规则.
1.1 基因选择
本文用BRS方法[5]对微阵列数据集按排列值打分排序,分别选择分值最高的c/2和最低的c/2个(c为偶数)有重大表达意义的基因.
1.2 分类规则形成
首先将基因选择部分产生的c个基因的排列值矩阵R按行建立比较关系,规则表示为[b1b2…bc]且b1,b2,…,bc∈{0,1},如果比较关系中包含矩阵J中第i行基因排列值,则bi∈{b1,b2,…,bc}为1,反之bi为0.然后获取满足规则关系的癌样本和正常样本的数量,分别计算其对应类别样本总数的比值Ct和Cn,二者之差的绝对值作为适应度F,如果Ct大于Cn,表示该规则为癌类别,用1表示;相反,该规则为正常类别,用0表示.利用BRGA算法优化初始规则集,从而获取最佳分类规则集.
设Ft为适应度阈值,适应度f大于Ft的规则为最优规则;Nt为最优规则数量阈值,最优规则集规则数m达到值Nt则收敛;K为规则种群繁殖的代数,每经过K代则将适应度阈值Ft下调一个合适的值F_ad,以防止算法无法收敛.
1.3 分类预测
最佳分类规则集用来预测测试样本的类别,计算测试样本对应规则中基因的排列值,判断规则集中每条分类规则是否满足测试样本,依次将测试样本归类,如果满足癌样本分类规则,将其归为癌类别1,如果不满足癌样本分类规则,则归为正常类别0,反之亦然.采用MV[5](Majority Voting)方法累计不同类别的规则适应度值,如果某类的适应度值总和大于另一类适应度值总和,那么最终预测测试样本属于该类别.
2 BRGA算法优化过程
将微阵列基因数据训练样本的基因表达值矩阵转换为其相应的排列值矩阵形式,用BRS方法打分,再排序,取分值最高和最低的能表达样本特征的少量基因.然后用BRGA算法产生符合特征基因排列值对应不相同类别的样本大小关系的规则,其中,符合某一样本类别不同基因排列值大小关系的规则应尽量不符合另一样本类别的相应基因排列值的大小关系,找出所有这样的规则,再用这些规则生成分类器对未知样本进行分类预测.
2.1 规则的形式
在少量特征基因的排列值矩阵形成的基础上,根据矩阵的维度M确定二进制规则的位数为M,R1,R2,…,RM对应排列值矩阵从上至下相应的行(1,2,…,M),规则中1表示矩阵对应二进制规则相应位置的行与其他行存在比较关系,0则表示在此规则中该行不存在比较关系.
例如,规则的位数固定为M位,矩阵排列值行比较关系R1<R2<R5;M=10,那么此关系可表示为[1 1 0 0 1 0 0 0 0 0],比较关系方向统一为大于或小于符号,R1,R2,R5表示每一样本在第1,2,5个基因位置相应的排列值.
2.2 规则的生成
BRGA方法基于遗传算法优化分类规则,将具有高适应度的分类规则归并至最佳分类规则集,最终达到最优的样本预测性能,极大地提高了算法的收敛性,BRGA算法伪代码示意图如图1所示.BRGA算法的步骤为:
1)初始化规则集.生成初始规则种群为M*N阶全零矩阵形式Popus,其中每一条规则rule(i)是位数为N,且值全为零的一维数组,且i∈{1,2,…,M},设定合适的适应度阈值Ft,最佳规则集规则数阈值Nt及K代调整系数F_ad.
2)变异运算.变异运算aberrance(popus)是随机改变种群规则中某一位置的值,通过对父代规则集变异形成子代规则集,由于微阵列数据的特征基因排列值在不同样本中差异较大,规则适应度值随比较关系所包含基因数增加基本呈递减趋势,因此,BRGA算法在变异运算中设置了限制规则中1的数量(即比较关系的基因数)阈值Lt,如果超过Lt将规则位全部清零,从而降低冗余规则的产生,并能有较防止过拟合,提高了分类性能.
3)计算规则适应度.函数Fit()计算Popus规则种群中每一条规则对应的适应度F,输出对应的规则rule及类别标签label.

图1 BRGA算法伪代码示意图Fig.1 BRGA pseudo-code schematic diagram
假设任一规则的适应度为F,如果满足该规则的某类样本的数量为a,另一类样本的数量为b,那么,该规则相应的适应度为F=|a-b|.很容易看出F值越大,则越能体现不同样本的差异.
那么,规则预测类别标签label可以表示为:如果a>b,样本为A类,比如癌症样本,则label=1;如果a<b,样本为B类,如正常样本,则label=0.
4)获取最佳规则.计算子代规则集的规则适应度F,如果F{F1,F2,…,Fn}中存在大于等于Ft的值,则将其对应的规则、适应度值及预测类别标签label保存至最佳规则集,去掉相同的规则,每隔K代,适应度阈值Ft向下调整幅度值F_ad,确保算法收敛,并判断规则数量是否等于Nt,如果相等则结束,否则重复步骤2)-4).
3 实验结果与分析
本文实验使用了公开的6个基因表达数据集,分别为前列腺癌[16],结肠癌[17],白血病[18],淋巴瘤[19],肺癌[20]和脑癌[21],这些数据集为二类和多类基因表达谱.仿真实验所使用的软件系统为MATLAB7.0,硬件环境为主频2 GHz的Intel双核微处理器、内存为2 GB的PC机.使用结肠癌数据集阐述BRGA算法实施过程,该数据集包含62个样本,2 000个基因表达水平,样本由40个肿瘤和22个正常样本组成.结肠癌基因表达值如表1所示.
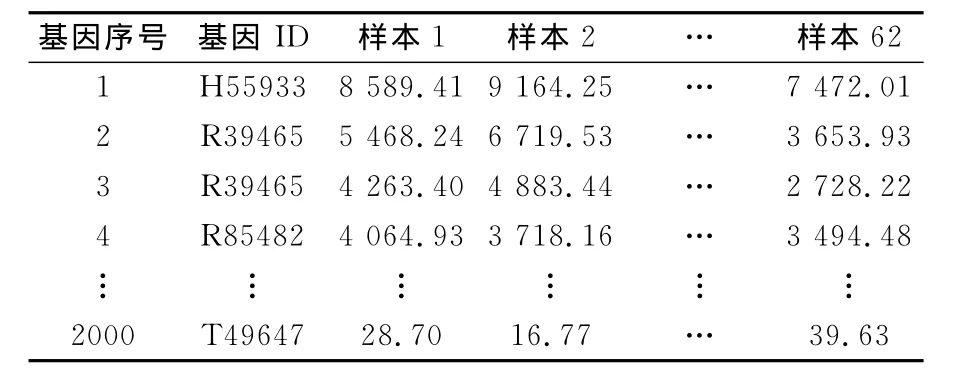
表1 结肠癌基因表达值Tab.1 Colon cancer gene expression values
在基因选择部分利用文献[5]使用的RBS方法将每个样本的每一基因表达值转换成相应的排列值(见表2).
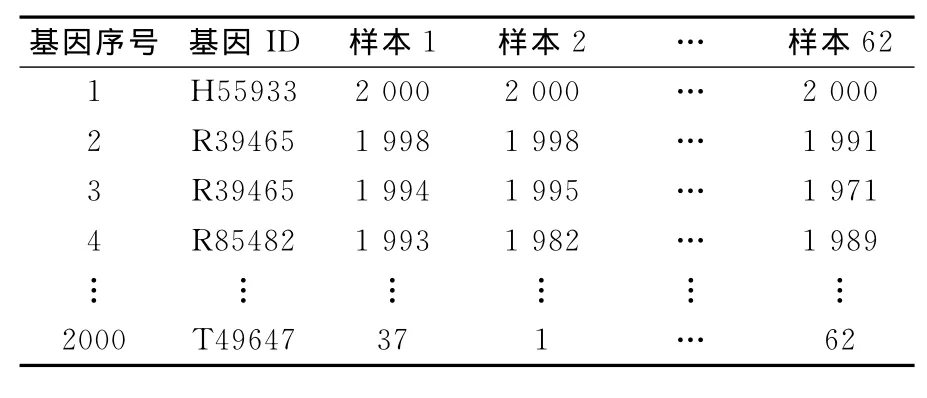
表2 结肠癌基因排列值Tab.2 Colon cancer gene rank values
从分类性能和运算耗时多方面因素综合考虑,在本实验中选择RBS分值最高和最低的具有重大表达意义的属性基因各10个(共计20个基因),作为形成分类规则的基础,如表3所示.
使用本文第2节提到的方法来初始化规则集,选择合适的适应度阈值Ft∈(0,1]及规则数阈值Nt,同时设定规则中比较关系所包含的基因数量Lt,通过对结肠癌数据集选择的上述20个重要的基因实施该方法,可获得一个具有一定数量Nt的最优预测规则数据集(包含类标签为1的癌症类标签,为0的正常类标签以及适应度值F),且每一规则适应度值均大于或等于Ft,同时,设定每隔K代Ft调整值F_ad,确保算法收敛于最优规则集.
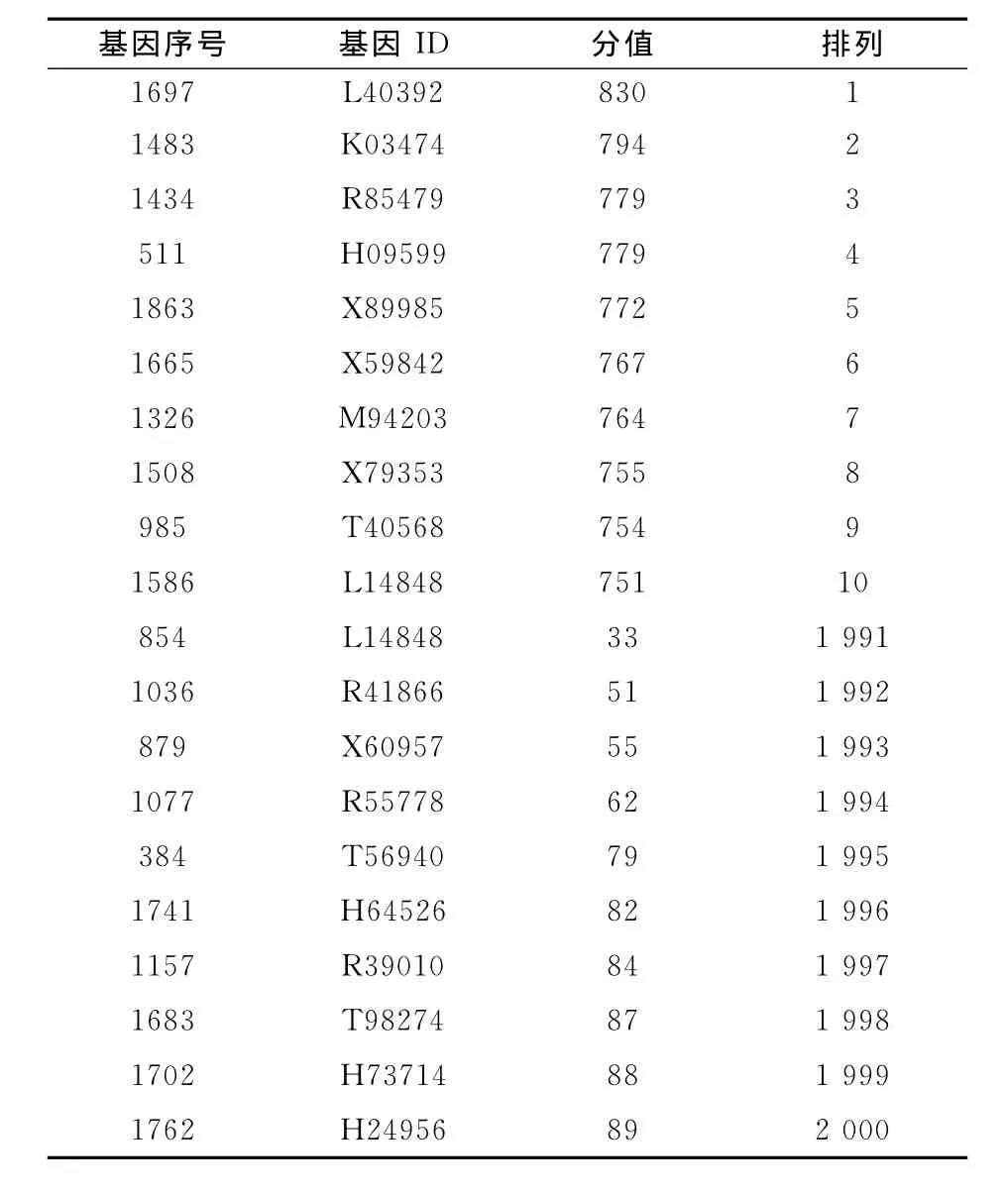
表3 20个结肠癌重要基因Tab.3 Twenty significant Colon cancer genes
用留一交叉校验法(Leave-One-Out Cross Validation,LOOCV)验证规则集对结肠癌数据集分类的性能,将62个样本分为训练集和测试集,包含61个训练样本和单个测试样本,此过程重复62次.对样本集中每个样本分别作一次预测,以此来检验算法的准确性.最后用规则集对每个样本分别累计其满足癌症类规则适应度与正常类规则适应度之和,两者相减,如果癌症类规则适应度大于正常规则适应度,则预测此样本属于癌症样本,反之为正常样本.表4为使用4条规则的最优规则集预测结肠癌测试样本的类别.
用同样的方法对另外3个两类数据集进行分类预测,得到LOOCV精确度与运算时间与文献[5]比较结果(见表5).
采用BRGA算法用于多类(n类)数据集分类时,先将正常样本和癌样本分为2大类,再依次把训练集中癌样本(n-1子类)的每一子类和剩余其他子类(n-2子类)的样本分为两类,用BRGA算法优化规则获得n个最佳分类规则集,即把多类问题转化为n个两类问题,再对n个两类最佳规则集的预测精确度求平均值,得到最终的分类性能.将BRGA算法和几种相关文献中提及的分类方法在性能上进行比较,结果如表6所示.

表4 结肠癌样本预测Tab.4 Colon cancer sample prediction

表5 算法性能及运算耗时比较Tab.5 Performance and elapsed time comparison

表6 算法性能比较Tab.6 Performance comparison
由表5和表6可以发现,本文提出的算法与其他文献中的算法比较具有较高的分类性能,且本文算法能快速地从复杂基因排列中提取最优的分类规则,极大地降低了计算复杂度及运算时间;算法简单易理解,稳定性高,对肿瘤和正常基因的分类及疾病的不同子类的划分都具有优良的性能.
图2为BRGA算法在6个不同数据集对应不同最优规则数量阈值Nt的分类预测性能分布图.由图1可知,当Nt值为5~10时,大部分数据集均能较好地收敛于最佳分类规则集,达到相对理想的分类性能;BRGA算法在二类和多类微阵列基因数据集的分类性能均能在合适的Nt值情形下达到高分类精确度,降低了冗余规则在分类规则中出现的概率,并具有较高的收敛性和较强的泛化能力.
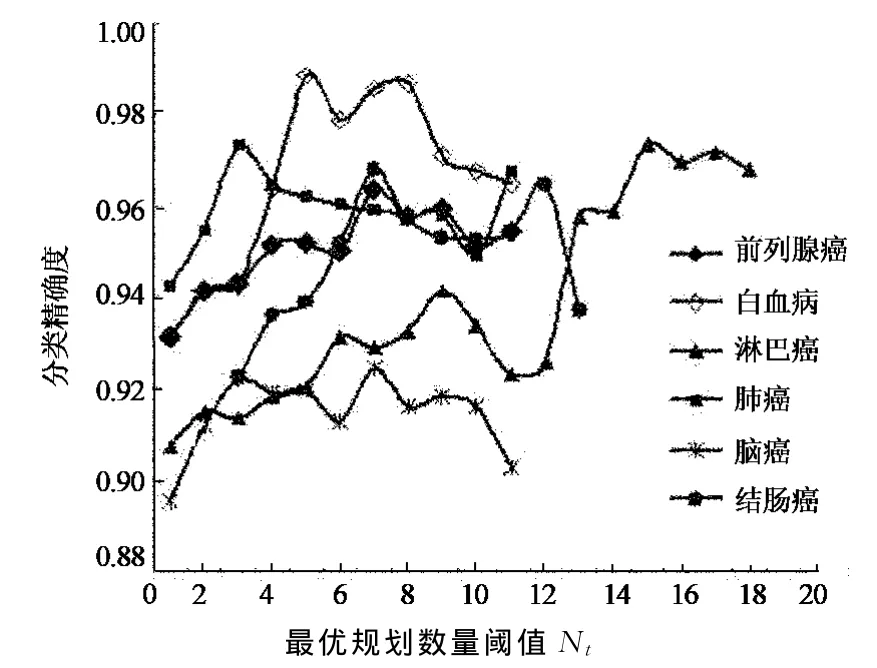
图2 BRGA性能分布图Fig.2 BRGA performance distribution map
4 结 论
本文提出的BRGA算法很好地解决了用微阵列基因表达值构建分类决策规则普遍速度慢的难题,通过调整适合规则的适应度值及相关参数对初始规则集进行优化,该算法能很快收敛于最优分类规则集.采用6个数据集验证了该算法的性能,实验结果表明,BRGA算法具有较高的精确度和极少的分类运算耗时(CPU time).当然,由于实验条件和生物学发展的局限性,该算法有待进一步提高和完善.
[1] HENGPRAPROHM S,MUKVIBOONCHAI S,THAMMASANG R,etal.A GA-based classifier for microarray data classification[C]//Proceedings of 2010 International Conference on Intelligent Computing and Cognitive Informatics(ICICCI 2010).Kuala Lumpur:IEEE Computer Society,2010:199-202.
[2] OOI C H,TAN P.Genetic algorithms applied to multi-class prediction for the analysis of gene expression data[J].Bioinformatics,2003,19(1):37-44.
[3] BANDYOPADHYAY S,MURTHY C A,PAL S K.Pattern classification with genetic algorithms[J].Pattern Recognition Letters,1995,16(8):801-808.
[4] BANDYOPADHYAY S,MURTHY C A,PAL S K.VGA-classifier:design and applications[J].IEEE Transactions on Systems,Man and Cybernetics-Part B,Cybernetics,2000,30(6):890-895.
[5] GANESH-KUMAR P,AMMU V,VICTOIRE T A A.Building decision rules using a novel data driven method for microarray data classification[C]//2011 International Conference on Process Automation,Control and Computing(PACC 2011).Coimbatore:IEEE Express Conference Publishing,2011:1-6.
[6] GOLUB T R,SLONIM D K,TAMAYO P,et al.Molecular classification of cancer:class discovery and class prediction by gene expression monitoring[J].Science,1999,286(5439):531-537.
[7] WU Wei,XING E P,MYERS C,et al.Evaluation of normalization methods for cDNA microarray data by k-NN classification[J].BMC Bioinformatics,2005,6:191-211.
[8] YOONKYUNG L,CHEOL-KOO L.Classification of multiple cancer types by multicategory support vector machines using gene expression data[J].Bioinformatics,2003,19(9):1132-1139.
[9] JAEWON L,JUNGBOK L,MIRA P,et al.An extensive comparison of recent classification tools applied to microarray data[J].Computational Statistics &Data Analysis,2005,48(4):869-885.
[10]KHAN J,JUN S,RINGNER M,et al.Classification and diagnostic prediction of cancers using gene expression profiling and artificial neural networks[J].Nat Med,2001,7(6):673-679.
[11]HONG J H,CHO S B.The classification of cancer based on DNA microarray data that uses diverse ensemble genetic programming[J].Artificial Intelligence in Medicine,2006,36(1):43-58.
[12]TANG E K,SUGANTHAN P N,YAO X.Gene selection algorithms for microarray data based on least square support vector machine[J].BMC Bioinf,2006,7:95-110.
[13]JOHNSON W E,LI C,RABINOVIC A.Adjusting batch effects in microarray expression data using empirical bayes methods[J].Biostatistics,2007,8(1):118-127.
[14]YU WANG,IGOR V T,MARK A H,et al.Gene selection from microarray data for cancer classification—a machine learning approach[J].Computational Biology and Chemistry,2005,29(1):37-46.
[15]RAMON D U,SARA A A.Gene selection and classification of microarray data using random forest[J].BMC Bioinformatics,2006,7:3.
[16]WELSH J B,SAPINOSO L M,SU A I,et al.Analysis of gene expression identifies candidate markers and pharmacological targets in prostate cancer[J].Cancer Res,2001,61:5974-5978.
[17]ALON U,BARKAI N,NOTTERMAN D A,et al.Broad patterns of gene expression revealed by clustering analysis of tumor and normal colon tissues probed by oligonucleotide arrays[J].PNAS,1999,96(12):6745-6750.
[18]Broad Institute.Cancer program data sets[EB/OL].[2012-01-01].http://www.broadinstitute.org/cgi-bin/cancer/datasets.cgi.
[19]ASH A A,MICHAEL B E,ERIC R D,et al.Distinct types of diffuse large B-cell lymphoma identified by gene expression profiling[J].Nature,2000,403(4):503-511.
[20]BHATTACHARJEE A,RICHARDS W,STAUNTON J,et al.Classification of human lung carcinomas by mRNA expression profiling reveals distinct adenocarcinoma subclasses[J].PNAS,2001,98(24):13790-13795.
[21]SCOTT L P,PABLO T,MICHELLE G,et al.Gene expression-based classification and outcome prediction of central nervous system embryonal tumors[EB/OL].[2012-01-01].http://www.broadinstitute.org/mpr/CNS/.
[22]SHI Chao,CHEN Li-hui.Feature dimension reduction for microarray data analysis using locally linear embedding[C]//Proceedings of 3rd Asia-Pacific Bioinformatics Conference(APBC 2005).Singapore:Imperial College Press,2005:211-217.
[23]TAN A,NAIMAN D,XU L,et al.Simple decision rules for classifying human cancers from gene expression profiles[J].Bioinformatics,2005,21(20):3896-3904.
[24]FUREY T S,CRISTIANINI N,DUFFY N,et al.Support vector machine classification and validation of cancer tissue samples using microarray data[J].Bioinformatics,2000,16(10):906-914.
[25]JUNBAI W,TROND H B,INGE J,et al.Tumor classification and marker gene prediction by feature selection and fuzzy c-means clustering using microarray data[J].BMC Bioinformatics,2003,4:60-71.
Obtaining Optimal Microarray Data Classification Rule by GA-based Optimizing
CHEN Xiang-tao†,CHEN Dong
(College of Information Science and Engineering,Hunan Univ,Changsha,Hunan 410082,China)
Based on Genetic Programming(GP),this paper proposed an approach called Best Rule Genetic Algorithm(BRGA)for optimizing classification rule,and gained the best classification rule set.This algorithm can adjust relevant parameters of classifier model,substantially improve the performance of classification by increasing appropriate iteration,therefore it has considerable flexibility and intelligibility.The performance of the proposed approach was evaluated by using six gene expression data sets through simulation.From the result,it is found that the proposed approach reduces computational complexity and redundancy with good classification accuracy and stability than approaches reported in other literatures so far.
Best Rule Genetic Algorithm(BRGA);microarrays;Genetic Programming(GP);classification rule;computational complexity
TP391
A
1674-2974(2012)08-0081-06*
2011-12-19
国家林业公益性行业科研专项经费资助项目(201104090)
陈湘涛(1973—),男,湖南新宁人,湖南大学副教授,博士
†通讯联系人,E-mail:xtchen2009@163.com
