蛋白质剪接在蛋白质研究和蛋白质工程中的应用
2012-01-24戴旭东刘相钦
戴旭东 孟 清 刘相钦
①博士研究生,②教授,东华大学生物科学与技术研究所,上海 201620;③教授,Department of Biochemistry &Molecular Biology,Faculty of Medicine,Dalhousie University,Halifax,Nova Scotia,Canada B3H1X5
蛋白质剪接(protein splicing)是由蛋白质内含子介导的在蛋白质水平上翻译后的加工过程。蛋白质内含子(intein)是指前体蛋白中的一段插入序列,它在蛋白质翻译后的成熟过程中能自我催化,使自身从前体蛋白中切除,并将其两侧的多肽片段以肽键连接,形成成熟的蛋白质。蛋白质内含子的发现丰富了基因表达和蛋白质翻译成熟过程的理论,而且在蛋白质研究和蛋白质工程中有广泛的应用。本文试图综述蛋白质剪接在蛋白质特异位点标记、蛋白质片段化标记同位素、蛋白质环化、蛋白质芯片、基因治疗等研究中的应用。
蛋白质内含子(intein)是指前体蛋白中的一段插入序列,它在蛋白质翻译后的成熟过程中能自我催化,使自身从前体蛋白中切除,并将其两侧称为蛋白质外显子(extein)的多肽片段以正常的肽键连接形成有功能的成熟蛋白质[1-2]。截至到2011年8月,已经有500多种蛋白质内含子(intein)被发现(inbase,http://www.neb.com/neb/inteins.html)[3]。它们分布在单细胞真核生物、细菌、古细菌、噬菌体和病毒的基因组中,但是在多细胞生物中尚未发现蛋白质内含子,而且它们主要分布在与DNA复制、转录、翻译有关的蛋白酶或转移因子上,这可能有利于蛋白质内含子的归巢(homing)和生存[4]。由于蛋白质剪接能把两个多肽以一个天然肽键相连(splicing),形成成熟的有活性的蛋白质,为蛋白质合成和翻译后修饰提供了新的途径,因而蛋白质剪接已经被广泛的应用到蛋白质研究和蛋白质工程中。例如,这项技术可以应用到蛋白质特异位点标记、蛋白质片段化标记同位素、蛋白质环化、基因治疗、蛋白质芯片等研究中[5-7]。
1 蛋白质内含子的生物学特性
1.1 蛋白质内含子
1990年,分别由Kane和Hirata领导的研究小组同时在酵母中发现蛋白质剪接(protein splicing)[8-9]。1994年,Perler将“插入片段”正式命名为“intein”,即蛋白质内含子。并将其定义为:在蛋白质成熟过程中被剪接掉的一段框内融合于前体蛋白的氨基酸序列,蛋白质内含子两端的序列称为“extein”,即外显子[10](图1)。
蛋白质内含子不同于RNA内含子,前者存在于蛋白质,后者则存在于mRNA前体。然而,蛋白质内含子与RNA内含子(intron)也有相似之处。首先,它们都插入到基因中,通过自身剪接从前体中剪接下来,将两边的前体连接。另外,二者都包含核酸内切酶结构域,可以通过类似的归巢机制在基因中转移。
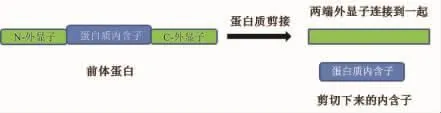
图1 蛋白质剪接示意图[1]
1.2 蛋白质内含子的结构
每一个蛋白质内含子都包含N端剪接结构域和C端剪接结构域。N端剪接结构包含两个序列模块即A和B,C端剪接结构包含两个序列模块即F和G。除此之外,标准蛋白质内含子在N端剪接结构域和C端剪接结构域之间还存在归巢核酸内切酶结构域,由模块C,D和E组成(图2)。模块A位于蛋白质内含子的N端,其N端的第一个氨基酸残基非常保守,一般是Cys或Ser;模块G位于蛋白质内含子的C端,其C端的第一个氨基酸残基非常保守,一般是Asn,第二个氨基酸残基也非常保守,一般是His;模块B中Thr-X-X-His序列的Thr和His非常保守,X是任何氨基酸残基;另外,C端蛋白质外显子的第一个氨基酸残基非常保守,一般是Cys,Ser或Thr。越来越多的证据,特别是突变分析和一些蛋白质内含子晶体结构的解析表明这些保守的氨基酸残基参与了蛋白质的剪接,如果它们发生突变,将会影响蛋白质剪接活性[11]。

图2 标准蛋白质内含子结构示意图[1]。两端分别是蛋白质内含子的剪接结构域,中间部分是归巢核酸内切酶结构域。A,B,C,D,E,F,G是蛋白质内含子的保守模块。
1.3 蛋白质的顺式剪接(cis-splicing)与反式剪接(trans-splicing)
蛋白质内含子根据其结构特征可分为三类:标准蛋白质内含子(canonical intein),微小蛋白质内含子(miniintein)和断裂蛋白质内含子(split intein)(图3)。标准蛋白内含子含有大约400个氨基酸并包括一个归巢核酸内切酶结构域,而微小蛋白质内含子只有大约140个氨基酸不包含归巢核酸内切酶结构域,只有蛋白剪切的功能。虽然很多蛋白内含子在序列上没有同源性,但是它们的晶体结构非常接近。微小蛋白质内含子的晶体结构包括12个β折叠,它们形成一个圆盘状的蛋白质,其剪切活性中心位于圆盘的中央[11-12]。对于标准蛋白质内含子和微小蛋白质内含子来说,蛋白质内含子和蛋白质外显子位于同一多肽链上,这时发生的蛋白剪接称为顺式剪接(cis-splicing)。断裂蛋白质内含子是蛋白质内含子中部区域特定位点发生断裂,被分裂成两部分即N端区域(IN)和C端区域(IC),分别由不同的开放阅读框(ORF)编码。断裂蛋白质内含子的IN和IC在剪接时相互识别,然后重建催化活性中心,进而发生蛋白质反式剪接(protein trans-splicing)。

图3 蛋白质的顺式剪接(cis-splicing)与反式剪接(trans-splicing)。IN:蛋白质内含子N端剪接结构域,IC:蛋白质内含子C端剪接结构域,EN:归巢核酸内切酶结构域。
1.4 蛋白质内含子的剪接机制
蛋白质内含子介导的蛋白质剪接是一个自我催化过程,只与蛋白质内含子剪接活性中心的保守氨基酸和C端外显子的第一个氨基酸有关。内含子可以不依赖其他任何因素催化蛋白质就可以剪接。
蛋白质顺式剪接(cis-splicing)包括4步亲核反应(图4)[13-14]。第一步,蛋白质内含子 N-端的第一个保守氨基酸Cys或Ser引起酰基化重排(acyl rearrangement),N-端蛋白质外显子与蛋白质内含子之间的肽键被打开,形成硫酯键或酯键。第二步,C-端蛋白质外显子的第一位氨基酸Cys,Ser或Thr的巯基或羟基攻击第一步反应形成的硫酯键或酯键,导致N-端蛋白质外显子以硫酯键或酯键连接到C-端蛋白质外显子的第一位氨基酸残基的侧链上。蛋白质内含子的N-端剪接点被切断,形成分支中间体(branched intermediate)。第三步,蛋白质内含子C-端的Asn或Gln侧链上的酰胺基攻击C-端剪接点的肽键,导致该剪接点断裂。至此,蛋白质内含子与两侧的蛋白质外显子分开。第四步,通过自发的S-N或O-N之间的酰基化重排,两个蛋白质外显子之间的硫酯键或酯键转变成正常的肽键。这样,就完成了标准的蛋白质剪接反应。
断裂蛋白质内含子介导的反式剪接(trans-splicing)起始于断裂蛋白质内含子IN和IC的相互识别和结合。IN和IC紧密结合后,断裂蛋白质内含子正确折叠而重建活性中心,之后按照标准蛋白质剪接途径完成蛋白质外显子的连接[15-17]。

图4 标准蛋白质内含子剪接机制[18]
2 天然断裂蛋白质内含子和人工断裂蛋白质内含子
2.1 寻找天然断裂蛋白质内含子
蛋白质反式剪接(trans-splicing)是基于断裂蛋白质内含子的一种剪接方式。断裂蛋白质内含子分为两种:一种是天然存在的,另一种是通过基因工程的方法断裂微小蛋白质内含子的方式获得。1998年,Paul Liu实验室在蓝藻sp.PCC6803的DnaE蛋白中首次发现了天然断裂蛋白质内含子,IN和IC在不同的阅读框中表达,二者在基因组中相距745226 bp[15]。这种两段蛋白组成的断裂蛋白质内含子在许多蓝藻中也被发现[19-20]。天然断裂蛋白质内含子的发现,暗示了标准蛋白质内含子在进化中丢失了归巢内切酶结构域,导致两端的剪接结构域在不同的阅读框中表达,而且这两段蛋白可以重新组装成为有活性的蛋白质内含子,这为构建人工断裂蛋白质内含子提供了理论依据。
2.2 构建人工断裂蛋白质内含子
归巢核酸内切酶结构域的作用是使蛋白质内含子基因可以在基因组中自由转移,与蛋白质剪接功能无关,而且科学家们还发现有些蛋白质内含子去除核酸内切酶结构域后也有剪接活性[15,17,21],蛋白质内含子的晶体结构解析也证明了核酸内切酶是独立的结构域,不参与蛋白质剪接[22]。所以科学家们通过基因工程的方法去除标准蛋白质内含子的归巢核酸内切酶结构域,并以此为断裂位点将微小蛋白质内含子断裂,使IN和IC在不同的阅读框中表达,构建了一系列人工断裂蛋白质内含子[17,23-24]。
2.3 新型断裂蛋白质内含子
典型蛋白质内含子去除归巢核酸内切酶结构域后,N端有110~130个氨基酸,C端有35~40个氨基酸。在蛋白质工程和蛋白质研究中,蛋白质内含子可以被应用于蛋白质的半合成。因为现有的多肽合成技术不能合成多于100aa的多肽,所以只有C端可以通过化学方法合成,而N端不可以通过化学合成的方式获得。
为了将N端合成多肽也能连接到与其互补的重组表达蛋白上,Paul Liu实验室首先在典型断裂位点(S0)以外设计了一系列断裂位点(图5),其中S0是归巢核酸内切酶的位置,其余大部分断裂位点是在预测的β-折叠之间的环状区域[25]。通过他们的测试,发现蛋白质内含子的四个断裂位点(S1,S6,S7和S8)具有蛋白质反式剪接活性。其中S1型的IN只有11个氨基酸,IC有125个氨基酸。随后,Paul Liu实验室又验证了在微小蛋白质内含子Ssp GyrB中S11断裂位点处有高剪接活性[26]。S11型断裂蛋白质内含子的IC只有6个氨基酸,IN有150个氨基酸。
新型断裂蛋白质内含子S1和S11的构建拓宽了蛋白质剪接在蛋白质研究和蛋白质工程中的应用。可以把荧光基团、多聚物等化学修饰分子直接通过合成到化学合成多肽上,通过反式剪接把带有蛋白修饰分子的多肽连接到重组蛋白上。新型断裂蛋白质内含子的构建成功,使断裂位点不再局限于S0位点,为人们设计断裂蛋白质内含子拓宽了思路。由于化学法合成多肽对氨基酸数目有限制,所以蛋白质内含子IN和IC的氨基酸数目越少,那么与IN或IC一同合成的目的蛋白的氨基酸数目就越多,寻找更小的IN或IC仍然是蛋白质内含子研究的一个方向。

图5 断裂蛋白质内含子序列中的断裂位置。154 aa长的微小蛋白质内含子Ssp DnaB,每隔10个位置空一格排列,选出的13个断裂位点(S1-S13)用箭头在上方标示,S0是归巢核酸内切酶结构域的位置,是典型的断裂位点。下划线的是β-折叠(β1-β12)[25]。
3 蛋白质剪接的应用
蛋白质剪接的独特性能是它能将两个多肽以一个天然肽键相连(splicing),为蛋白质合成和修饰提供了新的途径,因而在蛋白质工程及其他领域具有广泛的应用前景。在蛋白质反式剪接中,断裂蛋白质内含子的两个片段可以分别表达或合成,每个片段自身不能发生剪接反应,只有两个片段相互识别,重新构建活性中心才能发生反式剪接。蛋白质剪接可以应用涉及到蛋白质化学修饰、蛋白片段标记同位素、蛋白质环化、蛋白芯片、基因治疗等领域。
3.1 蛋白质剪接在蛋白质化学修饰中的应用
在蛋白质工程和蛋白质研究中,蛋白质的化学修饰技术是非常重要的。对蛋白质进行化学修饰的分子可以是荧光基团、多聚物、非天然氨基酸、酶的辅基、蛋白质翻译后修饰的基团等。化学修饰过的蛋白质会拥有一些新的性质:用生物物理探针修饰蛋白质,可以研究蛋白质的结构和功能;用多聚物修饰蛋白质药物,可以增加蛋白药物的稳定性,提高蛋白药物的利用度等。
氨基修饰或巯基修饰是常见的化学修饰方法,但是标准的化学标记方法做不到特异位点标记。氨基(N端氨基和Lysε位氨基)在蛋白质中的频率较高,如果一个蛋白质中的多个氨基被标记,就会出现蛋白沉淀和荧光猝灭。虽然在蛋白质中低频率出现的Cys使饱和修饰成为可能,但是如果Cys在一种蛋白中出现多次或者Cys位于蛋白的活性中心,就不能做到位点特异性标记。
有些新的化学方法可以进行特异位点标记,但是这些方法仅限于N端氨基的标记,而且还要求蛋白的N端是暴露在外面的,也就是说能否被标记还要依赖N端氨基酸残基的特点[27-29]。有些方法已经可以标记重组蛋白,但是这些方法存在不足之处,其中有一种方法是用一种配体与多肽标签共价相连,通过配体与重组蛋白的非共价结合达到标记的目的,这种方法很简便,但是非共价键标记不稳定,而且多肽标签对目的蛋白的结构和功能有影响[30-31]。蛋白质反式剪接为蛋白质特异位点修饰提供了另一种方法。选择特定的位点,将目的蛋白断裂成两段,分别与IN和IC融合表达。目的蛋白断裂位点的选择根据蛋白的不同而不同,其中一段目的蛋白只含有一个Cys或Lys。把要标记的化学分子标记到Cys或Lys上,再通过蛋白质反式剪接将目的蛋白的两个片段连接在一起[32-34](图6)。由于S1型断裂蛋白质内含子的N端仅含有11个氨基酸,S11型断裂蛋白质内含子的C端仅含有6个氨基酸,易于化学合成,使重组蛋白质的N-末端或C-末端标记更加方便。可以将要标记的分子和目的蛋白的片段直接通过化学合成的方法合成到IN或IC上,通过反式剪接将合成的片段和重组表达的片段拼接到一起。
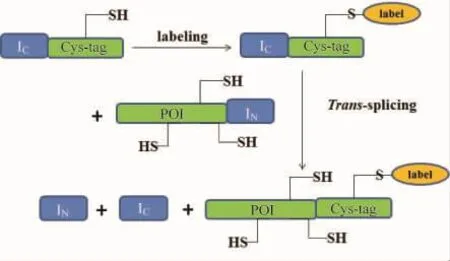
图6 利用蛋白质反式剪接对蛋白质进行特异位点修饰[35]。第一步,将目的蛋白(protein of interest,POI)断裂,分别与IN和IC融合表达,与IC融合表达的片段只含有一个Cys。第二步,将标记物选择性的标记到半胱氨酸上。第三步,两段蛋白发生反式剪接,形成带有标记物的蛋白。
3.2 蛋白质剪接在NMR中的应用
NMR(nuclear magnetic resonance,核磁共振)技术是继x射线晶体衍射技术后研究蛋白3D结构的又一重大进步,这项技术在生命科学、生物医学和材料学中将是至关重要的。NMR技术除了可以研究蛋白的3D结构以外,还可以对蛋白的构象变化以及蛋白间的相互作用进行研究。但是,NMR技术受限于被检测蛋白的分子量,原因是分子量增大会造成谱峰重叠,横向弛豫时间减少(线宽增加)。传统的NMR技术很难用于研究溶液和活细胞中30kDa以上的蛋白质。
对蛋白质进行片段化同位素标记使NMR技术得到进一步的改善,可以检测更大的蛋白质分子。蛋白质片段化标记的方法有天然化学连接(NCL)、表达蛋白连接(EPL)和反式剪接(PTS)等几种方法。天然化学连接(NCL)和表达蛋白连接(EPL)反应中包含两个蛋白,这两个蛋白分别含有a-thioester和a-Cyseine,a-thioester与a-Cyseine发生反应将两个蛋白片段连接在一起[36]。
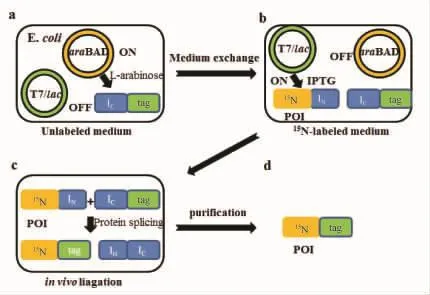
图7 利用蛋白质反式剪接对蛋白质进行片段化标记同位素[37]。a.含有Tag与IC的质粒首先在普通培养基中被诱导表达。b.更换含有同位素的培养基后,诱导表达目的蛋白(protein of interest,POI)与IN。c.在体内发生反式剪接。d.经过纯化,得到有片段被标记的蛋白。
体内(in vivo)反式剪接简化了标记步骤,成功的对多结构域蛋白进行片段同位素标记[37-38](图7)。在这种方法中,两个前体蛋白在同一细胞内两个不同的质粒中表达,两个质粒的诱导方式不一样。第一个质粒在含有同位素的培养基中进行诱导,然后把培养基换成不含同位素的培养基,紧接着诱导另一的前体蛋白。细胞内出现两种前体之后,会自发剪接反应。经过破碎细胞和纯化,得到片段标记的蛋白质。Hideo Iwai研究组利用这种方法分别检测了Src homology结构域和酵母中Sup35p蛋白的3D 结构[37-38]。
3.3 蛋白质剪接在蛋白质环化中的应用
蛋白质环化是指将线性蛋白质的N端和C端连接形成环形蛋白质骨架,是设计新酶和新药以及研究蛋白质结构与功能的新方法。蛋白质环化之后限制了蛋白质的构象变化,降低了熵值,环化后的蛋白质有更高的生物活性和更长的药物半衰期[39]。
通常利用化学方法环化蛋白质,这些方法包括:在N端和C端氨基酸残基间形成二硫键、肽键或是通过连接内部氨基酸的侧链。但是,这些化学方法一般只用于较小的蛋白质,不易应用于较大的蛋白质环化[40]。
Paul Liu和Ming-Qun Xu实验室利用蛋白质反式剪接技术分别对麦芽糖结合蛋白(42 kDa),酰基载体蛋白(9 kDa)和绿色荧光蛋白(27 kDa)等蛋白进行了成功的环化(图8)[41-43]。在这种方法中,IN和IC分别与目的蛋白质的C末端和N末端融合表达,经过蛋白质反式剪接之后,目的蛋白的N末端与C末端以肽键相连,形成环形蛋白质。

图8 利用蛋白质反式剪接技术环化蛋白质[41]。IN和IC分别与目的蛋白(protein of interest,POI)的C末端和N末端融合表达。经过蛋白质反式剪接之后,目的蛋白的N末端与C末端以肽键相连,形成环形蛋白质。
3.4 蛋白质剪接在基因治疗中的应用
在基因治疗研究中,由于有些基因治疗载体对外源基因的大小有限制,导致分子量大的外源基因不能应用到基因治疗中。Paul liu实验室利用蛋白质反式剪接成功的解决了这个问题,为基因治疗中蛋白表达提供了新的方法(图9)[44]。在实验中,选择分子量比较大的肌营养不良蛋白基因(dystrophin gene)和腺病毒载体(AAV)。6.3 kb的肌营养不良蛋白基因(dystrophin gene)不能被腺病毒载体(AAV)带入宿主细胞。肌营养不良蛋白基因cDNA被断裂成两段,并分别与断裂内含子的IN和IC的基因融合表达,将两段基因克隆到两个载体中,将两个载体共转到宿主细胞中,蛋白在宿主细胞中表达,通过蛋白质反式剪接形成成熟的蛋白,行使基因治疗的功能。

图9 蛋白质反式剪接在基因治疗中的应用[44]
3.5 蛋白质剪接在其他方面的应用
蛋白质反式剪接还可以应用到蛋白质研究的其他领域。利用反式剪接获得对宿主细胞有毒性的蛋白的全长序列,例如I-Tev I核酸内切酶[45]和b-amyloid(Ab)[46];可以利用蛋白质反式剪接重构球蛋白的活性,可以基于这个方法检测蛋白质-蛋白质相互作用[47];利用反式剪接重新构建herbicide-resistance蛋白活性,确保转基因植物的环境安全性[48];利用蛋白质反式剪接把目的蛋白固定到芯片上,应用于蛋白质组学研究[49]。
4 小结与展望
蛋白质剪接技术为蛋白质研究和蛋白质工程提供了新工具,在蛋白质特异位点标记、蛋白质片段化同位素标记、蛋白质环化和基因治疗等方面取得了一些成果。另外,蛋白质剪接技术可以应用在毒性蛋白的表达、蛋白质相互作用、蛋白固定等方面。新型断裂蛋白质内含子在此基础上扩大了断裂蛋白质内含子的应用范围,提供了在目的蛋白质的N-末端或C-末端标记任意化学基团的潜力。随着蛋白质剪接技术的日趋完善,蛋白质剪接还会应用在蛋白质研究的其他领域。
然而,蛋白质剪接技术还存在一些限制。例如,蛋白质内含子剪接活性受外显子的影响,所以会在目的蛋白的断裂位点插入一些外源氨基酸,这些氨基酸可能会对目的蛋白的结构与功能产生影响。所以在设计目的蛋白的断裂位点时要充分考虑到这一点。另外,有些人工构建的蛋白质内含子与有些蛋白融合表达时,会得到很少的可溶性蛋白,要经过变复性才能使蛋白质内含子有活性。尽管有这些限制,蛋白质剪接技术还是应用到了蛋白质研究与蛋白质工程的很多领域。
(2011年9月30日收到)
[1]LIU X Q.Protein-splicing intein:genetic mobility,origin,and evolution[J].Annu Rev Genet,2000,34:61-76.
[2]SALEH L,PERLER F B.Protein splicing in cis and in trans[J].Chem Rec,2006,6(4):183-193.
[3]PERLER F B.InBase:the Intein Database[J].Nucleic Acids Res,2002,30(1):383-384.
[4]HALL T M,PORTER J A,YOUNG K E,et al.Crystal structure of a Hedgehog autoprocessing domain:homology between Hedgehog and self-splicing proteins[J].Cell,1997,91(1):85-97.
[5]MOOTZ H D.Split inteins as versatile tools for protein semisynthesis[J].Chembiochem,2009,10(16):2579-2589.
[6]CHERIYAN M,PERLER F B.Protein splicing:a versatile tool for drug discovery[J].Adv Drug Deliv Rev,2009,61(11):899-907.
[7]VILA-PERELLO M,MUIR T W.Biological applications of protein splicing[J].Cell,2010,143(2):191-200.
[8]KANE P M,YAMASHIRO C T,WOLCZYK D F,et al.Protein splicing converts the yeast TFP1 gene product to the 69-kD subunit of the vacuolar H(+)-adenosine triphosphatase[J].Science,1990,250(4981):651-657.
[9]HIRATA R,OHSUMI Y,NAKANO A,et al.Molecular structure of a gene,VMA1,encoding the catalytic subunit of H(+)-translocating adenosine triphosphatase from vacuolar membranes of Saccharomyces cerevisiae[J].J Biol Chem,1990,265(12):6726-6733.
[10]PERLER F B,DAVIS E O,DEAN G E,et al.Protein splicing elements:inteins and exteins—a definition of terms and recommended nomenclature[J].Nucleic Acids Res,1994,22(7):1125-1127.
[11]SUN P,YE S,FERRANDON S,et al.Crystal structures of an intein from the split dnaE gene of Synechocystis sp.PCC6803 reveal the catalytic model without the penultimate histidine and the mechanism of zinc ion inhibition of protein splicing[J].J Mol Biol,2005,353(5):1093-1105.
[12]DING Y,XU M Q,GHOSH I,et al.Crystal structure of a mini-intein reveals a conserved catalytic module involved in side chain cyclization of asparagine during protein splicing[J].J Biol Chem,2003,278(40):39133-39142.
[13]XU M Q,PERLER F B.The mechanism of protein splicing and its modulation by mutation[J].EMBO J,1996,15(19):5146-5153.
[14]PAULUS H.Protein splicing and related forms of protein autoprocessing[J].Annu Rev Biochem,2000,69:447-496.
[15]WU H,HU Z,LIU X Q.Protein trans-splicing by a split intein encoded in a split DnaE gene of Synechocystis sp.PCC6803[J].Proc Natl Acad Sci U S A,1998,95(16):9226-9231.
[16]MOOTZ H D,MUIR T W.Protein splicing triggered by a small molecule[J].J Am Chem Soc,2002,124(31):9044-9045.
[17]WU H,XU M Q,LIU X Q.Protein trans-splicing and functional mini-inteins of a cyanobacterial dnaB intein [J].Biochim Biophys Acta,1998,1387(1-2):422-432.
[18]MATHYS S,EVAN JR T C,CHUTE I C,et al.Characterization of a self-splicing mini-intein and its conversion into autocatalytic N-and C-terminal cleavage elements:facile production of protein building blocks for protein ligation [J].Gene,1999,231(1-2):1-13.
[19]LIU X Q,YANG J.Split dnaE genes encoding multiple novel inteins in Trichodesmium erythraeum [J].J Biol Chem,2003,278(29):26315-26318.
[20]CASPI J,AMITAI G,BELENKIY O,et al.Distribution of split DnaE inteins in cyanobacteria[J].Mol Microbiol,2003,50(5):1569-1577.
[21]SHINGLEDECKER K,JIANG S Q,PAULUS H.Molecular dissection of the Mycobacterium tuberculosis RecA intein:design of a minimal intein and of a trans-splicing system involving two intein fragments[J].Gene,1998,207(2):187-195.
[22]ICHIYANAGI K,ISHINO Y,ARIYOSHI M,et al.Crystal structure of an archaeal intein-encoded homing endonuclease PI-PfuI[J].J Mol Biol,2000,300(4):889-901.
[23]SOUTHWORTH M W,ADAM E,PANNE D,et al.Control of protein splicing by intein fragment reassembly[J].EMBO J,1998,17(4):918-926.
[24]MILLS K V,LEW B M,JIANG S Q,et al.Protein splicing in trans by purified N-and C-terminal fragments of the Mycobacterium tuberculosis RecA intein[J].Proc Natl Acad Sci U S A,1998,95(7):3543-3548.
[25]SUN W,YANG J,LIU X Q.Synthetic two-piece and threepiece split inteins for protein trans-splicing[J].J Biol Chem,2004,279(34):35281-35286.
[26]APPLEBY J H,ZHOU K,VOLKMANN G,et al.Novel split intein for trans-splicing synthetic peptide onto C terminus of protein[J].J Biol Chem,2009,284(10):6194-6199.
[27]TSIEN R Y.Constructing and exploiting the fluorescent protein paintbox(Nobel Lecture)[J].Angew Chem Int Ed Engl,2009,48(31):5612-5626.
[28]MCCANN C M,BAREYRE F M,LICHTMAN J W,et al.Peptide tags for labeling membrane proteins in live cells with multiple fluorophores[J].Biotechniques,2005,38(6):945-952.
[29]GUIGNET E G,HOVIUS R,VOGEL H.Reversible site-selective labeling of membrane proteins in live cells[J].Nat Biotechnol,2004,22(4):440-444.
[30]KEPPLER A,GENDREIZIG S,GRONEMEYER T,et al.A general method for the covalent labeling of fusion proteins with small molecules in vivo[J].Nat Biotechnol,2003,21(1):86-89.
[31]CHEN I,HOWARTH M,LIN W,et al.Site-specific labeling of cell surface proteins with biophysical probes using biotin ligase[J].Nat Methods,2005,2(2):99-104.
[32]KURPIERS T,MOOTZ H D.Regioselective cysteine bioconjugation by appending a labeled cystein tag to a protein by using protein splicing in trans[J].Angew Chem Int Ed Engl,2007,46(27):5234-5237.
[33]LUDWIG C,PFEIFF M,LINNE U,et al.Ligation of a synthetic peptide to the N terminus of a recombinant protein using semisynthetic protein trans-splicing [J].Angew Chem Int Ed Engl,2006,45(31):5218-5221.
[34]VOLKMANN G,LIU X Q.Protein C-terminal labeling and biotinylation using synthetic peptide and split-intein [J].PLoS One,2009,4(12):e8381.
[35]KURPIERS T,MOOTZ H D.Site-specific chemical modification of proteins with a prelabelled cysteine tag using the artificially split Mxe GyrA intein[J].Chembiochem,2008,9(14):2317-2325.
[36]XU R,AYERS B,COWBURN D,et al.Chemical ligation of folded recombinant proteins:segmental isotopic labeling of domains for NMR studies[J].Proc Natl Acad Sci U S A,1999,96(2):388-393.
[37]ZUGER S,IWAI H.Intein-based biosynthetic incorporation of unlabeled protein tags into isotopically labeled proteins for NMR studies[J].Nat Biotechnol,2005,23(6):736-740.
[38]MUONA M,ARANKO A S,IWAI H.Segmental isotopic labelling of a multidomain protein by protein ligation by protein trans-splicing[J].Chembiochem,2008,9(18):2958-2961.
[39]KEE K S,JOIS S D.Design of beta-turn based therapeutic agents[J].Curr Pharm Des,2003,9(15):1209-1224.
[40]XU M Q,EVANS T J.Intein-mediated ligation and cyclization of expressed proteins[J].Methods,2001,24(3):257-277.
[41]EVANS T C Jr,MARTIN D,KOLLY R,et al.Protein trans-splicing and cyclization by a naturally split intein from the dnaE gene of Synechocystis species PCC6803[J].J Biol Chem,2000,275(13):9091-9094.
[42]VOLKMANN G,MURPHY P W,ROWLAND E E,et al.Intein-mediated cyclization of bacterial acyl carrier protein stabilizes its folded conformation but does not abolish function[J].J Biol Chem,2010,285(12):8605-8614.
[43]ZHAO Z,MA X,LI L,et al.Protein cyclization enhanced thermostability and exopeptidase-resistance of green fluorescent protein[J].J Microbiol Biotechnol,2010,20(3):460-466.
[44]LI J,SUN W,WANG B,et al.Protein trans-splicing as a means for viral vector-mediated in vivo gene therapy [J].Hum Gene Ther,2008,19(9):958-964.
[45]WU W,WOOD D W,BELFORT G,et al.Intein-mediated purification of cytotoxic endonuclease I-TevI by insertional inactivation and pH-controllable splicing [J].Nucleic Acids Res,2002,30(22):4864-4871.
[46]BOCKHORN J J,LAZAR K L,GASSER A J,et al.Novel semisynthetic method for generating full length beta-amyloid peptides[J].Biopolymers,2010,94(4):511-520.
[47]OZAWA T,UMEZAWA Y.Detection of protein-protein interactions in vivo based on protein splicing [J].Curr Opin Chem Biol,2001,5(5):578-583.
[48]EVANS T J,XU M Q,PRADHAN S.Protein splicing elements and plants:from transgene containment to protein purification[J].Annu Rev Plant Biol,2005,56:375-392.
[49]KWON Y,COLEMAN M A,CAMARERO J A.Selective immobilization of proteins onto solid supports through splitintein-mediated protein trans-splicing [J].Angew Chem Int Ed Engl,2006.45(11):1726-1729.
