第二代HIV-1整合酶抑制剂筛选靶点概述
2012-01-24张大为赵明明宋超张岗郭顺星
张大为,赵明明,宋超,张岗,郭顺星
HIV-1 整合酶是病毒复制所必需的酶[1]。在临床上,使用 HIV-1 整合酶(integrase,IN)抑制剂来治疗 HIV 感染被证明是非常有益的。目前,FDA 批准上市的第一个 HIV-1整合酶抑制剂 raltegravir 已作为一线药物用于临床[2-3]。与raltegravir 作用机制相同的另外一个药物 elvitegravir 目前正处于 III 期临床试验阶段[4]。Raltegravir 和 elvitegravir作为第一代整合酶抑制剂,其作用机制是抑制 HIV-1 整合酶链转移反应[5-6],因此,它们被称为链转移反应抑制剂(strand transfer inhibitors,STIs)。随着 raltegravir 的广泛使用,病毒耐药也随之出现[7]。为了规避病毒耐药和开发出能与 STIs 协同作用的整合酶抑制剂,我们需要筛选出靶向整合过程中的不同步骤或不同位点的化合物。这类化合物的作用机制与 STIs 不同,所以称作第二代整合酶抑制剂。通过查阅文献,本文综述了第二代整合酶抑制剂的筛选靶点,主要包括 IN 的酶催化功能和非酶催化功能。
1 整合酶的结构与功能
1.1 整合酶的结构
整合酶由 HIV-1 pol 基因 3' 末端编码,具有 288 个氨基酸残基(32 kD),有 3 个结构域,即一个 N 端结构域(NTD),一个催化核心区(CCD)和一个 C 端结构域(CTD)[8]。NTD 由 1 ~ 51 位氨基酸构成,包含一个高度保守的 HHCC(His-His-Cys-Cys)模序,可与 Zn2+形成HTH(helix-turn-helix)结构,体外实验表明 Zn2+对于整合酶的正确折叠、多聚化以及催化功能的行使具有关键作用;CCD 由 212 ~ 288 位氨基酸组成,包含一个保守且直接参与催化的 DDE(D64、D116、和 E152)模序;CTD 由212 ~ 288 位氨基酸残基组成,该结构域具有非特异性结合DNA 的能力[9]。由于 IN 可溶性差,科学家们至今尚未得到 HIV 整合酶完整的三维结构。2010 年,Hare 等[10]的一项研究得到了原型泡沫病毒(prototype foamy virus,PFV)的整合酶三维结构。作为逆转录病的 PFV,其整合酶与HIV 病毒整合酶结构十分类似,因此 PFV 整合酶与 DNA以及抑制剂的复合物晶体结构的获得为 HIV-1 整合酶三维结构的获得和 HIV-1 整合酶抑制剂的研究带来了可靠的参照。
1.2 整合酶的功能
整合酶的功能包含两个方面(图 1),即学界广泛接受的催化功能和正在不断被阐明的非催化功能。
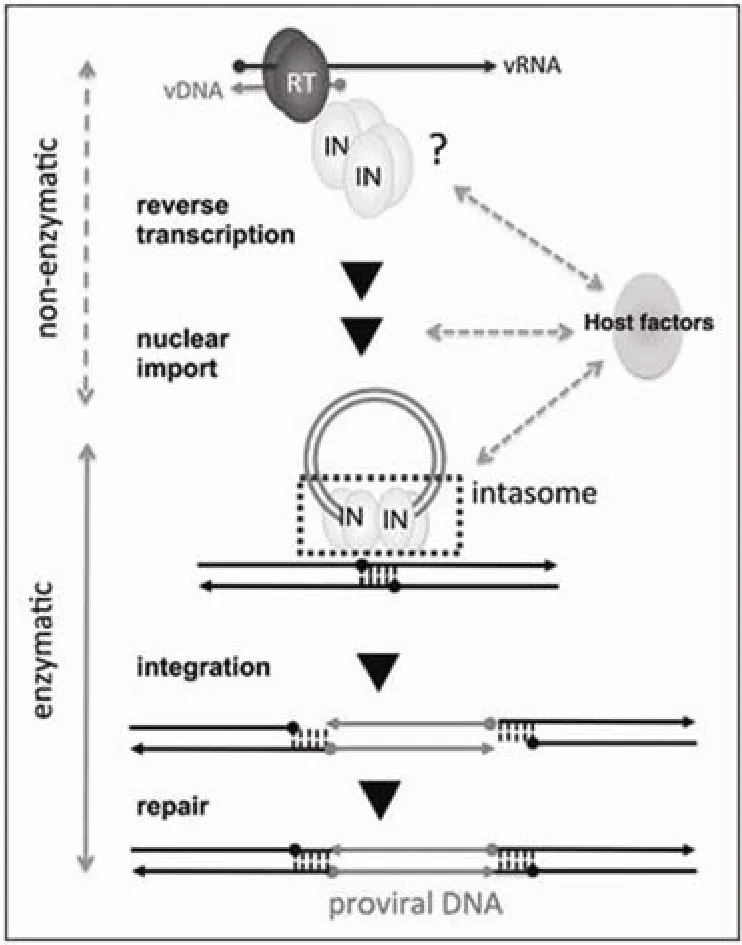
图 1 HIV-1 IN 的催化功能和非催化功能[8]
1.2.1 整合酶催化功能[11]整合酶催化的反应为整合反应。在整合过程中,整合酶完成两个相关且依次发生的酶促反应。首先是 3' 加工,整合酶与胞质中的病毒双链 DNA结合后,切除其 3' 末端的两个碱基,露出高度保守的 CA末端,用于结合细胞 DNA;第二步为链转移反应,3' 加工形成的核酸蛋白复合物入核,随后整合酶在宿主 DNA 上切出间隔 5 个碱基的交错切口,接着病毒 DNA 的 3' 端与宿主 DNA 的 5' 端共价连接起来。整合过程随着宿主细胞蛋白将整合过程中产生的缺口修复而结束。
1.2.2 整合酶非催化功能 遗传学分析发现,整合酶除参与整合外,还可能参与到 HIV-1 生活周期中的其他非酶促过程,包括病毒脱壳[12]、逆转录[13]、病毒 DNA 的核进入[14]以及病毒粒子装配和成熟过程中蛋白质的加工[15]。不难发现,整合酶的非催化功能是通过与宿主蛋白或者病毒蛋白的相互作用而实现的。
2 第二代整合酶抑制剂筛选靶点
整合酶抑制剂靶点的选择是基于整合酶的功能。目前,第一代整合酶抑制剂 STIs 是以整合酶催化功能第二步链转移反应为靶点的链转移反应抑制剂,第二代整合酶抑制剂筛选的靶点主要有整合酶 3' 加工和整合酶非催化功能,现将其综述如下。
2.1 整合酶 3' 加工反应
在病毒 RNA 完成逆转录后,整合酶二聚体分别与产物DNA 的两个末端结合,完成整合过程的第一步—— 3' 加工。3' 加工会触发整合酶与 DNA 作用形式的重构:整合酶四聚体与 3' 加工后的 DNA 结合形成整合体(intasome),接着该整合体复合物进入宿主胞核催化链转移反应[10,16-17]。研究发现,整合酶链转移反应抑制剂 raltegravir 和 elvitegravir特异性作用于链转移反应而不作用于 3' 加工反应[18]。最近,Hare 等[10]研究发现,raltegravir 或 elvitegravir 会与整合体中的 DNA 和 Mg2+形成复合体,抑制剂选择性结合整合体中的 DNA 会导致整合体下一步催化活性的丧失。因此,3' 加工的抑制剂作为一类新的整合酶抑制剂,能够与现有的链转移抑制协同作用,用于 HIV-1 的治疗。
迄今为止,尽管已经发现了能够抑制 3' 加工过程的抑制剂,但是尚未找到能够特异性抑制该过程的抑制剂。苯乙烯基喹啉类化合物(styrylquinoline,SQLs)在体外能够竞争性抑制病毒 DNA 同 IN 的结合,从而抑制 3' 加工过程。同时 SQLs 对链转移过程也有一定程度的抑制活性[19]。1-(5-氯吲哚-3-基)-3-羟基-3-(2-氢四唑-5-基)丙烯酮(5ClTEP)类衍生物能够同时抑制整合酶的 3' 加工和链转移活性,这类二酮酸抑制剂通过结合病毒 DNA 而抑制整合酶的 3' 加工活性[20]。
2.2 整合酶多聚化
在体内,蛋白质往往与一些能够改变其构象和功能的平衡相关,能够改变这种平衡的分子具有开发成为药物的潜力。整合酶单体、二聚体和四聚体之间存在一个动态平衡[21-23]。在胞浆中催化 3' 加工反应时,整合酶二聚体分别与病毒 DNA 的两端结合[24],此后结合有病毒 LTR-DNA的整合酶二聚体相互靠近形成四聚体,行使链转移功能[25]。Hayouka 等[26]发现,他们设计的一个 LEDGF 衍生肽能够通过阻断 IN 而抑制 HIV-1 的复制。该肽通过调节整合酶多聚化的平衡过程,使得整合酶二聚体转变为四聚体,而整合酶四聚体不能行使 3' 加工功能,导致链转移反应没有底物,最终整合酶因为 3' 加工的功能受到抑制而无法完成其功能(图 2)。此外,有研究发现,整合酶四聚体也不能直接与完成 3' 加工的 DNA 结合[21],这样的话,即使存在 3'加工产物,整合酶四聚体也不行使链转移功能。因此,Hayouka 等认为这样的抑制剂具有目前临床使用的整合酶抑制剂所没有的优点,即能够同时抑制整合酶的两步催化反应。
迄今为止,人们只发现了一个能够抑制整合酶多聚化的小分子 chiba-3003[27],其他已知具有该活性的抑制剂都是源于 IN 自身的衍生肽或者是源于 LEDGF 的衍生肽[26,28-29],而肽类药物与小分子药物相比,其吸收性较差,因此,开发整合酶多聚化小分子抑制剂势在必行。值得注意的是,虽然前期工作已取得了进展[30-32],但蛋白质相互作用小分子抑制剂的寻找依然充满挑战,因此我们的路还很长。
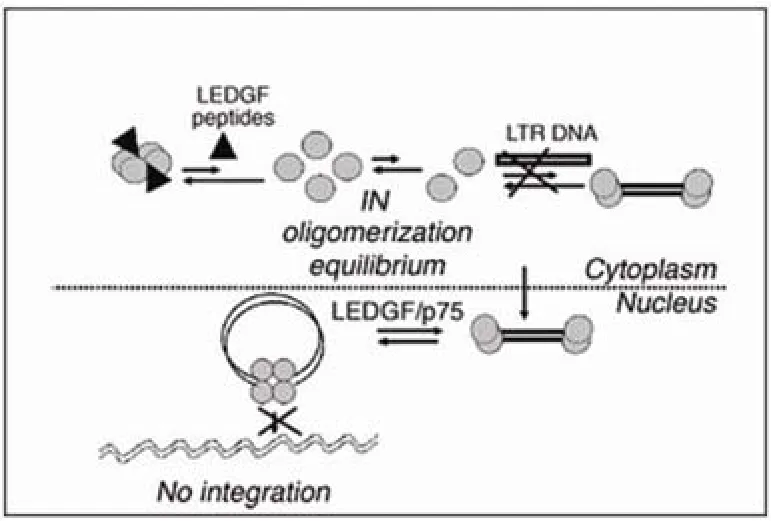
图 2 整合酶多聚化抑制剂作用模型[26]
2.3 整合酶与宿主辅因子相互作用
研究发现,整合酶和宿主辅因子的相互作用与 HIV-1复制的多个过程有关,比如逆转录、前整合复合体的形成、核定位、整合以及病毒装配[33]。这些相互作用一旦被证明对 HIV 感染宿主是必需的,就能作为药物开发的靶点。已报道的与 IN 具有蛋白-蛋白相互作用(protein-protein interation,PPI)的宿主蛋白有:INI1[34]、Rad18[35-36]、LEDGF[37]、EED[38]、importin 7[39]、Gemin2[40]、UNG2[41-42]、N-recognins[43]、APOBEC3F/G[44]、von Hippel Lindau binding protein 1(VBP1)[45]、p300 Transportin[46]、SR2(TNPO3)[47-48]、SAP18-HDAC1[49]和 JNK/PIN1[50]。
目前,这些 PPI 中被研究的最为清楚的是 IN 与LEDGF/p75 之间的相互作用。LEDGF/p75对于 HIV-1 的整合和复制非常关键[51-55],它将整合复合体锚定到宿主染色体上,引导整合体到整合反应发生的位点。研究发现,特异性干扰 IN 与 LEDGF/p75 之间相互作用的分子 CX06387能够抑制 HIV-1 在体外动物水平的复制,该分子未表现出细胞毒性,也没有和链转移酶抑制剂产生交叉耐药[56]。这表明寻找和设计针对整合酶与宿主辅因子相互作用的小分子抑制剂是可行的。但是,最近的一项研究发现,那些 CCD结构域发生突变而不能与 LEDGF/p75 结合的整合酶突变体,仍然可以和宿主染色体结合催化链转移反应,带有上述整合酶突变体的病毒突变株仍然能够维持一个低水平的复制状态,表明 IN 还具有一个非 LEDGF/p75 依赖的染色体结合机制[57],这限制了 IN 与 LEDGF/p75 之间相互作用抑制剂的有效性。
到目前为止,对于上述 PPI 的研究尚处初级阶段。这些 PPI 对于病毒的复制是否必需,以及将执行多种功能的细胞蛋白作为靶点是否会带来细胞毒性,对这两个问题的回答依然没有定论,甚至许多研究结果是相反的。尽管如此,我们应该注意到,将宿主蛋白作为抗病毒靶点,具有对耐药病毒株相对不敏感的优点,因此,整合酶与宿主辅因子之间相互作用的抑制剂具有诱人的应用前景。
2.4 整合酶翻译后修饰
整合酶的翻译后修饰包括:泛素化、乙酰化和磷酸化。这些修饰各自的作用是:调控 IN 胞内稳定[43,45,58]、整合效率[46,59-60]以及调节休眠 CD4 阳性 T 细胞对于病毒感染的敏感性[50]。例如,调节宿主染色体构象的乙酰基转移酶p300 和 GCN5 能够乙酰化 IN,进而增加 IN 对于 DNA的亲和力,提高 IN 链转移活性[46]。遗憾的是参与 IN 翻译后修饰的蛋白也参与宿主体内诸如蛋白质稳定、染色质重构以及胞内信号转导等主要通路,抑制这些蛋白的功能,对宿主细胞具有潜在的危害。因此,找到针对 IN 与 IN 翻译后修饰蛋白相互作用的特异性抑制剂还是值得期待的。
3 结语
整合酶抑制剂的作用机制要么是阻断其催化功能,要么是阻断其非催化功能。以催化功能作为靶点的抑制剂特异性作用于病毒蛋白,不会影响宿主蛋白功能,因此具有很小的宿主细胞毒性,但是,该类抑制剂的缺点十分明显:对于整合酶的突变非常敏感,易产生耐药性,致使其应用的有效性受到限制。而以非催化功能作为靶点的抑制剂阻断的是 IN同宿主蛋白之间的 PPI。与前一类抑制剂相比,该类抑制剂对 IN 的突变不敏感,不容易产生耐药性,但其缺点是因为会影响到宿主蛋白的功能而具有宿主细胞毒性。相比较而言,以 IN 非催化功能作为抗病毒靶点的抑制剂在未来更具开发潜力。在未来,一定会有更多的靶向不同步骤或不同位点的新结构整合酶抑制剂问世,通过协同作用增强活性、降低毒性、减缓病毒抗药性的产生,从而达到更好地控制艾滋病的效果。
[1] Engelman A, Mizuuchi K, Craigie R. HIV-1 DNA integration:mechanism of viral DNA cleavage and DNA strand transfer. Cell,1991, 67(6):1211-1221.
[2] Zolopa AR. The evolution of HIV treatment guidelines: current state-of-the-art of ART. Antiviral Res, 2010, 85(1):241-244.
[3] Thompson MA, Aberg JA, Cahn P, et al. Antiretroviral treatment of adult HIV infection: 2010 recommendations of the International AIDS Society-USA panel. JAMA, 2010, 304(3):321-333.
[4] Shimura K, Kodama EN. Elvitegravir: a new HIV integrase inhibitor.Antivir Chem Chemother, 2009, 20(2):79-85.
[5] Summa V, Petrocchi A, Bonelli F, et al. Discovery of raltegravir, a potent, selective orally bioavailable HIV-integrase inhibitor for the treatment of HIV-AIDS infection. J Med Chem, 2008, 51(18):5843-5855.
[6] Sato M, Motomura T, Aramaki H, et al. Novel HIV-1 integrase inhibitors derived from quinolone antibiotics. J Med Chem, 2006,49(5):1506-1508.
[7] Métifiot M, Marchand C, Maddali K, et al. Resistance to integrase inhibitors. Viruses, 2010, 2(7):1347-1366.
[8] Masuda T. Non-enzymatic functions of retroviral integrase: the next target for novel anti-HIV drug development. Front Microbiol, 2011,2:210.
[9] Pommier Y, Johnson AA, Marchand C. Integrase inhibitors to treat HIV/AIDS. Nat Rev Drug Discov, 2005, 4(3):236-248.
[10] Hare S, Gupta SS, Valkov E, et al. Retroviral intasome assembly and inhibition of DNA strand transfer. Nature, 2010, 464(7286):232-236.
[11] Delelis O, Carayon K, Saïb A, et al. Integrase and integration:biochemical activities of HIV-1 integrase. Retrovirology, 2008, 5:114.
[12] Briones MS, Dobard CW, Chow SA. Role of human immunodeficiency virus type 1 integrase in uncoating of the viral core. J Virol, 2010,84(10):5181-5190.
[13] Nishitsuji H, Hayashi T, Takahashi T, et al. Augmentation of reverse transcription by integrase through an interaction with host factor,SIP1/Gemin2 Is critical for HIV-1 infection. PLoS One, 2009, 4(11):e7825.
[14] Ikeda T, Nishitsuji H, Zhou X, et al. Evaluation of the functional involvement of human immunodeficiency virus type 1 integrase in nuclear import of viral cDNA during acute infection. J Virol, 2004,78(21):11563-11573.
[15] Mohammed KD, Topper MB, Muesing MA. Sequential deletion of the integrase (Gag-Pol) carboxyl terminus reveals distinct phenotypic classes of defective HIV-1. J Virol, 2011, 85(10):4654-4666.
[16] Craigie R. Structural biology: When four become one. Nature, 2010,464(7286):167-168.
[17] Cherepanov P. Integrase illuminated. EMBO Rep, 2010, 11(5):328.
[18] Espeseth AS, Felock P, Wolfe A, et al. HIV-1 integrase inhibitors that compete with the target DNA substrate define a unique strand transfer conformation for integrase. Proc Natl Acad Sci U S A 2000, 97(21):11244-11249.
[19] Deprez E, Barbe S, Kolaski M, et al. Mechanism of HIV-1 integrase inhibition by styrylquinoline derivatives in vitro. Mol Pharmacol,2004, 65(1):85-98.
[20] Marchand C, Zhang X, Pais GC, et al. Structural determinants for HIV-1 integrase inhibition by beta-diketo acids. J Biol Chem, 2002,277(15):12596-12603.
[21] Faure A, Calmels C, Desjobert C, et al. HIV-1 integrase crosslinked oligomers are active in vitro. Nucleic Acids Res, 2005, 33(3):977-986.
[22] Deprez E, Tauc P, Leh H, et al. DNA binding induces dissociation of the multimeric form of HIV-1 integrase: a time-resolved fluorescence anisotropy study. Proc Natl Acad Sci U S A, 2001, 98(18):10090-10095.
[23] Deprez E, Tauc P, Leh H, et al. Oligomeric states of the HIV-1 integrase as measured by time-resolved fluorescence anisotropy.Biochemistry, 2000, 39(31):9275-9284.
[24] Guiot E, Carayon K, Delelis O, et al. Relationship between the oligomeric status of HIV-1 integrase on DNA and enzymatic activity.J Biol Chem, 2006, 281(32):22707-22719.
[25] Chen A, Weber IT, Harrison RW, et al. Identification of amino acids in HIV-1 and avian sarcoma virus integrase subsites required for specific recognition of the long terminal repeat Ends. J Biol Chem, 2006,281(7):4173-4182.
[26] Hayouka Z, Rosenbluh J, Levin A, et al. Inhibiting HIV-1 integrase by shifting its oligomerization equilibrium. Proc Natl Acad Sci U S A,2007, 104(20):8316-8321.
[27] De Luca L, Barreca ML, Ferro S, et al. Pharmacophore-based discovery of small-molecule inhibitors of protein-protein interactions between HIV-1 integrase and cellular cofactor LEDGF/p75.ChemMedChem, 2009, 4(8):1311-1316.
[28] Hayouka Z, Levin A, Maes M, et al. Mechanism of action of the HIV-1 integrase inhibitory peptide LEDGF 361-370. Biochem Biophys Res Commun, 2010, 394(2):260-265.
[29] Maes M, Levin A, Hayouka Z, et al. Peptide inhibitors of HIV-1 integrase: from mechanistic studies to improved lead compounds.Bioorg Med Chem, 2009, 17(22):7635-7642.
[30] He MM, Smith AS, Oslob JD, et al. Small-molecule inhibition of TNF-alpha. Science, 2005, 310(5750):1022-1025.
[31] Zutshi R, Chmielewski J. Targeting the dimerization interface for irreversible inhibition of HIV-1 protease. Bioorg Med Chem Lett,2000, 10(17):1901-1903.
[32] McMillan K, Adler M, Auld DS, et al. Allosteric inhibitors of inducible nitric oxide synthase dimerization discovered via combinatorial chemistry. Proc Natl Acad Sci U S A, 2000, 97(4):1506-1511.
[33] Van Maele B, Busschots K, Vandekerckhove L, et al. Cellular co-factors of HIV-1 integration. Trends Biochem Sci, 2006, 31(2):98-105.
[34] Kalpana GV, Marmon S, Wang W, et al. Binding and stimulation of HIV-1 integrase by a human homolog of yeast transcription factor SNF5. Science, 1994, 266(5193):2002-2006.
[35] Mulder LC, Chakrabarti LA, Muesing MA. Interaction of HIV-1 integrase with DNA repair protein hRad18. J Biol Chem, 2002,277(30):27489-27493.
[36] Lloyd AG, Tateishi S, Bieniasz PD, et al. Effect of DNA repair protein Rad18 on viral infection. PLoS Pathog, 2006, 2(5):e40.
[37] Cherepanov P, Maertens G, Proost P, et al. HIV-1 integrase forms stable tetramers and associates with LEDGF/p75 protein in human cells. J Biol Chem, 2003, 278(1):372-381.
[38] Violot S, Hong SS, Rakotobe D, et al. The human polycomb group EED protein interacts with the integrase of human immunodeficiency virus type 1. J Virol, 2003, 77(23):12507-12522.
[39] Ao Z, Huang G, Yao H, et al. Interaction of human immunodeficiency virus type 1 integrase with cellular nuclear import receptor importin 7 and its impact on viral replication. J Biol Chem, 2007, 282(18):13456-13467.
[40] Hamamoto S, Nishitsuji H, Amagasa T, et al. Identification of a novel human immunodeficiency virus type 1 integrase interactor, Gemin2,that facilitates efficient viral cDNA synthesis in vivo. J Virol, 2006,80(12):5670-5671.
[41] Priet S, Navarro JM, Gros N, et al. Functional role of HIV-1 virion-associated uracil DNA glycosylase 2 in the correction of G:U mispairs to G:C pairs. J Biol Chem, 2003, 278(7):4566-4571.
[42] Priet S, Gros N, Navarro JM, et al. HIV-1-associated uracil DNA glycosylase activity controls dUTP misincorporation in viral DNA and is essential to the HIV-1 life cycle. Mol cell, 2005, 17(4):479-490.
[43] Tasaki T, Mulder LC, Iwamatsu A, et al. A family of mammalian E3 ubiquitin ligases that contain the UBR box motif and recognize N-degrons. Mol Cell Biol, 2005, 25(16):7120-7136.
[44] Luo K, Wang T, Liu B, et al. Cytidine deaminases APOBEC3G and APOBEC3F interact with human immunodeficiency virus type 1 integrase and inhibit proviral DNA formation. J Virol, 2007, 81(13):7238-7248.
[45] Mousnier A, Kubat N, Massias-Simon A, et al. von Hippel Lindau binding protein 1-mediated degradation of integrase affects HIV-1 gene expression at a postintegration step. Proc Natl Acad Sci U S A,2007, 104(34):13615-13620.
[46] Cereseto A, Manganaro L, Gutierrez MI, et al. Acetylation of HIV-1 integrase by p300 regulates viral integration. EMBO J, 2005, 24(17):3070-3081.
[47] Luban J. HIV-1 infection: going nuclear with TNPO3/Transportin-SR2 and integrase. Curr Biol, 2008, 18(16):R710-R713.
[48] Rain JC, Cribier A, Gérard A, et al. Yeast two-hybrid detection of integrase-host factor interactions. Methods, 2009, 47(4):291-297.
[49] Sorin M, Cano J, Das S, et al. Recruitment of a SAP18-HDAC1 complex into HIV-1 virions and its requirement for viral replication.PLoS pathogens, 2009, 5(6):e1000463.
[50] Manganaro L, Lusic M, Gutierrez MI, et al. Concerted action of cellular JNK and Pin1 restricts HIV-1 genome integration to activated CD4+ T lymphocytes. Nat Med, 2010, 16(3):329-333.
[51] Emiliani S, Mousnier A, Busschots K, et al. Integrase mutants defective for interaction with LEDGF/p75 are impaired in chromosome tethering and HIV-1 replication. J Biol Chem, 2005,280(27):25517-25523.
[52] Maertens G, Cherepanov P, Pluymers W, et al. LEDGF/p75 is essential for nuclear and chromosomal targeting of HIV-1 integrase in human cells. J Biol Chem, 2003, 278(35):33528-33539.
[53] De Rijck J, Vandekerckhove L, Gijsbers R, et al. Overexpression of the lens epithelium-derived growth factor/p75 integrase binding domain inhibits human immunodeficiency virus replication. J Virol,2006, 80(23):11498-11509.
[54] Vandekerckhove L, Christ F, Van Maele B, et al. Transient and stable knockdown of the integrase cofactor LEDGF/p75 reveals its role in the replication cycle of human immunodeficiency virus. J Virol, 2006,80(4):1886-1896.
[55] Llano M, Saenz DT, Meehan A, et al. An essential role for LEDGF/p75 in HIV integration. Science, 2006, 314(5798):461-464.
[56] Christ F, Voet A, Marchand A, et al. Rational design of small-molecule inhibitors of the LEDGF/p75-integrase interaction and HIV replication. Nat Chem Biol, 2010, 6(6):442-448.
[57] Zheng Y, Ao Z, Jayappa KD, et al. Characterization of the HIV-1 integrase chromatin- and LEDGF/p75-binding abilities by mutagenic analysis within the catalytic core domain of integrase. Virol J, 2010,7:68.
[58] Mulder LC, Muesing MA. Degradation of HIV-1 integrase by the N-end rule pathway. J Biol Chem, 2000, 275(38):29749-29753.
[59] Terreni M, Valentini P, Liverani V, et al. GCN5-dependent acetylation of HIV-1 integrase enhances viral integration. Retrovirology, 2009,7:18.
[60] Topper M, Luo Y, Zhadina M, et al. Posttranslational acetylation of the human immunodeficiency virus type 1 integrase carboxyl-terminal domain is dispensable for viral replication. J Virol, 2007, 81(6):3012-3017.
