基于16S rRNA部分序列探讨部分鳚亚目鱼类的分子系统进化关系
2012-01-12张源真姜志强
张源真, 王 伟, 姜志强, 张 钊
(大连海洋大学 生命与水产学院 农业部海水增养殖学重点开放实验室, 辽宁 大连 116023)
基于16S rRNA部分序列探讨部分鳚亚目鱼类的分子系统进化关系
张源真, 王 伟, 姜志强, 张 钊
(大连海洋大学 生命与水产学院 农业部海水增养殖学重点开放实验室, 辽宁 大连 116023)
通过PCR扩增获得了3科5属6种中国黄渤海海域的鳚亚目(Blennioidei)鱼类的线粒体16S rRNA基因序列片段约669bp碱基, 结合来自GenBank的5种鳚亚目其他科鱼类的相应基因片段, 并以眼斑雪冰鱼(Chionodraco rastrospinosus)为外群, 生成供系统发育分析的序列矩阵, 利用MEGA 4.0软件分析序列的碱基组成、差异百分比、转换/颠换值等, 应用最大简约法(MP)和邻接法(NJ)构建系统发育树。结果显示:在鳚亚目鱼类的 16S rRNA片段生成的序列矩阵中发现有碱基的插入缺失现象, 共有207 bp变异位点, 转换/颠换值为0.8, 碱基平均差异为3.36; 支持绵鳚(Enchelyopus elongates)归于鳚亚目绵鳚科(Zoarcidae),鳚(Azuma emmnion)归于鳚亚目线鳚科(Stichaeidae); 方氏云鳚(Enedrias fangi)和云鳚(Enedrias nebulosus)种间遗传距离只有0.01, 亲缘关系最近。
鳚亚目(Blennioidei); 线粒体DNA; 16S rRNA基因序列; 系统发育
鳚亚目(Blennioidei)鱼类是鲈形目(Perciformes)中较大的一个亚目, 已有标本和文献资料显示, 中国海区现有鳚亚目鱼类8科33属78种[1], 多为海洋鱼类, 世界性分布。由于其大多体型较小, 所以以鳚亚目鱼类为研究对象的报道相对较少。目前, 国内外对鳚亚目鱼类的研究较少, 大多局限于形态分类学方面[2-6], 对其分类区系、系统发生, 尤其是分子水平的系统发生研究更是鲜有报道。而鱼类亲缘关系等系统发生进化方面的研究是鱼类生物学的一个重要组成部分, 针对鳚亚目鱼类亚目与科之间及属种之间的系统关系仍不明确的现状[7-9], 研究鳚亚目鱼类分子系统进化关系具有重要意义。
线粒体 DNA(mitochondrial DNA, 简称mtDNA)结构简单, 母性遗传, 进化速度快且不发生重组, 是一种应用较广的分子标记[10-13], 其中一些基因已经被广泛用于系统进化研究[14-17]。16S rRNA的结构既具有保守性区域又具有高变性区域。保守性区域的变异能够反映物种的亲缘关系, 为系统发育重建提供线索; 高变性区域的变异能揭示出生物物种的特征核苷酸序列, 是属种鉴定的分子基础[18]。因此基于其在分类分析中的应用, 一般认为16SrRNA适用于种以上水平的变异分析[19]。作者以线粒体16S rRNA基因作为分子标记对鳚亚目的11种鱼类的部分序列进行分析, 应用NJ、MP 等法构建系统发育树, 通过分析其基因序列变异及种群遗传结构, 以便为鳚亚目物种的分类提供可参考的依据。
1 材料与方法
1.1 材料
实验材料为鳚亚目鱼类, 采样地点为以辽宁大连黑石礁附近海域为主的黄渤海海域(表1), 采集后在–20℃下保存待用。
研究直接测定了黄渤海鳚亚目鱼类共计 3科 5属 6种, 并结合 GenBank下载其他 5种鳚亚目 16S rRNA序列的相应片段, 共计7科10属11种鳚亚目鱼类进行分析, 并以鲈形目南极鱼亚目(Notothenioidei)鳄冰鱼科(Channichthyidae)的眼斑雪冰鱼(Chionodraco rastrospinosus)作为外类群, 用于构建系统发育树(表2)。
1.2 方法
1.2.1 基因组DNA的提取
取约0.05 g肌肉放入1.5 mL的离心管中, 加400 μL DNA裂解液, 加入 20 μL的10%的 SDS, 剪碎,再加3 μL蛋白酶K, 于55℃加热2 h, 每隔15 min轻摇一次, 加入 100 μL乙酸钾混匀, 静置 30 min,冷冻离心10 min(12 000 r/min, 4 ℃), 将上清液吸入另一个管中, 加1 mL 100%冰乙醇, 4 ℃或者–20 ℃放1 h, 冷冻离心, 将沉淀用4 ℃下 70 %冰乙醇洗涤2次(一次加200 μL), 室温干燥30 min, 加TE或双蒸水(dd H2O)50 mL溶解, 冰箱保存备用。
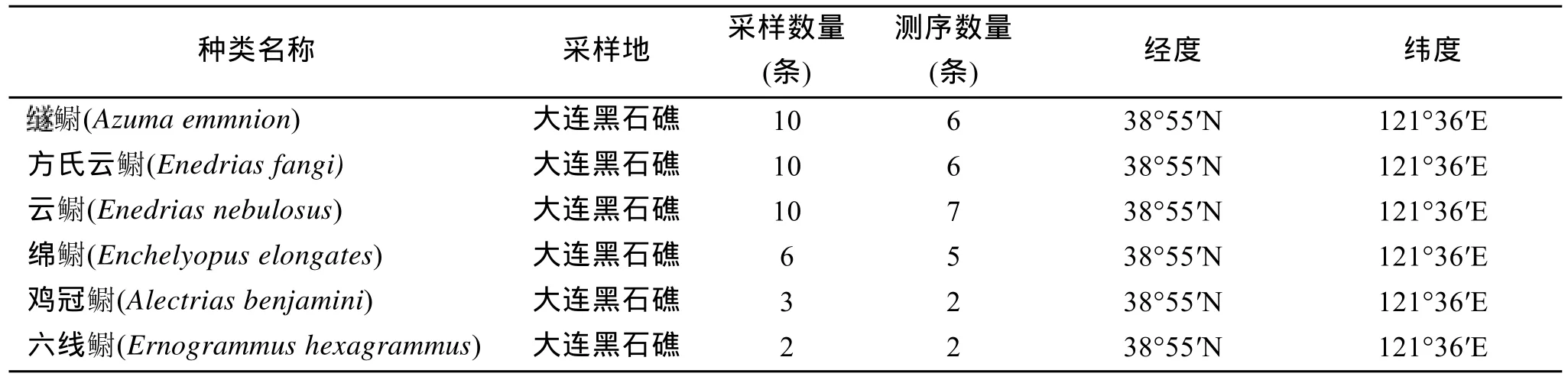
表1 鳚亚目鱼类采样地及采样和测序数量Tab. 1 Locality and numbers of Blennioidei

表2 实验所选用的鳚亚目鱼类16SrRNA基因信息Tab. 2 Information for 16SrRNA genes of Blennoidei
1.2.2 线粒体16SrRNA基因片段扩增
用于PCR扩增的引物为自行设计的16SAR(5’-TTAGTTTGGTGTCCCGAA-3’)和 16SBR(5’-GCAAGAGGCGATGTTTTT-3’), 由上海生工生物工程公司合成。PCR反应体系总体积为25 μL, 包括 2.0 mmol/L MgCl2, 0.2 mmol/L dNTP, 0.2 μmol/L每种引物,1 UTaqDNA聚合酶(TAKARA), 1×Taq聚合酶缓冲液以及50 ng基因组DNA。PCR反应程序为:95℃预变性3 min;94℃变性30 s, 56℃退火45 s,72℃延伸1 min, 共计35个循环; 最后72℃延伸10 min。扩增产物经琼脂糖凝胶回收纯化, 送至上海生工生物工程公司进行测序。
1.3 数据分析
用 DNAstar 和 ClustalX1.8 软件分析已比对验证过的鳚亚目16SrRNA基因序列[20], 用MEGA 4.0中的 P-Distance模型计算种内和种间遗传距离[21],采用邻接法 (neighbour-joining, NJ)、最大简约法(maximum parsimony, MP) 构建进化系统树, 系统树各分支的置信度采用重抽样法( bootstrap) , 通过1 000次循环评估系统树的可靠性。用DNA sp 5.10统计单倍型[22]。
2 结果
2.1 鳚亚目鱼类16SrRNA基因碱基组成
研究所用引物位于 16S基因 5′端, 扩增片段长度范围为651~674 bp, 平均669 bp。将实验获得序列连同 GenBank下载序列一并比对, 比对后序列同源长度为578bp(含插入缺失位点)。分析结果表明, 38个个体共存在 31个单倍型, 单倍型变异指数为0.0000~1.0000(平均0.6259), 核苷酸多样性为0.0000~0.04349(平均 0.0091); 11个物种中 45.45%的物种存在碱基插入和缺失现象, 共存在27个插入缺失位点, 其中, 方氏云鳚插入缺失现象最严重, 插入缺失位点数为12个; 多态性位点共计113个, 平均每个物种的多态性位点数为 10个, 其中鳚的多态性位点数最多(52)(表3)。38条鳚亚目鱼类 16S rRNA 序列的平均碱基组成分别为 T:23.10%、C:24.80%、A:30.50%、G:21.70%, 其中, A+T 含量(53.60%)明显高于 G+C含量(46.40%), 碱基组成表现出明显的偏倚性。3个密码子GC含量差异较大(表4), 其中第2密码子GC平均含量最高, 达51.50%; 第3位密码子次之, 平均为45.70%, 第1密码子最小, 平均仅为42.20%。表5列出了16S rRNA序列的核苷酸变异情况。在578个位点中, 不变位点有371个, 第1、2、3密码子的不变位点相当, 分别为120、127和123个; 第3密码子位点上的转换频率最高, 为15.88%; 第1密码子位点上的颠换频率最高, 为18.44%。
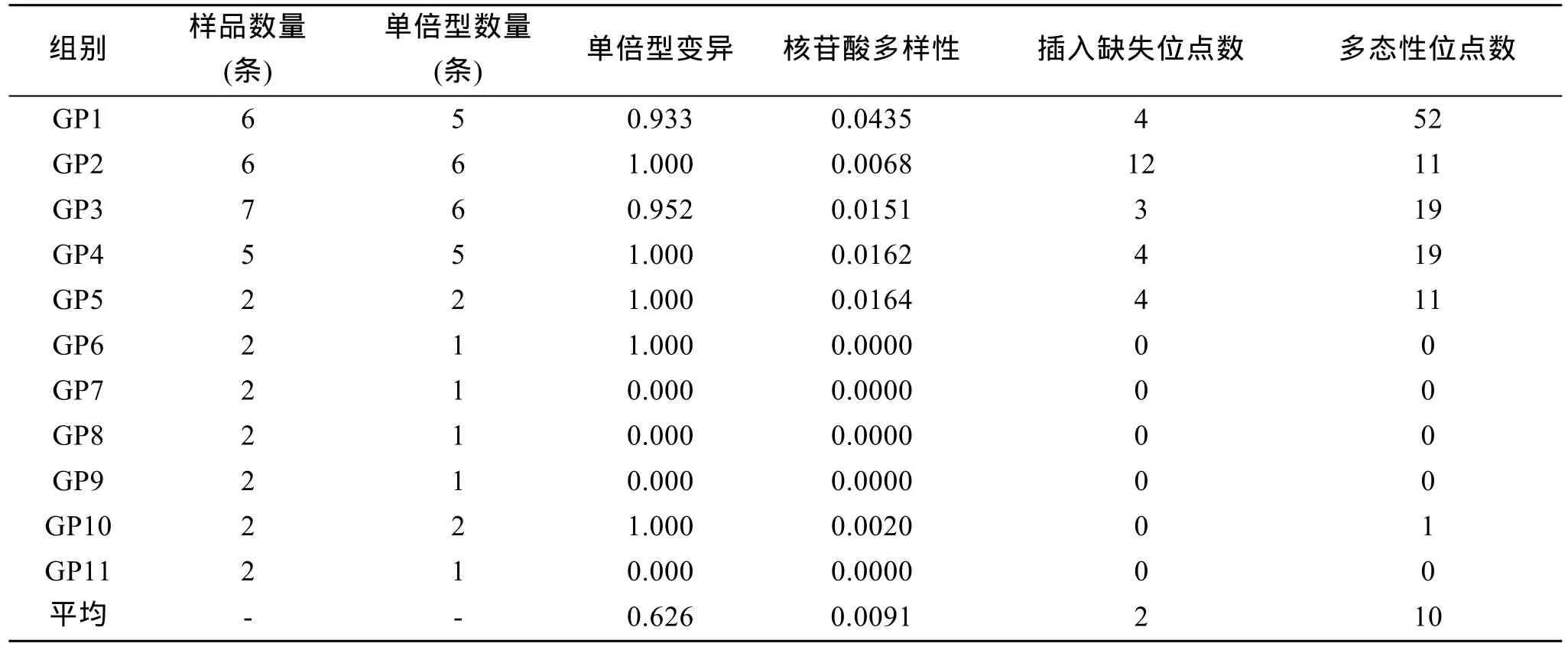
表3 11种鳚亚目鱼类16SrRNA基因序列特征Tab. 3 Characteristics of 16SrRNA sequences from 11 species of Blennoidei

表4 11种鳚亚目鱼类16SrRNA基因部分序列碱基平均分布概率(%)Tab. 4 Average nucleotide frequencies of 16SrRNA partial sequences of 11 Blennoidei (%)

表5 11种鳚亚目鱼类38个个体的16SrRNA部分序列密码子变异情况Tab. 5 Sequence variation of 16SrRNA gene of 11 Blennoidei species
2.2 种内及种间遗传距离
应用MEGA 4.0中P-Distance模型计算38条鳚亚目鱼类 16S rRNA基因序列的种内和种间的遗传距离(表6)。11个物种的种内平均遗传距离为0.0064,81.82%的种内遗传距离为0.0000。其中,鳚种内遗传距离最大(0.05)。11个物种的种间平均遗传距离为0.7236, 是种内遗传距离的113.06倍。其中, 同属于锦鳚科的方氏云鳚与云鳚之间的种间遗传距离最小(0.010)。

表6 11种鳚亚目鱼类的种间及种内遗传距离Tab. 6 Genetic distance within-species and between 11 Blennoidei species
2.3 鳚亚目鱼类分类及系统发育关系
基于鳚亚目鱼类的16S rRNA部分序列片段, 以眼斑雪冰鱼为外类群对鳚亚目进行系统发育分析。应用 MP和邻接分析法(NJ)法构建了分子系统发育关系树(图1)。用 1000次 Bootstrap(BP)表示统计分析后对该支的支持百分数, 其数值标于树分支的上面。采用MP法将鳚亚目鱼类分成两个类群, 其中方氏云鳚、云鳚、鳚、鸡冠鳚、绵鳚和与 Genebank下载的云鳚(FJ687233Enedrias nebulosus)聚为一群(I),花体草鳚、毛鳚、六线鳚、三鳍鳚、鲍乔无眉鳚和黄喉烟管鳚聚为另一群(II)。第一类群中绵鳚位于进化树的基部, 其他4种鱼分为两支, 其中方氏云鳚、云鳚和Genebank下载的云鳚聚在一起,鳚和鸡冠鳚聚在一起。另一类群中花体草鳚位于系统树的基部, 而毛鳚和黄喉烟管鳚聚在一起为一独立分支,鲍乔无眉鳚、六线鳚和三鳍鳚为另一分支, 其中六线鳚与三鳍鳚又聚在一起。邻接分析法所得系统树与MP树的拓扑结构略有差异(图1) 。NJ 树同样以极高的置信度值(100%) 将鳚亚目鱼类分成两个类群,不同的是第一分支明显分为两支, 云鳚和方氏云鳚聚在一起分支, 绵鳚、鸡冠鳚和鳚聚在一起。黄喉烟管鳚位于第二分支的基部。
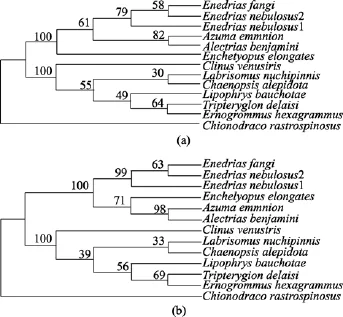
图1 MP法(a)、NJ 法(b)构建鳚亚目鱼类系统发育树Fig. 1 MP (a) and NJ (b) tree based on the analysis of 16SrRNA genes from 11 Blennoidei species
3 讨论
3.1 鳚亚目鱼类线粒体16SrRNA基因结构特征
本研究扩增了6种鳚亚目鱼类的16S rRNA 基因片段序列,其中六线鳚,方氏云鳚和云鳚在GenBank中已有记录, 与其对应物种进行相似性检验发现, 二者相似性均在 95% 以上, 说明同源性较高, 适合做下一步的分析。基于16S rRNA序列计算得到的转换颠换比(Si/Sv)小于 2.0, Knight[23]认为该比例小于2.0则说明突变已经达到饱和状态, 受进化噪音影响的可能性较大, 在生物的进化过程中, 转换较颠换更易发生, 因此本研究得到的数据间接说明了鳚亚目鱼类之间维持着一定的遗传变异水平。本研究得到的鳚亚目鱼类 16S rRNA 基因片段存在AT偏倚现象, 在碱基组成中, A+T含量明显高于G+C, 这一结果与其他研究者在无脊椎动物和鱼类等得到的结果相吻合[24-26]。此外,本研究得到的鳚亚目鱼类16S rRNA基因存在碱基插入缺失现象, 这种现象在脊椎动物中较为常见, 郑文娟[27]等认为科鱼类16SrRNA基因存在着碱基插入缺失现象。
3.2 鳚亚目鱼类分类关系的探讨
从表3~表6可见, 利用16S rRNA基因序列对中国黄渤海海域的 6种鳚亚目鱼类的碱基差异进行比较,发现方氏云鳚与云鳚的碱基差异非常小, 种间遗传距离仅为 0.01, 这说明方氏云鳚和云鳚亲缘关系很近,而六线鳚与其他 5种鱼的碱基差异比较大,亲缘关系较远。由图1可见, 中国黄渤海海域的5种鱼类在分类地位上较Genebank下载的关系较远。但是从图1可以看出, 六线鳚与Genebank中的三鳍鳚聚为一支, 推测它们可能具有较近的亲缘关系。从进化树的结果来看, 与传统形态学分类结果大致一致。李明德[7-8]等以鳚亚目鱼类为研究对象, 根据形态学特征将鳚亚目鱼类共分为两个亚目, 其中将锦鳚科、绵鳚科和线鳚科归为绵鳚亚目,三鳍鳚科、鳚 科和矶鳚科归为鳚亚目, 将鳚归为锦鳚科; 刘静[1]等从鳚亚目地理分布角度进行研究, 得到与李明德不一致的结论,其将锦鳚科、绵鳚科和线鳚科归为鳚亚目,将鳚归为线鳚科; 刘禅馨[9]等对鳚亚目鱼类的形态学研究与刘静的研究结果具有相似之处, 其将锦鳚科和线鳚科归为鳚亚目,绵鳚科归为绵鳚亚目,将鳚归为线鳚科,绵鳚归为绵鳚科。而本研究中,线鳚科的六线鳚与鳚科、矶鳚科及三鳍鳚科的鱼聚为一支,鳚和鸡冠鳚聚在一起, 支持刘静[1]和刘禅馨[9]的分类结果; 绵鳚与鳚、鸡冠鳚聚为一支, 支持刘静[1]等将绵鳚划归至鳚亚目的结果。本研究结果还显示方氏云鳚、云鳚、六线鳚和三鳍鳚处于系统进化树的顶端, 代表着最新演化的种类。此外, 从形态学上六线鳚与鸡冠鳚同属线鳚科, 而系统发育树的结果显示鸡冠鳚与鳚聚在一起, 可以认为可能属于同一科,六线鳚与三鳍鳚聚在一起, 可能属于同一科, 这与传统的形态学分类结果有一定出入。有分子系统学家认为, 当所分析的序列长度一定时, 尽量选择较多的分类群有助于获得更准确的结论[28-29],由于本研究的物种类别及数量有限, 因此关于六线鳚和鸡冠鳚的分类地位和亲缘关系等还有待于进一步研究论证。在研究鳚亚目时对比 MP、NJ两种不同的系统发育方法发现, MP树中绵鳚和黄喉烟管鳚所处的位置与在NJ树中的不一致, 推测原因可能是以NJ法为代表的距离法可以使种间的进化距离在度量树中得到直观的体现, 但由于无法避免将性状数据转化为距离时的信息丢失; 而最大简约法(MP) 可以解决空位问题, 对进化过程假设较少, 但其基于最小进化原则的合理性仍存在争议[30]。
3.3 方氏云鳚和云鳚的分类地位
方氏云鳚和云鳚同属于鳚亚目, 锦鳚科, 云鳚属。云鳚属主要形态特征是体侧扁而延长, 似带状,被鳞; 口小, 斜裂。上下颌及犁骨齿细小; 颚骨无齿;鳃盖膜宽而相连, 与峡部分离, 被圆鳞, 细小, 无侧线。背鳍低而延长, 均由鳍棘组成, 始于头后;臀鳍亦延长, 前方有二鳍棘, 胸鳍短圆, 腹鳍短小,喉位, 具一鳍棘及一鳍条, 尾鳍短圆, 无后匙骨; 躯干部脊椎骨横突合成脉弧,无幽门盲囊[31]。在传统形态学分类中,云鳚与方氏云鳚这2个种的主要特征很接近, 唯有头长与胸鳍长的比例有些差别(头长为胸鳍长的两倍以上者为云鳚, 1.5倍左右者为方氏云鳚), 所以一般在形态上很难准确区分两者。为了更好地探讨云鳚和方氏云鳚的亲缘关系, 构建了16SrRNA的系统进化树。从分子水平看,本研究得到的方氏云鳚和云鳚的种间遗传距离最小(仅为0.01), NJ和MP树中显示,方氏云鳚和云鳚二者聚在一起, 表示两者亲缘关系较近。有些学者则认为在进行物种系统进化研究时, 由于一些变异不能及时积累, 导致较保守的基因不适用于分析一些进化年龄较短、分化速度快的物种间的亲缘关系[32-33], 而且单个基因能提供的信息往往较为有限[34]。因此说明16S rRNA基因可能更适合科水平及以上分类阶元的系统发育研究, 不适于种水平间的研究。若要更好地阐明鳚亚目鱼类系统进化问题, 还需更大范围地采集更多种类及更大数量的样本进行进一步验证; 同时应采用进化速率不同的多个基因(包括线粒体和细胞核基因)对鳚亚目进行研究, 以保证结论的可靠性。基于此, 鳚亚目鱼类系统进化关系仍需进一步深入研究。
[1]刘静, 田明诚. 中国鳚亚目鱼类地理分布和区系特征[J]. 海洋科学集刊, 2006, 47:158-168.
[2]毕远溥.方氏云鳚渔业生物学及其在辽宁沿海的渔业[J]. 水产科学, 2005, 24(9):27-28.
[3]姜志强, 孟庆金, 苗治欧.大连地区方氏云鳚繁殖生物学的研究[J]. 大连水产学院学报, 1997, 12 (3):1-6.
[4]姜志强, 秦克静.大连地区方氏云鳚的年龄和生长[J]. 大连水产学院学报, 1990, 5(1):33-41.
[5]Domingues V S, Almada V S, Santos R S, et al. Phylogeography and evolution of the tripleWnTripterygion delaisi(Pisces, Blennioidei)[J]. Marine Biology, 2007,150(3):509-519.
[6]Akihiko Yatsu. Phylogeny of the family pholididae(Blennioidei) with a redescription ofPholis scopol I[J].Japanese Journal of Ichthyology, 1985, 32:273-282.
[7]李明德, 罗阳, 郭斌. 中国鱼类名录Ⅻ-鳚亚目、鱼衔亚目、喉盘鱼亚目、鰕虎鱼亚目、微体鱼亚目、刺尾鱼亚目、带鱼亚目、鲭亚目、鲳亚目、金枪鱼亚目、攀鲈亚目、刺鳅亚目[J]. 海洋通报, 2003, 10(5):64-70.
[8]李明德, 罗阳, 郭斌. 中国鱼类名录 XI-—隆头鱼亚目、尤鰧鱼亚目、绵鳚亚目、玉筋鱼亚目[J]. 海洋通报, 2003, 10(4):64-70.
[9]刘禅馨, 秦克静. 辽宁动物志[M]. 辽宁:辽宁科学技术出版社, 1987. 295-307.
[10]Avise J C, Arnold J, Ball R M, et al. Intraspecific phylogeography:the mitochondrial bridge between population genetics and systematics[J]. Annu Rev Ecol Syst,1987, 18:489-522.
[11]Brown W M. Evolution of animal mitochondrial DNA[M]. Mass:Sinacer Associates, 1983:62-68.
[12]Wilson A C, Cann R L, Carr S M, et al. Mitochondrial DNA and two perspectives on evolutionary genetics[J].Biol J Linn Soc, 1985, 26:375-400.
[13]Harrison R G. Animal mitochondrial DNA as a genetic marker in population and evolutionary biology[J].Trends Ecol Evol, 1989, 4:6-11.
[14]Pondella D J, Craig M T, Frank J P. The phylogeny ofParalabraxand allied taxa inferred from patial 16S and 12S mitochondrial ribosomal DNA sequences[J]. Mol Phylogenet Evol, 2003, 29(1):176-184.
[15]Near T J, PesaventoJ J, Cheng C H. Mitochondrial DNA, morphology, and the phylogenetic relationships of Antarctic icefishes[J]. Mol Phylogenet Evol, 2003,28(1):87-89.
[16]Clements K D, Gray R D, Howard C J. Rapid evolutionary divergences in reef fishes of the family Acanthuridae[J]. Mol Phylogenet Evol, 2003, 26(2):190-201.
[17]任岗. 12种石鲈科鱼类线粒体16SrRNA基因的部分序列分析[J]. 热带海洋学报, 2007, 26(3):48-52.
[18]江世贵, 张殿昌, 吕俊霖, 等. 3种野鲮亚科鱼类16S rRNA基因序列分析[J]. 南方水产, 2005, 3(1):1-5.
[19]蒙子宁. 黄海带鱼、小带鱼RAPD和线粒体16SrRNA基因序列变异分析[J]. 自然科学进展, 2003, 11(13):1170-1178.
[20]Hebert P D N, Cywinska A, Ball S L, et al. Biological identifications through DNA barcodes[J]. Proc R Soc Lond B, 2003a, 270:313-322.
[21]Kumar S, Tamura K, Nei M. MEGA3:Integrated software for molecular evolutionary genetics analysis and sequence alignment[J]. Briefings in Bioinformatics,2004, 5:150-163.
[22]Rozas J, Sán chez-DelBarrio J C, Messeguer X. DnaSP DNA polymorphism analyses by the coalescent and other methods[J]. Bioinformatics, 2003, 19:2496-2497.
[23]Knight A, Mindell D P. Substitions bias, weighting of DNA sequence evolution, and the phylogenetic positions of feas'viper[J]. Syst Biol, 1993, 42(1):18-31.
[24]Gao T, Zhang X, Chen D, et al. Study on mitochondrial DNA cytochrome b gene of Chinese sea bass,La-teolabraxsp.[J]. Journal of Ocean University of Qingdao, 2001, 31(2):185-189.
[25]高天翔, 张秀梅, 朱杰, 等. 养殖牙鲆细胞色素 b基因序列的初步研究[J]. 中国水产科学, 2003, 10(2):93-96.
[26]马春艳, 赵峰, 孟彦羽, 等. 基于线粒体细胞色素 b基因片断序列变异探讨 3种鲳属鱼类系统进化[J].渔业科学进展, 2009, 30(5):144-150.
[27]郑文娟, 朱世华, 邹记兴, 等. 基于 16S rRNA 部分序列探讨 12种鳞科鱼类的分子系统进化关系[J].水产学报, 2008, 32(6):847-854.
[28]Pollock D D, Zwickl D J, McGuire J A, et al. Increased taxon sampling is advantageous for phylogenetic inference [J]. Systematic Biol, 2002, 51(4):664-671.
[29]Derrick J, Hillis D M. Increased taxon sampling greatly reduces phylogenetic error[J]. Systematic Biol, 2002,51(4):588-598.
[30]丁少雄, 王颖汇, 王军. 基于16S rDNA 部分序列探讨中国近海 30种石斑鱼类的分子系统进化关系[J].动物学报, 2006, 52(3):505-513.
[31]Shaklee J B, Tamaru C S, Waples R S. Speciation and evolution of marine fishes studied by the electrophoretic analysis of proteins[J]. Pacific Science, 1982,36(2):141-157.
[32]Wang H Y, Tsai M P, Dean J, et al. Molecular phylogeny of Gobioid fishes (Perciformes:Gobioidei) based on mitochondrial 12S rRNA sequences[J]. Mol Phylogen Evol, 2001, 20(3):390-408.
[33]Summerer M, Hanel R, Sturmbauer C. Mitochondrial phylogeny and biogeographic affinities of sea breams of the genus Diplodus(Sparidae)[J]. J Fish Biol, 2001,59:1638-1652.
[34]Cao Y, Sorenson M D, Kumazawa Y, et al. Phylogenetic position of turtles among amnitotes:Evidence from mitochondrial and nuclear genes[J]. Gene, 2000,259:139-148.
Molecular phylogenetic relationship of blennioidei based on partial sequence of mitochondrial 16S ribosomal RNA gene
ZHANG Yuan-zhen, WANG Wei, JIANG Zhi-qiang, ZHANG Zhao
(Key Laboratory of Marine Aquaculture, College of Fisheries and Life Science, Ministry of Agriculture, Dalian Ocean University, Dalian 116023, Liaoning)
Sep., 01, 2011
Blennioidei; mitochondrial DNA (mtDNA); 16S ribosomal RNA (16S rRNA);molecular phylogeny
The 16S ribosomal RNA partial sequences of 6 species within 5 genera from Blennioidei in China yellow and bohai seas were amplified using PCR technique. Approximately, 669 bp gene fragments were obtained. These data were combined with other homologous sequences of 5 species from Blennioidei, which were downloaded from the GenBank.Chionodraco rastrospinosuswere designed as outgroup. Nucleotide composition frequencies,P-Distance genetic distance and transition/transversion ratios were analyzed with MEGA 4.0 software. Molecular phylogenetic trees were constructed by the maximum parsimony (MP) and neighborjoining (NJ) methods. There were insertions and deletions in the aligned base pairs. Two hundred and seven bp variable sites were found in the analysis matrix. The average ts/tv ratio and average percentage divergence among all pairwise comparisons were 0.8 and 3.36, respectively. According to the partial sequence of mitochondrial 16S ribosomal RNA gene, the classification system of Blennioidei was acceptable, which is classified as Pholidae, Zoarcidae, Stichaeidae, Clinidae,Blenniidae, Chaenopsidae and Tripterygiidae.Enedrias fangiandEnedrias nebulosushad a close relationship, as the genetic distance between the two species of partial 16S ribosomal RNA gene was only 0.01.
Q959 文献标识码:A 文章编号:1000-3096(2012)09-0089-07
2011-09-01;
2011-12-06
辽宁省重大科技公关项目(2008203002);辽宁省教育厅计划项目(2009A175)
张源真(1986–), 女, 内蒙古呼和浩特人, 硕士研究生, 主要从事鱼类群体遗传学研究, E–mail:lipc@sohu.com; 王伟, 通信作者,E–mail:wangwei@dlou.edu.cn
谭雪静)
