阪崎肠杆菌3种检测方法的应用与分析
2012-01-04牛振东刘文杰孙伟高春
牛振东,刘文杰,孙伟,高春
(北京市药品检验所,北京 100035)
阪崎肠杆菌3种检测方法的应用与分析
牛振东,刘文杰,孙伟,高春
(北京市药品检验所,北京 100035)
分别采用美国FDA、ISO/TS 22964-2006和GB4789-2010阪崎肠杆菌检测方法,同时采用缓冲液蛋白胨水(BPW)作为前增菌液,对FAPAS(Food Analysis Performance Assessment Scheme)提供婴幼儿配方奶粉进行阪崎肠杆菌定性检测,同时应用VITEK2生化鉴定和16SrDNA序列分析技术对分离的可疑菌落进行确认。结果表明:mLST-Vm选择性增菌肉汤和DFI显色平板可以提高工作效率和阳性检出率,可疑菌落易于识别,VITEK2(全自动微生物生化鉴定系统)和16SrDNA测序技术应用可以实现阪崎肠杆菌高效、快捷的检测。
婴儿配方奶粉;阪崎肠杆菌;FAPAS
0 引 言
阪崎肠杆菌(Enteribacter Sakazakii),又名黄色阴沟肠杆菌,为肠杆菌科肠杆菌属的一个种,革兰氏阴性杆菌,有周鞭毛,能运动,无芽孢,氧化酶实验阴性。1980年建议将该菌作为肠杆菌科的一新种,并改名为阪崎肠杆菌[1,10],2008年阪崎肠杆菌被重新分类一个新属,即克罗诺杆菌属(Cronobacter spp.),包括5个新种和3个亚种[13-14]。 阪崎肠杆菌(Enteribacter Sakazakii)是婴儿配方奶粉中重要的污染源,可导致婴儿脑膜炎和小肠结肠炎等疾病,其发病率高达80%[2,9],阪崎肠杆菌与沙门氏菌列为婴儿配方奶粉的A类致病菌[3]。
现代分子生物学的发展为阪崎肠杆菌检测提供了便利,Mohan等人根据阪崎肠杆菌外膜蛋白A(om-pA)基因设计引物,建立PCR检测方法[11]。本研究应用VITEK2生化鉴定系统和16S rDNA序列分析技术[16]可以实现阪崎肠杆菌的准确鉴定。
1 实 验
1.1 样品来源
2011年3月份参加FAPAS能力验证样品(婴儿配方奶粉),样品A和样品B。
1.2 培养基、试剂和仪器
培养基:月桂基硫酸盐胰蛋白胨肉汤(LST broth),肠道杆菌增菌肉汤(EE broth),结晶紫中性红胆盐葡萄糖琼脂(VRBGA),胰蛋白酶大豆琼脂(TSA),万古霉素(Vancomycin,Vm),缓冲蛋白冻水溶液(BPW),阪崎肠杆菌显色培养基(DFI琼脂)。试剂:Platinum PCR SuperMix,DL 2000 Marker,GoldView。
仪器:全自动微生物生化鉴定系统(VITEK2)(bio Merieux)
1.3 方法
1.3.1 样品前增菌:
无菌取检样10 g加入已预热至44℃装有90 mL缓冲蛋白胨水的锥形瓶中,缓慢摇动至充分溶解,37℃±1℃培养18~22 h。
1.3.2 FDA阪崎肠杆菌检测方法[4]
移取上述增菌液10 mL转种到装有90 mL无菌EE肉汤的三角烧瓶中,36℃孵育过夜。充分混合每个瓶中的增菌培养液,每份增菌液用直径3 mm(10 μL)的接种环分别分区划线接种2个VRBGA平板和2个显色培养基平板,将平板置36℃±1℃培养18~22 h。挑取VRBGA平板上的粉紫色疑似菌落划线接种TSA平板,25℃ ±1℃培养48~72 h。挑取TSA平板上黄色菌落进行营养琼脂平板纯培养,革兰染色,做氧化酶试验。革兰染色阴性,氧化酶试验阴性者进行VITEK2 compact(全自动微生物生化鉴定系统)生化鉴定。
1.3.3 ISO/TS 22964-2006[5]和GB4789-2010阪崎肠杆菌检测方法[6]
移取1 mL上述增菌液转种于10 mL的mLST-Vm肉汤,44℃ ±0.5℃培养(24±2)h。轻轻混匀过夜培养的mLST-Vm肉汤,各取增菌液1环分别划线接种于2个DFI平板,44℃±1℃培养(24±2)h(ISO/TS 22964-2006),36℃±1℃培养(24±2)h(GB4789-2010)。挑取1~5个蓝绿色,直径为1~3 mm的可疑菌落,划线接种于TSA平板,25℃培养 (48±4)h。自TSA平板上直接挑取黄色可疑菌落,进行氧化酶试验,氧化酶试验阴性的菌落进行VITEK2 compact(全自动微生物生化鉴定系统)生化鉴定。
1.3.4 VITEK2 compact生化鉴定
挑取可疑菌落(TSA平板上的纯培养物),进行革兰氏染色镜检及氧化酶实验,按照VITEK2仪器操作指南配置菌悬液 (革兰氏阳性菌GP卡和革兰氏阴性菌GN卡制成0.6~1.0麦氏浓度),选择相应的卡片上机检测。
1.3.5 16S rDNA序列分析
基因组DNA提取双歧杆菌标准菌株和样品分离得到的中双歧杆菌的基因组DNA提取方法参照文献[7]。16SrDNA序列的扩增用上述制备的基因组DNA为16SrDNA PCR扩增的模板。PCR的2个扩增引物27f(5’-AGAGTTTGATCCTGGCTCAG-3’)和1492r(5’-TACGGCTACCTTGTTACGACTT-3’)分别位于16S rDNA的8-27和1493-1514位的碱基片段(以E.coli的16S rDNA碱基位置为准)。PCR扩增的片段约为1.5 kb。PCR扩增反映程序:95℃预变性5 min,之后的30个循环包括95℃ 预变性1 min,55℃“退火”1 min,72℃延伸2min。30个循环后72℃延伸10min,最后4℃保存备用。扩增得到的PCR产物直接送到公司测序,测序由英骏生物技术有限公司完成。
2 结 果
2.1 样品检测结果
FAPAS提供的样品A和样品B,结果在A样品中未检出阪崎肠杆菌,能力验证提供的干扰菌落为大肠埃希菌,B样品中检出阪崎肠杆菌。
2.2 阪崎肠杆菌的菌落形态
VRBGA平板上呈紫红色大菌落,周围有一圈紫色的不透明的胆汁酸沉淀环,圆形凸起,直径2~3 mm。DFI平板上呈蓝绿色菌落(阪崎肠杆菌具有产生α-葡萄糖苷酶,该培养基中含有5-溴4氯-3-吲哚-α-D葡萄糖苷酶,因此,阪崎肠杆菌在此平板上呈现蓝绿色菌落)。阪崎肠杆菌在TSA平板能够快速生长,随着培养时间的延长菌落直径逐渐增大,且呈黄色菌落。菌株均为革兰阴性杆菌,菌体短杆状、卵圆形、球杆状,成对或呈链状排列,无芽胞。
2.3 VITEK2compact生化鉴定
VITEK2(全自动微生物生化鉴定系统)鉴定结果,A样品可疑菌落鉴定为大肠埃希菌,B样品中的可疑菌落鉴定为阪崎肠杆菌。检测结果如表1。

表1 样品A和B的VITEK2鉴定结果
2.4 3种标准中鉴定方法比较
参考GB4789.40-2010、FDA和ISO/TS 22964-2006方法对FAPAS提供的样品进行检测,并且比较3种检测方法的优越性,不同之处如表2所示。

表23 种检测方法的比较
2.5 16SrDNA序列分析结果
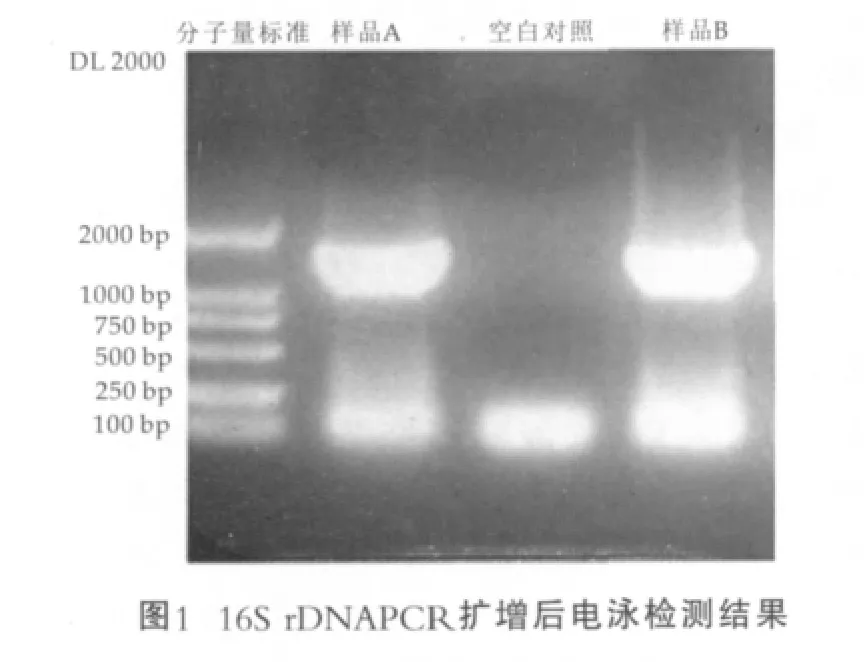
分别挑取从样品A和B中分离得到的纯培养物,A样品和B样品进行PCR扩增,在1%的琼脂糖凝胶电泳鉴定,电泳检测图谱如图1所示,同时用灭菌水作为空白对照。纯化后的PCR产物用ABI3700基因测序仪测序。测序结果经NCBI数据库比对(GenBank+EMBL+DDBJ+PDB sequences),结果样品A的16S rDNA序列与大肠埃希菌FUA1242(NCBIAccession HQ169124.1)、FUA1241(NCBI Accession HQ169122.1的16S rDNA序列同源性均达到了99%,可以推断样品A属于阪崎肠杆菌,样品B的16S rDNA序列与阪崎肠杆 菌fmb01(NCBIAccessionJF330127.1)、G4026(NCBI Accession HQ880305.1)的16S rDNA序列同源性均达到了99%,可以推断样品B属于阪崎肠杆菌。
3 讨 论
(1)FDA检验方法中使用VRBGA结晶紫葡萄糖琼脂平板于分离、鉴定阪崎肠杆菌的培养基,由于肠杆菌科的许多菌属如产气肠杆菌、阴沟肠杆菌等肠杆菌均可在VRBGA平板上形成与阪崎肠杆菌相类似的特征性菌落,因此该平板选择性差,增加了检测难度,易造成检出假阳性结果[8]。阪崎肠杆菌能产生α-D-葡萄糖苷酶,可分解底物5-溴-4-氯-3-吲哚-α-D-吡喃葡糖甙产生蓝绿色特征性菌落,易于识别,ISO+TS+22964-2006和GB4789.40-2010将选择性培养基修改为DFI,提高了检出率,也易于可疑菌落的识别。但是,从表1可以看出,ISO和GB4789的不同点在于选择性培养基的培养温度不同,ISO采用44℃,GB4789采用的是36℃,两种培养温度都能够达到检测效果,但是通过实验发现,DFI平板在44℃培养时水分蒸发太大,平板容易干燥,相反,36℃培养时平板的水分蒸发的速度减慢,平板不容易干燥,这样就能够更好的保证检测结果。
(2)EE肉汤作为增菌液,由于该肉汤适合所有肠道菌的生长,因此会造成细菌生长的竞争性抑制,不利于目标菌落的筛选,且阪崎肠杆菌可耐受较高的生长温度和耐高盐,加入万古霉素可很好的抑制革兰阳性菌的生长。ISOISO+TS+22964-2006和GB4789.40-2010将阪崎肠杆菌检测方法进行了改进,在前增菌的培养基中采用了mLST-Vm,而且培养温度提高至44°C,这样就可以有利于阪崎肠杆菌的筛选。
(3)现行的培养法作为细菌检测鉴定中的标准,在鉴定和检测过程中起着重要且决定性作用,由于培养时间长,步骤繁琐成为其的缺点。生化鉴定仍然是目前的主要手段,API20E是国标和FDA标准中规定的鉴定试验,API20E在实验操作[15],结果判断方面仍有很多不便和不足之处,需要在反应孔内添加菌悬液,培养24 h,再次添加辅助试剂利用颜色反应对结果进行判断,实验人为因素较大,需要时间长,不利于快速检测阪崎肠杆菌,因此,本研究采用VITEK2鉴定系统进行生化鉴定,缩短了检测时间,完全机器化操作,避免了人为干扰,可以实现快速高通量的鉴定。分子生物学技术的飞速发展,为进一步研究和探索适合阪崎肠杆菌检测及鉴定的分子生物学方法提供了技术支持。目前针对阪崎肠杆菌的分子生物学检测方法有BAX病原菌检测系统[12]、Riboprinter全自动微生物分析系统,16 srDNA测序的分子生物学鉴定技术。本研究中应用16SrDNA序列分析技术和VITEK2全自动生化鉴定系统对分离的细菌进行了鉴定,这两项技术检测快速,简便,检测成本较低,为快速检测与鉴定阪崎肠杆菌提供了有效的手段,同时可以为其他食源性致病菌的快速检测提供了技术支持。
[1]NAZAROWEE-WHITE M,FARBERB J M.En Terobacter Sakazakii:A Review[J].International Journal of Food Microbiology,1997(34):103-113.
[2]FARMER J J,ASBURY M A,HICKMAN F W,et al.The Enterobacteriaceae Study Group(USA):Enterobacter sakazakii:A New Species of"Enterobacteriaceae"Isolated from Clinical Specimens[J].Int JSyst Bacteriol,1980,30:569-584.
[3]Joint FAO/WHO workshop on Enterobacter sakazakii and other microorganisms in powdered infant formula,Geneva,2-5.February 2004[EB/OL].http: //www.who.int/foodsafety/publications/micro/en/es.pdf.
[4]US Food and Drug Administration Center for Food Safety and Applied Nutrition.Isolation and Enumeration of Enterobacter Sakazakii from Dehydrated Powdered Infant Formula[S].Revised,2002.
[5]ISO/TS 22964:2006.Milk and Milk Products-Detection of Enterobacter sakazakii[S].
[6]食品安全国家标准.食品微生物学检验:阪崎肠杆菌检验.GB 4789.40—2010[S].
[7]ZHU H,QU F,ZHU L H.(1993)Isolation of Genomic DNA from Plants,Fungi and Bacteria Using Benzyl Chloride.[J]Nucleic Acids Res.,1993,21:5279-5280.
[8]陆峥,王丽丽,王迪,等.三种培养基在阪崎肠杆菌检测中的应用与分析[J].中国卫生检验杂志,2009,19(3):600-602.
[9]WILLIS J,ROBINSON J E.Enterbacter Sakazakii Meningitis in Nuonates[J].Pediatric Infectious Disease Journal,1988,7:196-199.
[10]陆贞宇.阪崎肠杆菌的生物学特性及其检验技术研究进展[J].中国热带医学,2010,10(10):1275-1277.
[11]MOHAN N M K,VENKITANARAYANAN N K.Cloning and Sequencing of the OmpA Gene of Enterobacter Sakazakii and Developmentof an OmpA-Targeted PCR for Rapid Deteetion of Enterobacter Sakazakii in Infant Formula[J].Applied Environmental Microbiology,2006,72(4):2539-2546.
[12]Government of Canada Laboratory Procedure.MELP-27.Dupont Qualicon BAX System Methood For The Detection of Enterobacter Sakazakii In Selected Foods[Z].Health Products And Food Branch OTTA WA,2003.
[13]IVERSEN C,MULLANE N,BARBARA MCCARDELL,et al.Cronobacter Gennov,Cronobacter gen.nov.,A New genus to Accommodate The Biogroups of Enterobacter sakazakii,etc[J].Int J Syst Evol Microbiol,2008,56(6):1442-1447.
[14]JEAN EUZÉBY.Notification That New Names and New Combinations Have Appeared in volume 58,part 6,of the IJSEM[J].Int.J.Systematic Evolutionary Microbiol,2008,58(6):1995-1996
[15]IVERSEN C,WADDINGTON M,FARMER J J,et al.The Biochemical Differentiation of Enterobacter Sakazakii Genotypes[J].BMC Microbiol,2006,6:94.
[16]焦振全,刘秀梅.16S rRNA序列同源性分析与细菌系统分类鉴定[J].国外医学:卫生分册,1998.25(1):12-16.
Analyzing and application three detection methods for Enterobacter sakazakii
NIU Zhen-dong,LIU Wen-jie,SUN Wei,GAO Chun
(Beijing Institute for Drug Control,Beijing 100035,China)
Using the U.S.FDA,ISO/TS 22964-2006 and GB4789-2010 of E.sakazakii detection methods,BPW pre-enrichment broth for detection of E.sakazakii,in FAPAS(Food Analysis Performance Assessment Scheme)providing infant formula Formula,while application of VITEK2 compact system and 16SrDNA sequencing to confirm the perspective colonies.The results showed that mLST-Vm selective enrichment broth and DFI chromogenic panel can improve efficiency and positive rate,easy to identify suspected colonies.,Enterobacter sakazakii were detected rapidly and accurately by VITEK2 compact system and 16SrDNA sequencing.
infant formula;Enterobacter sakazak ii;FAPAS
TS252.7
A
1001-2230(2012)09-0042-04
2012-04-09
牛振东 (1979-),男,主管药师,从事保健食品、药品微生物限度检查。
