平菇基因组DNA提取方法的研究
2011-11-02许文涛苏春元罗云波王一南谷新晰戴蕴青田洪涛
闫 燕,许文涛,苏春元,罗云波,王一南,谷新晰,戴蕴青,田洪涛,*
(1.河北农业大学食品科技学院,河北保定071001; 2.中国农业大学食品科学与营养工程学院,北京100083; 3.农业部转基因生物食用安全监督检验测试中心(北京),北京100083)
平菇基因组DNA提取方法的研究
闫 燕1,2,3,许文涛2,3,苏春元2,罗云波2,王一南3,谷新晰1,戴蕴青2,3,田洪涛1,*
(1.河北农业大学食品科技学院,河北保定071001; 2.中国农业大学食品科学与营养工程学院,北京100083; 3.农业部转基因生物食用安全监督检验测试中心(北京),北京100083)
以实验室自行培育的平菇为实验材料,分别使用CTAB法、SDS-CTAB法以及高盐酶解法提取平菇基因组DNA,并通过紫外分光光度计、限制性酶切及PCR检测来衡量基因组质量。结果表明,使用CTAB法很难得到高质量的基因组DNA,OD260/280仅为1.5左右,而且伴有较多的杂质,电泳条带拖尾严重;使用EcoRI限制性酶对其进行酶切分析,效率较低,同时PCR检测不能得到有效扩增;而使用优化后的SDS-CTAB法及高盐酶解法提取平菇DNA,能够获得质量高、纯度好的基因组DNA,OD260/280基本保持在1.8~2.0之间,电泳条带较为清晰均一,使用EcoRI限制性酶对其进行酶切分析,酶切效果较好,并且DNA质量能够满足PCR检测的模板要求。说明SDS-CTAB法以及高盐酶解法提取的平菇DNA能够满足分子水平操作的要求,两者相比,前者成本较低,后者产率较高。
平菇,基因组DNA,提取方法
1 材料与方法
1.1 材料与仪器
平菇 实验室自行培育、分离,研究部分为菌皮、菌肉、菌褶以及菌柄四部分,分别标记为1、2、3、4号;DL2000 DNA marker(300ng/5μL) 大连宝生物工程有限公司;RNase、溶壁酶(500U/mL)、Taq DNA聚合酶(5U/μL)、各10mmol/L的四种脱氧核糖核苷酸(dATP,dCTP,dGTP,dTTP)混合液 购自Sigma (美国)公司;CTAB缓冲液,100mmol/L Tris-HCl (pH8.0),6mol/LNaCl,20mmol/LEDTA,20g/L CTAB,苯酚/氯仿溶液1∶1(v/v),10×PCR Buffer,100mmol/L Tris-HCl,500mmol/L KCl,15mmol/L MgCl2,0.2%β-巯基乙醇(v/v),2%SDS缓冲液,蛋白酶K(20mg/mL),1×TE缓冲液,1mol/L Tris-HCl (pH8.0)100mL,500mmol/L EDTA(pH8.0)20mL;其它试剂或溶剂 均为分析纯或生化纯。
电热恒温水浴锅HH-SY2HUI 北京市长风仪器仪表公司;ASP-3700型紫外分光光度计 美国ACTGene公司;电子天平FA100413 上海越平科学仪器有限公司;电泳仪 北京市六一厂;PHX型智能生化培养箱 宁波莱福科技有限公司;PCR仪 BIO -RAD ALD-1244;紫外投射仪GelDoc-It PN 95-0441-02 ENFO-980179振荡摇床。
1.2 DNA的提取方法
1.2.1 CTAB法 摘取平菇菌盖、菌肉、菌褶及菌柄四部分,加入少许液氮进行研磨,分别称取大约200mg样品,加入800μL预热到65℃的CTAB提取液,充分振荡,放入65℃水浴锅中1~2h。取出EP管,于4℃,12000r/min离心10min。吸取1mL上清液移入新管中,加入等体积的饱和酚∶氯仿∶异戊醇(25∶24∶1),颠倒混匀,于 4℃,12000r/min离心10min。吸取上清液移入新管中,加入等体积氯仿∶异戊醇(24∶1)。小心倒掉上清液,加入2/3体积的异丙醇,混匀后,置于-20℃下放置30min。取出样品,于4℃,12000r/min离心10min,倒掉上清液后,加入700μL 70%乙醇,用枪头吹吸,使沉淀重悬于乙醇中,于4℃,12000r/min离心10min,小心倒掉上清液,将离心管倒置于吸水纸上,控干上清液,置于超净工作台上吹干10~15min。晾干后,加入50μL提前在水浴锅中预热的灭菌水,使沉淀充分溶解,加入2μL RNase放置于37℃水浴锅中消化30min,即为所得DNA提取液。
1.2.2 SDS-CTAB提取法 摘取新鲜的平菇菌盖、菌肉、菌褶及菌柄四部分,加入少量海砂,进行液氮研磨,取2.0mL EP管提前放入液氮遇冷,分别称取大约200mg样品,迅速加入 600μL预热到 65℃的CTAB提取液,充分重悬沉淀,放入65℃摇床中1h,1200r/min。取出样品,加入400μL SDS提取液和3μL蛋白酶K,充分混匀并放回摇床中。取出 EP管,于4℃,12000r/min离心10min。吸取1mL上清液移入新管中,加入等体积的氯仿∶异戊醇(24∶1),颠倒混匀,于4℃,12000r/min离心10min。吸取上清液移入新管中,重复前一步操作。小心倒掉上清液,加入2/3体积的异丙醇,混匀后,置于-20℃下放置30min。取出样品,于4℃,12000r/min离心10min,倒掉上清液后,加入700μL 70%乙醇,用枪头吹吸,使沉淀重悬于乙醇中,于4℃,12000r/min离心10min,小心倒掉上清液,将离心管倒置于吸水纸上控干上清液,置于超净工作台上吹干10~15min。晾干后,加入50μL提前在水浴锅中预热的灭菌水,使沉淀充分溶解,加入2μL RNase放置于37℃水浴锅中消化30min,即为所得DNA提取液。
1.2.3 高盐酶解法 摘取平菇菌盖、菌肉、菌褶及菌柄四部分,加入少量海砂,进行液氮研磨,称取大约200mg样品,加入400μL TE缓冲液,充分振荡。向每支样品加入2μL溶壁酶,颠倒混匀,放入摇床30℃,1200r/min,1h。取出样品,于4℃,12000r/min离心10min,小心将上清液移入新管中,加入400μL CTAB提取液充分混匀,放入摇床中65℃,1200r/min,30min。取出样品向其中加入200μL SDS提取液和3μL蛋白酶K,放回摇床中65℃,1200r/min,30min。取出样品,于4℃,12000r/min离心10min,吸取1mL上清液移入新管中,加入等体积的氯仿∶异戊醇(24∶1),颠倒混匀,于4℃,12000r/min离心10min。吸取上清液移入新管中,重复上述抽提一次,吸出上清液,加入600μL异丙醇,1/5体积的醋酸钾,混匀后,置于-20℃放置下20min。取出样品,于4℃,12000r/min离心 10min,小心倒掉上清液,加入700μL 70%乙醇,用枪头吹吸,使沉淀重悬于乙醇中,于4℃,12000r/min离心10min,小心倒掉上清液,将离心管倒置于吸水纸上晾干上清液,置于超净工作台上吹干10~15min。晾干后,加入50μL提前在水浴锅中预热的灭菌水,使沉淀充分溶解,加入2μL RNase放置于37℃水浴锅中消化30min,即为所得DNA提取液。
1.3 平菇基因组DNA的电泳检测及纯度分析
基因组DNA提取后,用1×TAE溶液配制1%的琼脂糖液,加入0.5μg/mL的EB,制成琼脂糖凝胶。取2μL DNA溶液与0.5μL点样缓冲液混合后点样。随后在120V恒定电压条件下进行电泳,时间约为20min。电泳结束后,将琼脂糖凝胶置于凝胶成像仪上或紫外投射仪上成像,分析电泳结果。
取提取后的平菇DNA溶液2μL,用ASP-3700型紫外分光光度计测定260nm和280nm下的紫外吸收值及所提取样品的浓度。
1.4 平菇基因组DNA的酶切分析
在0.5mL离心管中依次加入3μg平菇DNA,2μL 10×Buffer,0.5μL EcoRI(10U/μL)以及2μL乙酰BSA(10μg/μL),加无菌ddH2O至终体积20μL,瞬时离心后在37℃水浴锅中温育4h。用1%琼脂糖凝胶对酶切产物进行电泳(120V恒压电泳20min),电泳结束后,将琼脂糖凝胶置于凝胶成像仪上或紫外投射仪上成像,分析酶切结果。
1.5 PCR检测
对所提基因组DNA,选用18sRNA作为内标进行PCR扩增,2%的琼脂糖凝胶电泳检测DNA的质量。引 物 序 列 为 Primer - F 5'-CCTGAGAAACGGCTACCAT-3',Primer-R5'-CGTGCTAGGATTGGGTAAT-3'(上海生工生物工程有限公司合成)。PCR反应的总体积为30μL,其中,10×PCR buffer 3μL,dNTP 2.4μL,Primer-F 1.5μL,Primer-R 1.5μL,Taq酶0.2μL,模板2μL(约50ng)。扩增条件:95℃3min,95℃15s,58℃5min,72℃10min,35个循环,反应仪器为ABi2700 thermal cycle。
2 结果与分析
2.1 DNA的纯度及浓度
3种方法提取的DNA质量结果见表1~表3和图1,图表中编号A、B、C分别表示CTAB法、SDSCTAB法以及高盐酶解法,1~4分别表示平菇的菌皮、菌肉、菌褶、菌柄四个部位。紫外分光光度计测定的260nm和280nm下的紫外吸收值分别代表了盐浓度、核酸、蛋白质等有机物的吸光度值。高质量的DNAOD260/280应在 1.8~2.0之间,当 OD260/280小于 1.8时,DNA中存在蛋白质的污染,当 OD260/280大于2.0时,DNA提取物中RNA含量较高。从表中数据分析得出,在方法B、C中OD260/280基本保持在1.8~2.0之间,说明SDS-CTAB法以及高盐酶解法所提取的DNA质量较好;CTAB法中OD260/280值低于1.8,说明溶液中的蛋白质等有机物的污染比较明显。
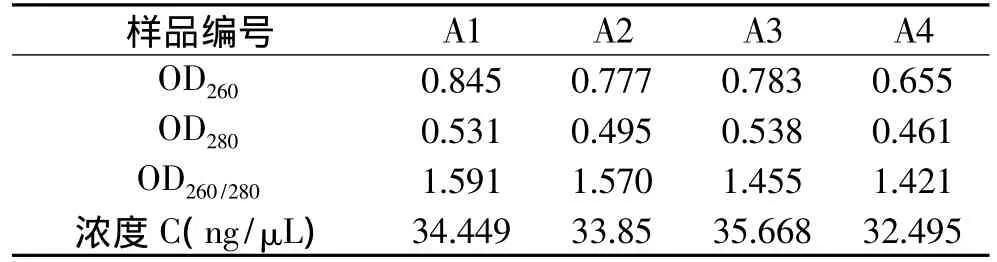
表1 CTAB法
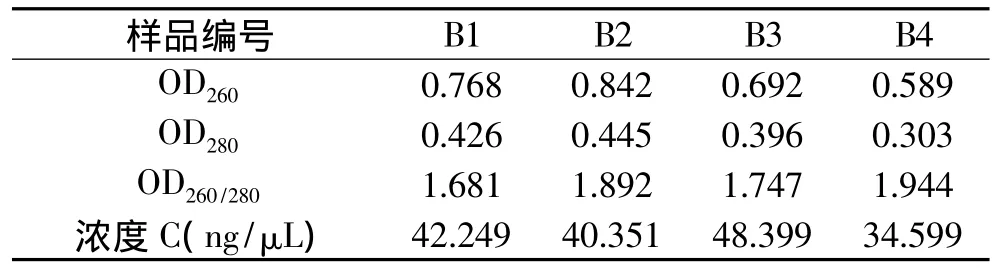
表2 SDS-CTAB法
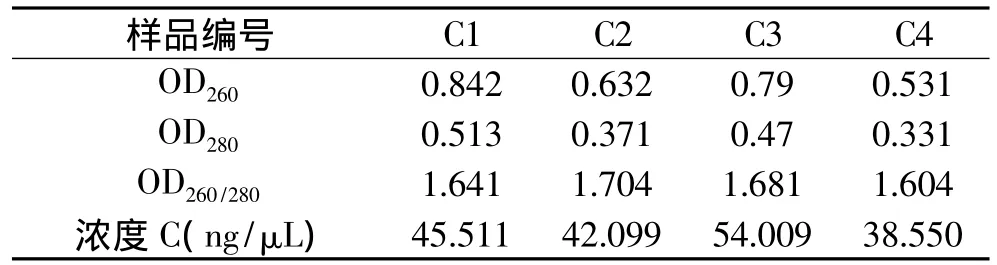
表3 高盐酶解法
2.2 DNA分子的完整性
DNA电泳结果表明(图1),CTAB法提取的DNA量较少,而且存在严重拖尾现象,并且有断裂DNA干扰,经RNase消化后,仍有很多RNA残留。使用SDS-CTAB提取的平菇各组织DNA产率相对较高,电泳条带无拖尾,经RNase酶消化后,仅残留少量的RNA。与上述两种方法相比,高盐酶解法提取的平菇各组织DNA产率最高,而且电泳条带均一。其次,通过比较可以看出,经过相同时间的RNase消化,CTAB法提取的平菇基因组 DNA中RNA残留量最多,若要进行后续实验,需延长RNase酶的消化时间。
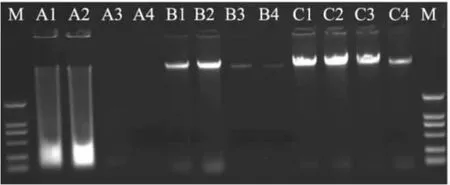
图1 三种方法提取平菇基因组DNA的电泳图谱
2.3 平菇基因组DNA的酶切鉴定
对使用CTAB法(A1~A4)、SDS-CTAB法(B1~B4)以及高盐酶解法(C1~C4)提取的平菇DNA进行酶切鉴定,酶切产物的电泳图谱如图2所示。可以看出,CTAB法所提取的平菇DNA条带有拖尾现象,很难被EcoRI酶切;而SDS-CTAB法、高盐酶解法所提取的平菇DNA样品都能被EcoRI酶切,酶切图谱呈弥散状,说明高质量的基因组DNA杂质很少,而且其盐浓度不会抑制限制性内切酶的活性,而质量较差的基因组DNA限制性内切酶的酶切效率很低,这两种方法提取的基因组DNA纯度较好,可以满足酶切等分子生物实验的要求。
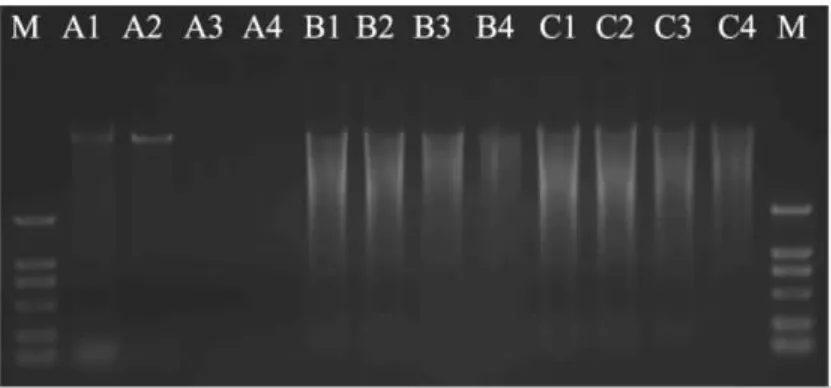
图2 三种方法提取的基因组EcoRI酶切图谱
2.4 平菇基因组的PCR扩增分析
以CTAB法、SDS-CTAB法以及高盐酶解法提取的DNA进行PCR扩增,所得PCR产物电泳图谱如图3所示。以CTAB方法提取的基因组作为模板,如A1~A4所示,电泳条带模糊,甚至不能得到有效扩增,很可能是由于RNA、多糖、多酚等杂质的存在,从而影响了基因组DNA的纯度,抑制了PCR反应的进行;而以SDS-CTAB法、高盐酶解法所提取的平菇组织DNA为模板扩增的目的条带清晰、整齐,扩增效率高。结果表明,SDS-CTAB法、高盐酶解法两种优化方法能够有效地提取高质量的平菇基因组DNA,并且可以满足相关分子生物学实验的要求。

图3 PCR扩增图谱
3 讨论
为获得高质量、高纯度的DNA基因组,本实验选择了平菇的菌皮、菌肉、菌褶、菌柄分别进行提取。菌褶中含有大量的担子,是有性繁殖的重要途径,因此也是细胞含量较丰富的组织,并且相对菌肉来说取材容易,是提取子实体基因组的首选位置。平菇菌皮由于紫外线的照射通常大部分呈灰色,含有较多的酚类物质。Katterman,F.R.H和 Shattuck,V.l. An[5]曾报道由于光照强度的影响,野生型菌比温室培养菌含有更高的单宁酸以及多酚复合物。因此提取菌褶时,会出现大量的蛋白杂质,从而很可能影响DNA的质量。菌柄由于较为严重的木质化、纤维化,细胞组织老化、变形,增加了提取的难度,因此只能获得较为有限的基因组DNA,而以这部分组织的基因组作为模板进行PCR扩增,如图3中A4所示,不能得到有效扩增。其次,老化组织比幼龄组织包含更多的多酚、萜类物质[6],这类物质通常为灰色,在研磨时从液泡中释放出来,不可避免地与DNA结合,在后续工作中很可能成为影响模板DNA质量的因素。因此,取材时应选用新鲜幼龄样品作为研究对象。
通过已有文献报道的平菇DNA提取实验方法,我们可以总结出以下几处共同点:首先,对样品进行前处理,如用液氮对组织研磨[7-8],对菌丝进行冻干处理,加入玻璃珠或其它具有破坏性的微小颗粒进行细胞研磨[9],加入裂解液进行剧烈振荡等[10],使用缓冲液对DNA进行抽提,并且对DNA进行纯化[11]。而这些方法的不同之处在于提取DNA所需时间,最终提取DNA的纯度、含量以及所用的抽提试剂等方面。针对众多的提取方法,我们尝试使用CTAB提取方法,加入β-巯基乙醇和PVP[7]等进行提取,得到基因组是粘性很强的灰褐色提取物,不能满足PCR等后续实验的模板DNA质量要求。
如何获得高质量、高产率的基因组DNA取决于细胞破碎是否彻底[12],而平菇细胞壁较厚,需要特殊的处理才能使基因组有效释放。在样品前处理时加入海砂,增加了样品之间的摩擦,使得液氮研磨的更加充分、快捷,从而缩短了样品在外界的暴露时间,降低了交叉污染率,也可有效抑制胞内酶对核酸的降解作用。
在裂解过程中,通过均质化使得样品充分混匀,减少了传统方法中水浴锅使用时进行振荡带来的不便。由于裂解液中加入高浓度的CTAB与SDS,可有效地与酸性多糖相结合形成复合物,并且蛋白酶K在SDS存在的情况下能有效地与蛋白结合,减少蛋白等抑制因子的存在,加入高盐成分,能有效地在抽提过程中减少多糖、多酚物质的污染。Sangwan等曾报道使用氯仿∶异戊醇(24∶1)代替酚仿抽提,原因在于后者获得的基因组质量较差,酚污染严重[13]。正如SDS-CTAB法使用氯仿∶异戊醇(24∶1)获得了较纯的基因组DNA,并且减少了饱和酚在PCR反应中的抑制作用,从而利用异丙醇沉淀基因组DNA。
但由于平菇属于高等真菌,细胞壁中主要以几丁质构成其骨架结构,与细菌、植物都有很大差异,对于传统的提取方法很难使得细胞内含物有效释放,因此,方法B在裂解前加入溶壁酶进行酶解,从而有效破碎细胞壁,使得内含物充分释放,获得高产率的基因组DNA。另外,高浓度的NaCl与醋酸钾能获得较高的产率,Jobes[14]等人就采用过这个方法。
综上所述,本文中SDS-CTAB法、高盐酶解法成功提取出质量较好、纯度较高的平菇子实体基因组DNA,两种方法相比,前者成本较低,后者产率较高,用这两类方法提取的平菇基因组DNA为菌种鉴定、基因分型等方面提供了可靠的保证,同时对于真菌基因组的提取起到很大辅助作用,也为今后进一步开展高等真菌分子生物学方面的研究奠定了基础。
[1]Kües U,Liu Y.Fruiting body production in basidiomycetes[J].Appl Microbiol Biotechnol,2000,54:141-152.
[2]Liu D,Sue C,Rob B,et al.Rapid mini-preparation of fungal DNA for PCR[J].Clinical Microbiology,2000,38(1):471.
[3]Michaels S D.Removal of polysaccharides from plant DNA by ethanol precipitation[J].Biotechniques,1994,17:274-276.
[4]Mendoza G,Delira A,Trejo M,et al.A rapid method for isolation of total DNA from pathogenic filamentous plant fungi[J].Genetics and Molecular Researc,2010,9(1):162-166.
[5]Katterman F R H,Shattuck V.An effective method of DNA isolation from the mature leaves of Gossypium species that contain large amounts of phenolic terpenoids and tannis[J].Preparative Biochemistry,1983,13:347-359.
[6]Porebski S L,Bailey G,Baum B R.Modification of CTAB DNA isolation methods for plants containing high polysaccharide and polyphenolcomponents[J].PlantMolecularBiology Reporter,1997,15:8-15.
[7]Bekesiova I,Nap J P,MLynarova L.Isolation of high quality DNA and RNA from leaves of the carnivorous plant Drosera rotundifolia[J].Plant Molecular Biology Reporter,1999,17: 269-277.
[8]Towner P.Prification of DNA//Brown T A[J].Essential Molecular Biology,1991:47-68.
[9]Murray M G,Thompson,W F.Rapid isolation of high molecular weight plant DNA[J].Nucleic Acids Research,1980,8:4321-4325.
[10]Huang J C,Ge X J,Sun M.Modified CTAB protocol using a silica matrix for isolation of plant genomic DNA[J].Bio Techniques,2000,28:432-434.
[11]Li Q B,Chai Q,Guy C L.DNA extraction method for RAPD analysis from plants rich in soluble polysaccharides[J].Plant Molecular Biology Report,1994,12:215-220.
[12]Jhingan A K.A novel technology for DNA isolation[J]. Methods Molecular Cell Biology,1992,3:15-22.
[13]Sangwan N S,Sangwan R S,Kumar S.Isolation of genomic DNA from defatted oil seed residue of opium poppy(Papaver sominiferum)[J].Plant Molecular Biology Reporter,2000,18:265 -270.
[14]Guillermaut P,Marchal D L.Isolation of plant DNA:A fast,inexpensive and reliable method[J].Plant Molecular Biology Reporter,1992,10:60-65.
Study on the methods of extraction of Pleurotus ostreatus genomic DNA
YAN Yan1,2,3,XU Wen-tao2,3,SU Chun-yuan2,LUO Yun-bo2,WANG Yi-nan3,GU Xin-xi1,DAI Yun-qing2,3,TIAN Hong-tao1,*
(1.Agricultural University of Hebei,College of Food Technology,Baoding 071001,China; 2.China Agricultural University,College of Food Science and Nutrition Engineering,Beijing 100083,China; 3.Supervision and Testing Center for GMOs Food Safety,Ministry of Agriculture,Beijing 100083,China)
Pleurotus ostreatus(culture by ourselves)genomic DNA were extracted by three methods,which were the methods of CTAB method,SDS-CTAB method,and high-concentration salt precipitation method,respectively,analyzed by ultraviolet spectrophotometer,digested by restriction enzymes of EcoRI and PCR amplified.The results proved Pleurotus ostreatus genomic DNA was really difficult to isolated genomic DNA by CTAB method,the ratio of OD260/280was only 1.5,digested by EcoRI inefficiency and PCR amplification useless.The other two methods were proved efficiently,the ratio of OD260/280was between 1.8~2.0 and had a clear fragment through agarose gel electrophoresis,digested by EcoRI efficiency and could be used as template for PCR amplification analysis,while the effect of extract genomic DNA by SDS-CTAB purification method and high-concentration salt enzymolysis method could be used for downstream analyses,the former was lower cost and the latter was more efficiency.
Pleurotus ostreatus;genomic DNA;extraction methods
TS255.1
A
1002-0306(2011)09-0190-04
平菇(Pleurotus ostreatus)又名糙皮侧耳,属于丝状真菌,是真菌植物门的子实体,它是一种高蛋白,富含多种维生素以及多糖成分的高级食品,在食品保健、医疗卫生、生物化工等方面起着很重要的作用[1]。针对平菇的这些应用,通常利用分子生物学手段进行研究,如基因的组成、基因的表达等。基因组提取是分子生物学的基础性研究,高效、稳定的基因组提取方法不仅可以减少工作量,并且能缩短鉴定、检测周期,给实验的顺利进行提供可靠的帮助。目前已报道的平菇基因组提取方法中较为普遍应用的是CTAB法(溴代十六烷基三甲胺),即在带有培养基的情况下对丝状菌丝提取基因组,而培养基中带有大量的糖、琼脂等物质给实验带来了很大的不便[2]。此外,平菇不同生长阶段,所产生的次级代谢产物不同[3],根据实验需要,要对平菇子实体DNA进行研究,但平菇这类高等真菌存在较厚的细胞壁或胶囊状物质,不利于裂解细胞释放DNA[4],并且由于大量的粘性多糖以及多酚类物质的存在,因此CTAB法很难得到高质量、高纯度的DNA分子。本研究以优化的基因组提取方法和传统方法进行比较,旨在探讨高效、稳定提取平菇子实体各部分基因组DNA的方法,从而为优化高等真菌基因组的提取方法提供了有力参考,同时也为今后进一步开展食用菌分子生物学研究以及检疫检测奠定了基础。
2010-12-08 *通讯联系人
闫燕(1986-),女,硕士研究生,研究方向:微生物酶资源开发。
国家高技术研究发展计划(863)项目(2006AA10Z440);国家自然基金 (30800770);转基因生物重大专项(2008ZX08012-001)。
