基因组改组技术选育苯基乳酸高产菌株
2011-10-25王立梅
王 丹,陈 光,王立梅
(1.吉林农业大学生命科学学院,吉林长春 130118;2.常熟理工学院生物与食品工程学院,江苏常熟215500;3.苏州市食品生物技术重点实验室,江苏苏州 215500)
基因组改组技术选育苯基乳酸高产菌株
王 丹1,陈 光1,王立梅2,3,*
(1.吉林农业大学生命科学学院,吉林长春 130118;2.常熟理工学院生物与食品工程学院,江苏常熟215500;3.苏州市食品生物技术重点实验室,江苏苏州 215500)
以Lactobacillus paracaseiW2为出发菌株,应用紫外诱变和亚硝基胍诱变筛选出5株突变菌株(U4、U7、U12、N3、N6),采用突变菌株进行多母本递推式原生质体融合,最终筛选出一株正向突变株F8,其发酵液中苯基乳酸含量为1.92g/L,比原始菌株提高了1.37倍。并且确定了最佳破壁条件为:溶菌酶浓度20g/L,变溶菌素浓度为15μg/mL;最佳融合条件为:常温下,聚乙二醇的浓度为50%,作用时间为10min。
基因组改组,副干酪乳杆菌,苯基乳酸
苯基乳酸(Phenyllactic Acid,PLA)作为一种新型的天然生物防腐剂具有较高的抑菌活性,能够抑制真菌以及金黄色葡萄球菌、单核细胞增生李斯特菌等多种细菌污染,从而延长食品贮藏期,保持食品原有风味[1-3]。目前苯基乳酸的制备方法主要有化学合成和生物合成[4-6]。通过微生物转化生产的苯基乳酸比传统化学防腐剂更加安全、无毒害、有益于身体健康,而倍受人们青睐[7]。但与此同时生物防腐剂也存在着抑菌浓度高、用量大、价格贵等不利因素[8],获得高产并且稳定的苯基乳酸生产菌株有利于降低其生产成本,为实现工业化生产提供理论依据。基因改组技术(genome shuffling)是在经典基因重组基础上发展的一种新的、更为有效的定向育种技术[9]。近几年来国内外很多学者已成功运用基因改组技术进行菌种改良。Patnaik等人对一株野生乳杆菌Lactobacillus(LB-WT)进行基因改组提高其耐酸性[10];Zhang应用基因改组技术有效地提高泰乐菌素产量4倍之多[11];林俊等人利用基因改组技术快速提高扩展青霉碱性脂肪酶产量,较出发菌株提高317%[12]。关于应用基因改组技术进行苯基乳酸生产菌株的菌种改造至今还未见报道。本实验旨在对苯基乳酸产生菌株进行基因改组以期获得苯乳酸高产菌株,为苯基乳酸的开发利用提供一条新的途径。
1 材料与方法
1.1 材料与设备
副干酪乳杆菌Lactobacillus paracaseiW2 本实验室筛选并在中国微生物菌种保藏管理委员会普通微生物中心保藏;D-苯乳酸标准品、溶菌酶、变溶菌素均购于Sigma公司;甘氨酸、聚乙二醇PEG6000、甲醇(色谱纯) 购于北京鼎国生物技术公司;其它试剂均为国产分析纯;MRS培养基(%) 蛋白胨1,肉浸膏1,酵母抽提物0.5,葡萄糖2,三水醋酸钠晶体0.5,Tween 80 0.1,柠檬酸三铵0.2,K2HPO40.2,MgSO4·7H2O 0.02,MnSO4·2H2O 0.005,琼脂1.5;改进CM培养基(%)[13]牛肉膏0.5,蛋白胨1,葡萄糖0.5,酵母膏0.5,NaCl 0.5,甘氨酸1.5,青霉素400U/mL,pH 7.2;再生培养基(%)[13]MRS培养基中添加MgCl20.43,蔗糖6.85,小牛血清0.5。
LC-20AT岛津高效液相色谱日本岛津公司;CR22GⅡ高速冷冻离心机日本东京日立KoKi株式会社。
1.2 实验方法
1.2.1 原生质体制备 将出发菌株培养至对数生长期后,接种于CM培养基中,分别培养2、4、6h,离心洗涤重悬于高渗溶液中,使用溶菌酶和变溶菌素共同破壁,在显微镜下观察原生质体形成情况,涂于再生平板。以破壁前原始菌株作对照,计算原生质体形成率和再生率[14]。
1.2.2 原生质体灭活
1.2.2.1 紫外灭活 将原生质体重悬于高渗溶液中,紫外灯下照射5、10、15、20、25s,各取1mL菌液涂平板,取灭活前原生质体溶液做对照,计算原生质体致死率。
1.2.2.2 热灭活 将原生质体溶液置于60℃水浴处理10、20、30、40、50min,计算原生质体致死率。
1.2.3 原生质体融合 将灭活的原生质体分别重悬于不同浓度的PEG6000中,室温作用5、10、15、20min,经高渗溶液离心洗涤涂布于再生培养基,37℃培养6d后,挑取融合平板上碳酸钙透明圈较大的菌落,通过摇瓶发酵筛选出苯基乳酸产量提高10%以上的菌株作为下一轮原生质体融合的出发菌株,进行多轮递推式原生质体融合。原生质体融合率计算参照文献[14]。
1.2.4 色谱条件 色谱柱:C18柱;流动相:A为0.05%三氟乙酸甲醇溶液,B为0.05%三氟乙酸水溶液;检测波长:210nm;柱温:30℃;总流速:1mL/min;进样量为5μL。
2 结果与分析
2.1 原生质体的形成与再生条件研究

图1 培养时间对原生质体形成及再生的影响
2.1.1 培养时间对原生质体形成的影响 由图1可以看出,处于不同生长时期的菌体细胞对溶菌酶的敏感性不同,2h和4h培养的菌体细胞代谢旺盛,细胞壁处于新生状态,有利于溶菌酶的渗透,易于原生质体的形成和再生,而6h的菌体细胞壁相对变厚,肽聚糖网结构更加致密,对溶菌酶抗性增强,此时的细胞破壁率和再生率都很低[14]。由于2h的菌体浓度低不利于细胞融合,故4h为最佳菌体培养时间。
2.1.2 酶浓度对原生质体形成的影响 如表1所示,单独使用溶菌酶不能完全破坏菌体细胞壁,原生质体无法释放出来。同时使用溶菌酶和变溶菌素处理,才能得到质壁分离的原生质体。随着酶浓度增加,原生质体的形成率也在提高,当使用最大溶菌酶浓度30g/L,变溶菌素浓度为15μg/mL时,原生质体形成率达到85%,但再生率只有26%。根据前人研究的结果,采用原生质体形成率与再生率乘积达到最大值时的酶浓度作为原生质体制备的最佳酶解浓度[15],确定溶菌酶浓度20g/L,变溶菌素浓度为15μg/mL。
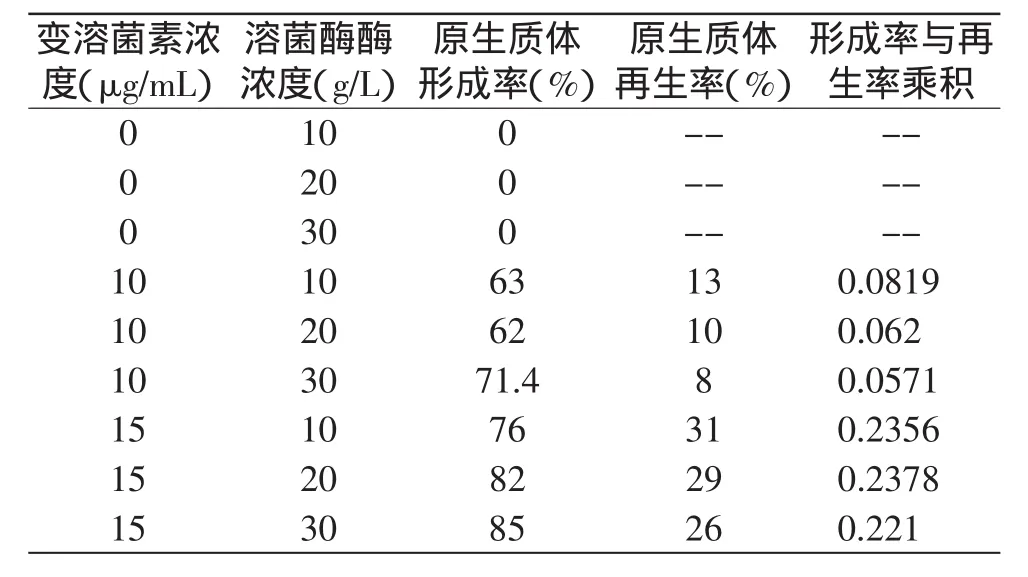
表1 溶菌酶和变溶菌素对原生质体形成和再生的影响
2.1.3 青霉素和甘氨酸对原生质体形成和再生的影响 青霉素和甘氨酸起到了破坏细胞壁中肽聚糖骨架的作用,增强了菌体细胞对溶菌酶的敏感性,易于形成原生质体[15]。从图2可知,对未经前处理的菌体细胞进行破壁原生质体形成率很低,只有20%。在培养基中分别添加甘氨酸和青霉素,原生质体形成率分别提高到57%和71%,同时加入甘氨酸和青霉素与单一添加甘氨酸或青霉素相比,细胞破壁效果更好,使原生质体再生率提高至43%。可见不同培养条件和生长环境对细菌的生长代谢和形态结构都会产生作用,青霉素和甘氨酸起到了破坏细胞壁中肽聚糖骨架的作用,增强了菌体细胞对溶菌酶的敏感性,易于形成原生质体[15]。

图2 青霉素和甘氨酸对原生质体形成及再生的影响
2.2 原生质体灭活条件研究
2.2.1 紫外灭活 如表2所示,原生质体在紫外线照射15s仍有存活,20s以后才能将原生质体全部灭活。
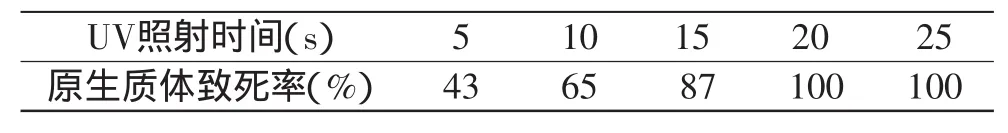
表2 紫外线照射时间与原生质体存活率的关系
2.2.2 热灭活 如表3所示,将原生质体置于60℃水浴10~20min仍有不同程度的原生质体存活,30min可将原生质体全部灭活。

表3 热灭活时间与原生质体致死率的关系
2.3 原生质体融合条件的研究
2.3.1 PEG浓度对原生质体融合率的影响 如图3所示,PEG浓度在30%~50%之间时原生质体融合率随着浓度增加而上升,当PEG浓度达到50%时融合率达到了0.076%,PEG可使细胞之间接触点处的膜脂类分子发生侧向流动而相互亲和,增加了细胞发生融合的可能[16]。继续提高PEG浓度至60%,融合率骤然下降,因此采用50%的PEG室温下进行原生质体融合较为适合。

图3 PEG浓度对原生质体融合的影响
2.3.2 融合时间对原生质体融合率的影响 在细菌原生质体融合时,PEG作用于原生质体的时间也至关重要。由图4可以看出,随着融合时间的延长,融合率降低,由于延长PEG作用时间使原生质体破裂死亡,导致再生率下降[17]。融合时间5min,融合率达到了0.089%,说明原生质体融合是在PEG接触原生质体后立即发生的,但是要达到基因重组必须维持一定作用时间,故选用10min为原生质体融合的最佳时间。

图4 融合时间对原生质体融合率的影响
2.4 原始菌株与突变株苯基乳酸产量的比较
将原始菌株与突变株分别进行摇瓶发酵,实验结果见图5,紫外诱变和亚硝基胍诱变获得的突变株苯基乳酸产量仅提高10%~20%,与原始菌株差别不大。原生质体融合筛选出四株菌F5、F7、F8、F13,经过多轮递推式原生质体融合其苯基乳酸产量均提高了1~2倍,最高产量达到了1.92g/L,比原生菌株提到了1.37倍。

图5 原始菌株与突变菌株苯基乳酸产量比较
3 结论
3.1 采用含有1.5%甘氨酸和400U/mL青霉素的MRS培养基培养获得菌体细胞,确定最适培养时间为4h。
3.2 采用溶菌酶和变溶菌素对副干酪乳杆菌进行破壁,当溶菌酶浓度为20g/L,变溶菌素浓度为15μg/mL时,原生质体形成率达到80%以上。
3.3 将破壁得到的原生质体进行融合,最佳融合条件为室温下PEG6000浓度为50%,作用时间为10min,在该条件下原生质体融合率达到0.089%。多轮融合后最终筛选出一株苯基乳酸高产菌株,其发酵液中苯基乳酸产量为1.92g/L,比出发菌株提高了1.37倍。
[1]Dieulevenx,Lemarinier S,GuegUen M.Antimicrobial spectrum and target site of D-3-Phenyllactic acid[J].International Journal of Food Microbiology,1998,40(3):177-183.
[2]Dieulevenx V,Van Der Pyl D,Chataud J,et al.Purification and characterization of anti-Listeria compounds produced by Geot richum candidum[J].Applied and Environmental Microbiology,1998,64(2):800-803.
[3]STROM K,SJOGREN J,BROBERG A,et al.Lactobacillus plantarum MiLAB 393 produces the antifungalcyclic dipeptides cyclo (L-Phe-L-Pro) and cyclo (L-Phe-trans-4-OH-L-Pro) and 3-phenyllactic acid[J].Applied and Environmental Microbiology,2002,68(9):322-327.
[4]Kazuaki N,Mamoru.Method for produeing of tieally aetive phenyllacti aeid[P].JP2003192633.
[5]邓喜玲,陈学敏,周淑芳,等.苯基乳酸的合成[J].西北药学杂志,2001,16(1):36-37.
[6]Valerio F,Lavermicocca P,Pascale M,et al.Production of phenyllactic acid by Lactic acid bacteria:an approach to the selection of strains contributing to food quality and preservation[J].International Journal of Food Microbiol Lett,2006,233:289-295.
[7]Lavermicocca P,Valeriof.Antifungal activity of phenyllactic acid against molds isolated from bakery products[J].Applied and Environmental Microbiology ,2003,69(1):634-640.
[8]Chifiriuc MC,Veronica L,Dracea O,et al.Drastic attenuation of Pseudomonas aeruginosa Pathogenicity in a holoxenic mouse experimental model induced by subinhibitory concentrations of phenyllaetie aeid (PLA)[J].International Journal of Moleeular Seienees,2007,8(7):583-592.
[9]HopwoodDA,WrightHM,BillML,etal.Geneticrecombination through protoplast fusion in streptomyces[J].Nature,1997,268:171-174.
[10]Patnaik R,Louie S,Gavrilovic V,et al.Genome shuffling of Lactobacillus for improved acid tolerance[J].Nat Biotechnol,2002,20:707-712.
[11]Zhang Y X,Kim P,Victor A,et al.Genome shuffling leads to rapid phenotypic improvement in bacteria[J].Nature,2002,15:644-646.
[12]林俊,施碧红,施巧琴,等.基因组改组技术快速提高扩展青霉碱性脂肪酶产量[J].生物工程学报,2007,23(4):672-677.
[13]施巧琴,吴松刚.工业微生物育种学[M].北京:科学出版社,2003:308.
[14]OsamuTanaka,Sadahiro Ohmomo.Efficient protoplast regenerationforsomehomofermentativelactobacilliandpediococci[J].Applied and Environmental Microbiology,2001,177:36-40.
[15]于雷,刘景圣.应用基因组改组选育耐糖L-乳酸高产菌株[J].食品科学,2007,28(9):369-373.
[16]CONNELL H,LEMMON J,TANNOCK G W.Formation and regeneration of protoplasts and spheroplasts of gastrointestinal strainsofLactobacilli[J].AppliedandEnvironmentalMicrobiology,1988,54:1615-1618.
[17]LEEWICKNERLJ,CHASSYBM.Productionandregeneration of Lactobacillus casei protoplasts[J].Applied and Environmental Microbiology,1984,48:994-100.
Breeding of strain highly producing phenyllactic acid by genome shuffling
WANG Dan1,CHEN Guang1,WANG Li-mei2,3,*
(1.College of Food Engineering,Jilin Agricultural University,Changchun 130118,China;2.Department of Biotechnology and Food Engineering,Changshu Institute of Technology,Changshu 215500,China;3.The Key Laboratory of Food and Biotechnology,Suzhou 215500,China)
The genome shuffing was used to improve the production of phenyllactic acid by Lactobacillus paracasei W2.The mutants (U4,U7,U12,N3,N6)obtained by ultraviolet irradiation and nitrosoguanidine were used to the parental strains of protoplast fusion.After several rounds of genome shuffling,a forward mutant strain F8 was screened,its phenyllactic acid production was 1.92g/L,which increased 1.37 times more than the parental strain.The optimum concentration of mutanolysin and lysozyme were 15μg/mL and 20g/L,respectively.The optimum conditions of protoplast fusion were 50%PEG6000 and 10min at room temperature.
genome shuffling; Lactobacillus paracasei; phenyllactic acid
TS201.3
A
1002-0306(2011)10-0208-04
2010-10-11 * 通讯联系人
王丹(1984-),女,硕士研究生,研究方向:酶工程。
江苏省科学技术厅(BE2008391);常熟市科技局(CN200921)。
