1株近平滑假丝酵母的分离及其鉴定
2011-09-25潘欣
潘欣
(成都理工大学旅游与城乡规划学院,四川成都610059)
1株近平滑假丝酵母的分离及其鉴定
潘欣
(成都理工大学旅游与城乡规划学院,四川成都610059)
采用平板稀释法从土壤及水果中分离出1株近平滑假丝酵母(Candida parapsilosis),YPD培养基培养酵母,利用分子生物学方法对其rRNA基因内转录间区(ITS区)进行了克隆测序,并与GenBank中已有的有关序列进行比较及系统发育分析。测序结果表明该序列长度为547 bp,与GenBank中近平滑假丝酵母同源率在98.5%~100%之间,进化分析表明与C.parapsilosis(EF193067)、C.parapsilosis UOA/HCPF(FJ872013)属于一单独分支中,形态结果及分子鉴定表明近平滑假丝酵母培养成功,为其进一步开发利用提供了依据。
近平滑假丝酵母;分离;鉴定;进化树
近平滑假丝酵母(Candida parapsilosis),属假丝酵母属,可产生多种代谢产物,如还原型谷胱甘肽(GSH)[1]、氧化还原酶[2]及木糖还原酶[3]等,是具有催化[4]、消旋转化能力的菌株[5],也可作为发酵剂用于肉制品发酵[6],因此具有很好的发展前途。目前,国内外对近平滑假丝酵母的研究不多,主要集中在其产生还原酶等生物催化剂方面,对其分子鉴定、进化关系和系统发育研究较少。鉴于此,本研究从土壤及水果中分离得到1株近平滑假丝酵母,并对其培养特性、细胞特征进行了描述,提取了总DNA,结合分子生物学方法对近平滑假丝酵母进行鉴定,并作了系统发育分析,以期为近平滑假丝酵母的进一步开发利用提供理论依据。
1 材料与方法
1.1 材料
1.1.1 菌种近平滑假丝酵母纯化菌种CP1215,分离自成都理工大学校园土壤。
1.1.2 试剂Taq DNA聚合酶、dNTP、pBS-TⅡ快速连接试剂盒,天根生化科技(北京)有限公司;IPTG、X-gal、氨苄青霉素,上海生物工程有限责任公司;分子量标准DL 2000,宝生物工程(大连)有限公司。
1.1.3培养基YPD培养基:蛋白胨2%,酵母抽提物1%,葡萄糖2%,pH自然,制成斜面或平板,加2%琼脂粉制成YPD固体培养基,121℃灭菌20 min备用,冷却至50℃左右加氨苄西林至终浓度100 μg/mL;LB培养基:蛋白胨1%,酵母抽提物0.5%,NaCl 1%,pH 7.0,加2%的琼脂制成斜面或平板,121℃灭菌20 min备用,冷却至50℃左右加氨苄西林至终浓度100 μg/mL。
1.2 方法
1.2.1 近平滑假丝酵母分离与纯化土壤(10份)取自成都理工大学校园,将新鲜土壤样品5 g放入100 mL无菌三角瓶内,加入无菌水及玻璃珠,制成10-1~10-8的土壤悬液。用微量移液器吸取0.5 mL土壤稀释液于YPD培养皿中,将接种物均匀涂布,于28℃恒温箱倒置培养5~7 d。水果(10份)购自成都理工大学菜市场,切成0.5 cm×0.5 cm小块,接入YPD培养皿。待长出菌落后,在菌落边缘用无菌接种环挑取菌落,移入新YPD培养皿中,继续于28℃恒温条件下培养,待长出菌落后显微镜观察显微特征。
1.2.2 近平滑假丝酵母培养收集将纯化后的近平滑假丝酵母接种于YPD液体培养基,28℃,180 r/min振荡培养3~5 d,10000 r/min离心20 min收集细胞,用吸水纸尽量吸干水分,4℃保存备用。
1.2.3 DNA的提取将酵母细胞用蒸馏水洗涤2次后,用等体积玻璃珠裂解液重悬,涡旋混匀15 min,5%蜗牛酶37℃摇床孵育3 h,1%SDS、100 μg/mL蛋白酶K,37℃水浴30 min,酚-氯仿抽提2次取上清,加入2倍体积75%乙醇沉淀DNA,100%乙醇洗涤,晾干后用超纯水溶解DNA,提取的DNA以0.8%琼脂糖凝胶电泳检测,-20℃冰箱保存备用。
1.2.4 ITS区PCR扩增及克隆采用真菌通用引物ITS4和ITS5,由上海生工生物工程有限公司提供,其序列如下:ITS4(5'-TCCTCCGCTTATTGATATGC)和ITS5(5'-TTGGAAGTAAAAGTCGTAACAAG)。PCR扩增体系50 μL:超纯水41 μL,10×Buffer(含1.5 mmol/L Mg2+)5.0 μL,10 mmol/L dNTP 1.0 μL,引物各1.0 μL,模板DNA 0.5 μL,Taq DNA聚合酶0.5 μL。反应参数:94℃预变性5 min;94℃变性1 min,50℃退火1 min,72℃延伸1 min,循环30次;72℃延伸10 min。扩增产物经胶回收,按pBS-TⅡ快速连接试剂盒步骤转化,将阳性转化子穿刺LB培养基培养后,送上海生工生物工程技术服务有限公司测序。
1.2.5 ITS区序列的分析利用Cluster W软件对ITS序列进行整理,DNAstar及Blast(http:// blast.ncbi.nlm.nih.gov/Blast.cgi)进行同源性及聚类分析。
2 结果与分析
2.1 近平滑假丝酵母的培养特性
28℃下YPD培养基上,菌株CP1215产生的菌落粘稠、污白色,显微镜下为单细胞个体,呈球形、椭圆形等,未见芽殖及假丝菌(图1)。
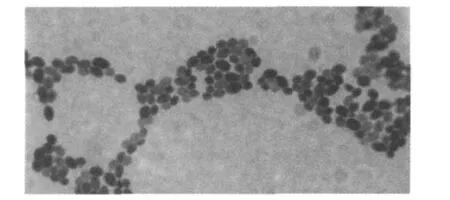
图1 近平滑假丝酵母细胞形态(100×)Fig.1The morphology of C.parapsilosis cells(100×)
2.2 近平滑假丝酵母ITS区序列
电泳结果显示,近平滑假丝酵母CP1215 ITS区长度约550 bp左右(图2),测序结果表明该序列长度为547 bp(图3),GC%=40.04%,而Gen-Bank中该序列长度在544~548 bp之间,GC%含量在40.11%~40.44%之间。近平滑假丝酵母ITS区与GenBank中登录的已知菌株C.parapsilosis wb176(AF455530),C.parapsilosisMCCC2E00272(EF193067),C.parapsilosisIFM 52623(AB109289),C.parapsilosisHK31d (EF198017),C.parapsilosisUOAHCPF (FJ872013),C.parapsilosis IFM 2465(AB109235)及C.parapsilosis IFM 2466(AB109292)同源率在98.5%~100%之间,证明本实验成功培养出近平滑假丝酵母。
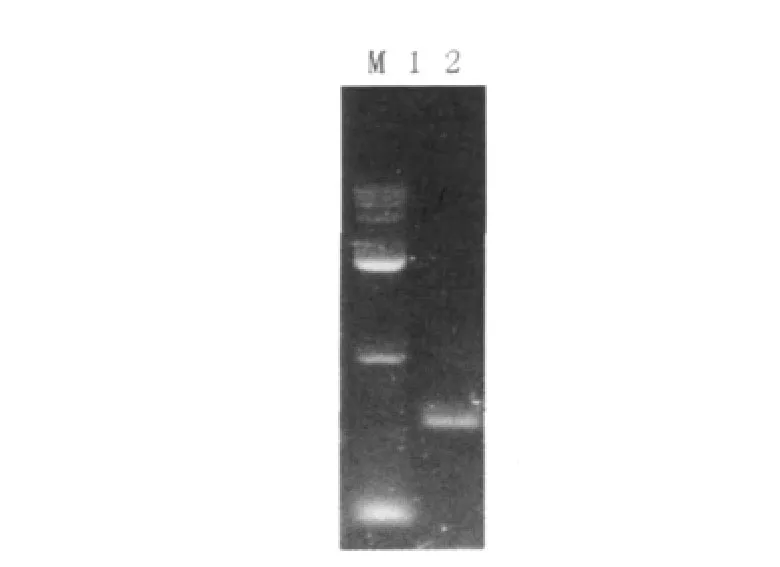
图2 ITS区PCR电泳Fig.2Electrophoresis of ITS PCR product
2.3 近平滑假丝酵母CP1215 ITS区系统发育分析

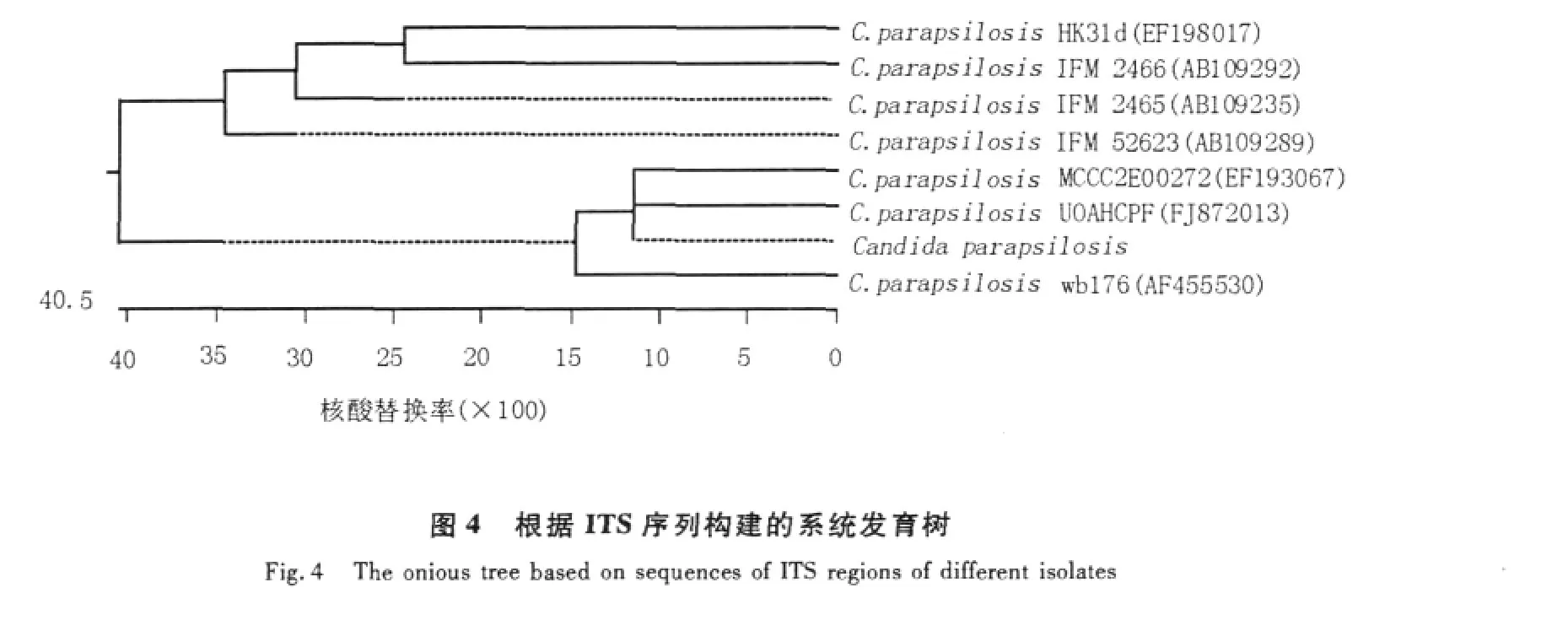
选用DNA纯度符合要求的总DNA作为模板扩增其ITS区,回收PCR产物作连接、转化及鉴定后测序,选取C.parapsilosiswb176 (AF455530)、C.parapsilosisMCCC2E00272 (EF193067)、C.parapsilosisIFM52623 (AB109289)、C.parapsilosisHK31d (EF198017)、C.parapsilosisUOA/HCPF (FJ872013)、C.parapsilosisIFM2465 (AB109235)及C.parapsilosisIFM2466 (AB109292)等作分析比较。将所得出的近平滑假丝酵母的ITS区序列分别与GenBank中已有的序列进行比较,利用分子生物学软件分析构建了系统发育树(图4)。从系统发育树可以看到,本试验所得菌株与GenBank中近平滑假丝酵母同源率在98.5%~100%之间。
3 讨论
艾丽静[1]曾用18S rDNA鉴定产还原型谷胱甘肽酵母菌,其与GenBank中菌株同源性为99.75%,本研究表明其与C.parapsilosis (EF193067)、C.parapsilosisUOA/HCPF (FJ872013)属于一单独分支中,而此进化关系与其代谢产物之间是否存在联系,尚需下一步研究分析。已有研究表明近平滑假丝酵母是合成手性化合物的重要生物催化剂来源菌株[7]。本研究结果证明ITS区序列分析可以作为假丝酵母类真菌的新的鉴定方法,与经典形态学方法结合,为近平滑假丝酵母的分类鉴定及进一步开发利用提供依据。
[1]艾丽静,饶志明,沈微,等.产还原型谷胱甘肽酵母菌的筛选与初步鉴定[J].食品与发酵工业,2006,32(3):18-21.
[2]张荣珍.近平滑假丝酵母立体选择性还原酶表达、结构解析与改造[D].江南大学博士论文,2009.
[3]曲有鹏.近平滑假丝酵母木糖还原酶基因的克隆及表达研究[D].哈尔滨工业大学硕士论文,2007.
[4]娄文勇,郭强,郁惠蕾,等.近平滑假丝酵母细胞催化乙酰基三甲基硅烷不对称还原反应[J].催化学报,2009,30(12): 1276-1280.
[5]任艳秋,徐岩,穆晓清,等.醇类物质诱导培养改善近平滑假丝酵母去消旋转化能力[J].催化学报,2005,5(5):554-557.
[6]赵俊仁,孔保华.自然发酵风干肠中酵母菌生产性能的研究[J].食品科技,2010,35(10):27-31.
[7]羊明,徐岩,穆晓清,等.近平滑假丝酵母NAD(H)依赖型次级醇脱氢酶的分离纯化及酶学性质[J].应用与环境生物学报,2007,13(1): 121-125.
Isolation,Purification and Identification of a Candida parapsilosis strain
PAN Xin
(Chengdu Uni.of Technol.,Chengdu 610059)
A Candida parapsilosis(Cp)strain was isolated using YPD medium plate dilution method from soil and fruits,identified by molecular biological method with rRNA gene cloning and sequencing in its ITS transcription domain;and made the comparison with relevant sequence available in Genbank and phylogenetic analysis.The results of the sequencing showed that homologous rate of the Cp strain was 98.5%~100%with the one in Genbank.Evolution analysis showed that it belonged to independent branch of Cp(EF193067)and Cp UOA/HCPF(FJ872013).Its morphology and molecular identification indicated that the Cp strain was successfully cultivated and this provided a foundation for its further development and utilization.
Candida parapsilosis;isolation;identification;phylogenetic tree
Q939.5,Q812
A
1005-7021(2011)01-0043-04
潘欣女,博士,讲师。主要从事真菌及生态学的研究。E-mail:zoulk124@163.com
2011-01-04;修回日期:2011-01-23
