突变型聚酮合成酶酮还原酶域的表达及序列分析
2011-07-26李凌凌吕早生
李凌凌,吕早生,李 涛,沈 辉
(武汉科技大学化学工程与技术学院,湖北 武汉 430081)
聚酮化合物,是广泛存在于自然界中的结构多样的次生代谢产物,具有很大的医药应用价值,包括很多抗生素和抗真菌药。聚酮是由聚酮合成酶(Polyketide synthase,PKS)催化一系列的小分子前体进行重复缩合反应而合成的。目前为止,至少有3种不同类型的聚酮合成酶[1],其中Ⅰ型聚酮合成酶是一种模块型的多功能酶,如负责催化合成红霉素A中大环内酯环的6-脱氧红霉内酯B合成酶(6-Deoxyerythronolide B synthase,DEBS),DEBS有6套结构和功能相近的模块,每套模块催化完成一轮碳链的延伸和还原,其上有酮合成酶(Ketosynthase,KS)、酰基转移酶(Acyl transferase,AT)和酰基载体蛋白(Acyl carrier protein,AC)3个基本酶域,且可能含有酮还原酶(Ketoreductase,KR)、脱水酶(Dehydratase,DH)和烯醇还原酶(Enoyl reductase,ER)[2]。Ⅱ型聚酮合成酶是一种可重复催化反应的多酶复合体,负责合成芳香族聚酮,如放线菌素。聚酮合成酶中的酮还原酶属于短链脱氢酶(Short-chain dehydrogenase/reductase,SDR)超家族。2004年,Korman等[3]和Hadfield等[4]通过比较芳香族聚酮的酮还原酶与其它短链脱氢酶超家族成员的结构差异,提出芳香族聚酮的酮还原酶(如放线菌素聚酮还原酶ActKR)与其它SDR家族成员的差异为α6和α7间的10个氨基酸残基,该区域是SDR家族的底物结合口袋的一部分,是最不保守的序列,负责控制底物特异性。2006年,Keatinge-Clay等[5]研究了糖多孢红霉菌聚酮合成酶模块1的酮还原酶域EryKR1的结构,提出了该酶域与其它酶域间的界限、多聚体的组成方式以及控制催化立体选择性还原反应的机制等,但未提及该酶域的底物结合口袋等。同年Bali等[6]在大肠杆菌中异源表达了糖多孢红霉菌聚酮合成酶的酮还原酶域,并研究了该酶域的底物特异性,结果发现此酶域对结构中含有环己酮的底物都有活性,但该研究并没有以这些底物进行生物催化,得出有关转化率和产率的数据。
作者在此通过比对EryKR1酶域和ActKR酶的氨基酸序列和二维结构,根据ActKR中控制底物特异性位点为α6和α7间的10个氨基酸残基EHYSDIWEVS,推测EryKR1酶域中控制底物特异性位点的氨基酸残基,接着利用重叠PCR技术,扩增出推测的控制底物特异性位点的氨基酸序列,替换成ActKR对应位点的EHYSDIWEVS的突变型EryKR1酶域基因eryKR1M,将此DNA片段克隆到pET-28a上,构建了重组质粒pET-eryKR1M,并转化到大肠杆菌BL21中,且研究由此获得的重组菌株与野生型重组菌E.coliBL21(pET-eryKR1)2在针对环己酮、2-辛酮、苯乙酮和4-氯乙酰乙酸乙酯进行生物催化羰基还原上的区别,旨在研究EryKR1酶域中α6和α7间的氨基酸残基替换对上述底物催化特异性的改变情况,进而分析探讨EryKR1酶域的底物特异性位点。
1 实验
1.1 材料
1.1.1 菌种和质粒
糖多孢红霉菌A226(SaccharopolysporaerythraeaA226)、大肠杆菌BL21、DH5α及表达载体pET-28a由安徽大学张部昌教授惠赠;E.coliBL21 (pET-gdh1)菌株(即异源表达枯草芽孢杆菌葡萄糖脱氢酶基因gdh的大肠杆菌重组菌)以及E.coliBL21 (pET-eryKR1)2菌株(即异源表达野生型EryKR1酶域基因eryKR1的大肠杆菌重组菌)均为自行构建保藏。
1.1.2 培养基
LB培养基按照文献[7]配制,筛选抗性菌株时,加终浓度100 μg·mL-1的卡拉霉素;TSB培养基(%):Tryptic Soy Broth 3 g,用于S.erythraea的液体培养[8]。
1.1.3 工具酶和化学试剂
限制性核酸内切酶、蛋白酶K、RNase酶、T4DNA连接酶、IPTG,Takara公司;DNA纯化和质粒少量快速提取试剂盒,北京道普生物技术开发中心;dNTP、pfu DNA聚合酶,北京天为时代生物技术有限公司;引物合成和测序工作由上海生工公司完成;Tryptic Soy Broth,Fulka;4-氯乙酰乙酸乙酯(97%),Alfa Aesar;R-4-氯-3-羟基丁酸乙酯(98%),AcRos;其它试剂均为国产。
1.2 方法
1.2.1eryKR1M基因的克隆及表达质粒的构建
糖多孢红霉菌A226基因组DNA的提取参照文献[8]进行。根据文献[5]报道的EryKR1酶域的边界,设计扩增突变型eryKR1M基因的两端引物,为便于克隆,在正向引物和反向引物前分别加上NdeⅠ和BamHⅠ酶切位点,引物LKR:5′-gccatatggacgaggtttccgcgctgcg-3′(gc为保护性碱基,catatg为NdeⅠ酶切位点),引物RKR:5′-gcggatcctcacgcgcccacccgcggttcggc-3′(gc为保护性碱基,ggatcc为BamHⅠ酶切位点);用于扩增中间突变位点的重叠PCR引物:引物ActKRL:5′-gagcactactcggacatctgggaggtgtcgcccgagac-ggcctgccgggc-3′,引物ActKRR:5′-cgacacctcccagatgtcggagtgctcgcggaagcggtcggccaccg-3′;PCR扩增体系和条件见文献[9]。获得的PCR特异片段产物回收纯化后,经上海生工测序部测序,再利用NCBI的Nucleotide blast(http://blast.ncbi.nlm.nih.gov/Blast.cgi)[10]进行比对,以确认是否为对应位点发生所需突变的目标基因。PCR扩增产物、载体的双酶切及连接、大肠杆菌转化等分子操作,参照文献[7]按常规方法进行;PCR产物及双酶切产物的回收纯化和质粒小量提取按照产品说明书进行。
1.2.2 酶的诱导表达和SDS-PAGE分析
将E.coliBL21(pET-eryKR1M)重组菌按1%的接种量接入到含有100 μg·mL-1卡拉霉素的LB培养基中,37 ℃培养至A600=1.0,加入终浓度为1 mmol·L-1的IPTG,37 ℃诱导表达4 h后,4000 r·min-1、4 ℃离心10 min,收集菌体,将收集的菌体进行SDS-PAGE分析,以检测目的蛋白的表达量[11]。
1.2.3 重组菌催化还原羰基底物的反应
双重组菌耦合还原4种羰基底物(环己酮、2-辛酮、苯乙酮和4-氯乙酰乙酸乙酯)的反应体系参照文献[12]。上述转化体系于30 ℃、100 r·min-1振荡反应6 h后,6000 r·min-1离心10 min,收集上清。气相色谱检验乙酸乙酯萃取转化液上清获得的有机相,用于分析产物得率。分析环己酮、苯乙酮、4-氯乙酰乙酸乙酯及2-辛酮转化液的气相色谱条件参照文献[12]。还原反应的产率按文献[11]计算。
1.2.4 序列分析
采用DNAssist软件进行蛋白质的氨基酸序列比对,采用PSIPRED Server进行蛋白质的二维结构预测(http://bioinf.cs.ucl.ac.uk/psipred/),采用Swiss-model进行蛋白质三维结构预测(http://swissmodel.expasy.org/),以了解突变对酶的影响。
2 结果与讨论
2.1 对EryKR1酶域的序列分析
根据Hadfield等的报道,放线菌素聚酮还原酶ActKR的底物结合口袋是由β4、β5和β6折叠的羧基端和α5的氨基端以及α6-环-α7形成,且α6和α7之间的10个氨基酸残基是芳香族聚酮还原酶(如ActKR)和其它SDR家族成员最大的差异,决定了SDR家族成员的不同的底物特异性[4]。因此利用DNAssist软件和PSIPRED Server对EryKR1酶域和放线菌素聚酮还原酶ActKR分别进行氨基酸序列比对(如图1所示)和二级结构预测(如图2所示),发现EryKR1酶域的二级结构(图2b)基本符合ActKR酶的二级结构(图2a)的模式特点,推测EryKR1的底物结合口袋也由α6-环-α7组成,且其中的氨基酸残基RHGVIEMP对应于放线菌素聚酮还原酶的α6和α7之间的10个氨基酸残基EHYSDIWEVS,可能决定了EryKR1酶域和ActKR酶的不同的底物特异性。

框中为α6-环-α7间的氨基酸残基,黑色标记是相同残基,灰色标记是相似残基
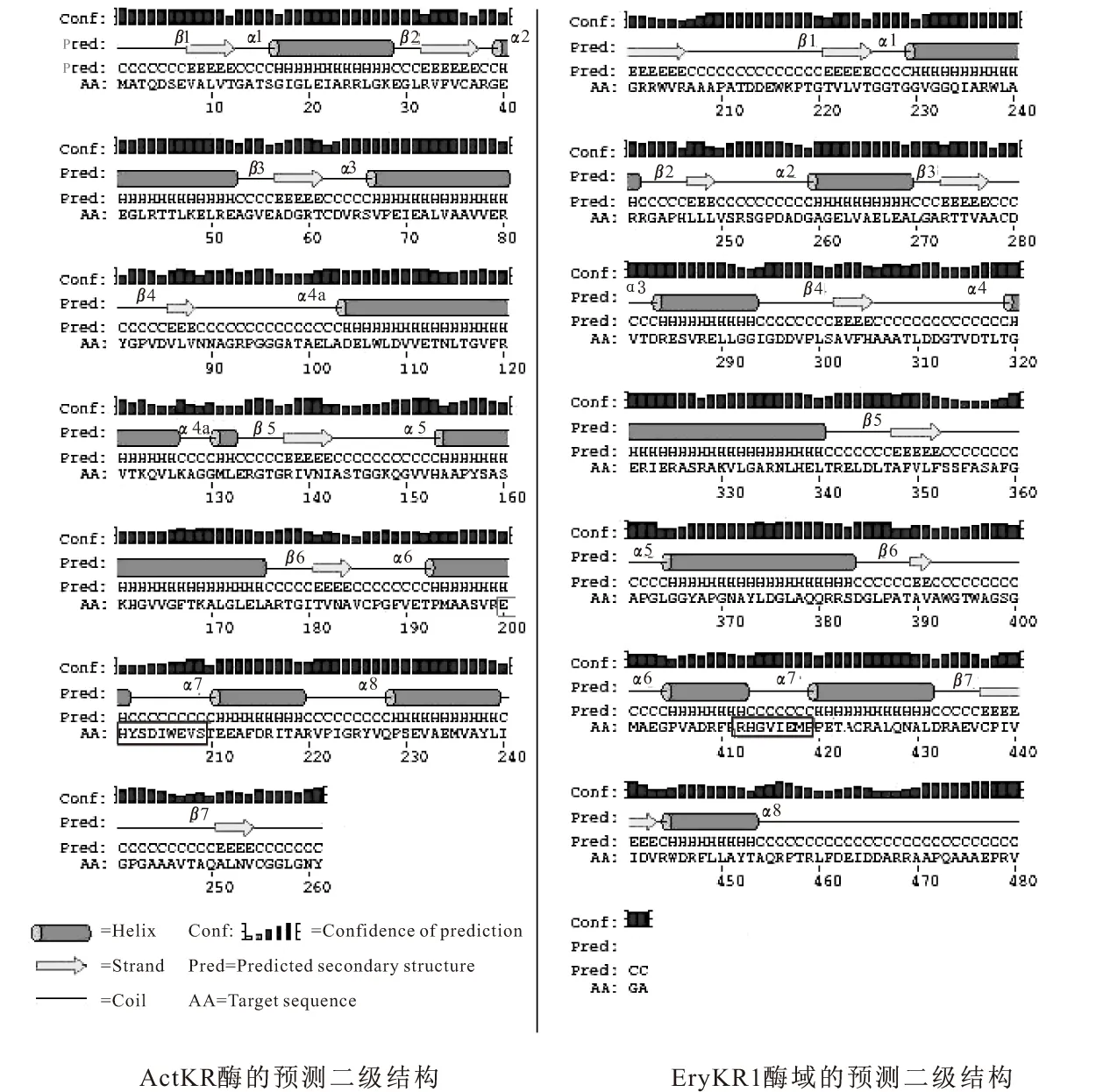
图2 PSIPRED Server进行的二维结构预测(方框中为α6-环-α7间的氨基酸残基)
2.2 pET-eryKR1M重组质粒的构建及鉴定
以糖多孢红霉菌基因组DNA为模板,以引物LKR和ActKRR进行PCR扩增,获得PCR产物eryKR1M-L,再以引物RKR和ActKRL进行PCR扩增,获得PCR产物eryKR1M-R,将这些PCR产物分别进行1.0%和2.5%琼脂糖凝胶电泳,结果如图3所示。
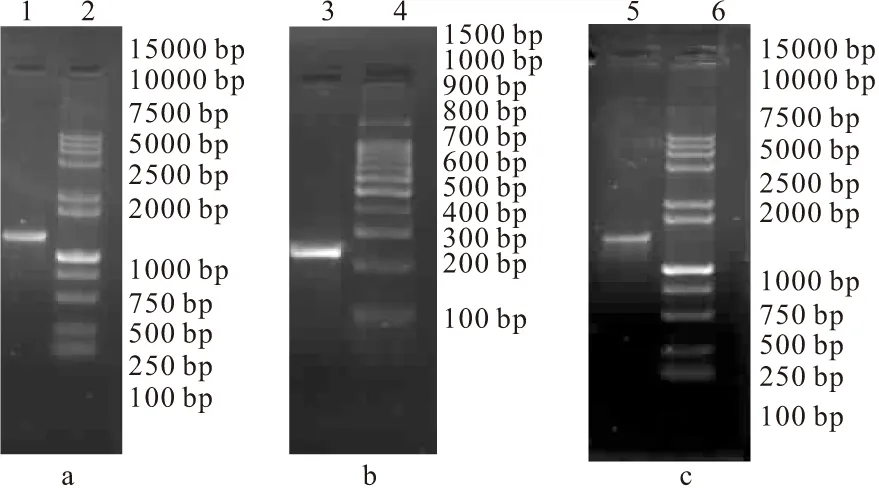
1、3、5分别为eryKR1M-L、eryKR1M-R、eryKR1M的PCR回收纯化产物 2、4、6为DNA分子质量标准
由图3a、3b可见,PCR产物eryKR1M-L位于1000~2000 bp之间,与目标基因突变位点左侧的序列长度(1250 bp)吻合,而片段eryKR1M-R位于200~300 bp之间,与目标基因突变位点右侧的序列长度(210 bp)吻合。以片段eryKR1M-L和eryKR1M-R(体积比为3∶1)为模板,再以引物LKR和RKR进行扩增,利用重叠PCR获得产物eryKR1M,以1.0%琼脂糖凝胶进行电泳(图3c),片段大小位于1000~2000 bp之间,与目标基因的序列长度(1450 bp)吻合。将eryKR1M片段克隆到pET-28a载体上,构建了重组质粒pET-eryKR1M。对该质粒进行双酶切鉴定(图4),证实了质粒pET-eryKR1M中克隆的基因片段与目标片段的大小一致。
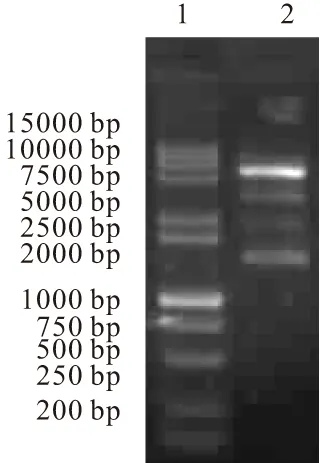
1.DNA分子质量标准 2.pET-eryKR1M重组质粒经NdeⅠ和Bam HⅠ双酶切产物
利用NCBI提供的核苷酸比对功能,对重组质粒的测序结果进行序列分析,得出测序结果与登录号为AY661566.1报道的糖多孢红霉菌聚酮合成酶基因簇(总长为32 299 bp)中4384~5756 bp之间的碱基序列的相似度达到99%,其中AY661566.1序列中的第5572~5595个碱基aggcacggcgtcatcgagatgcct(编码的氨基酸序列为RHGVIEMP)已替换成测序结果中的gagcactactcggacatctgggaggtgtcg(编码的氨基酸序列为EHYSDIWEVS),如图5所示。说明野生型eryKR1基因中编码α6-环-α7之间的氨基酸残基RHGVIEMP的碱基序列已突变成了放线菌素聚酮还原酶对应位点EHYSDIWEVS相应的碱基序列。

图5 pET-eryKR1M测序结果的核苷酸比对分析(方框标记的是突变位点)
2.3 突变型eryKR1M基因的诱导表达
表达载体pET-eryKR1M转化到E.coliBL21(DE3)后,获得了E.coliBL21 (pET-eryKR1M)重组菌,以E.coliBL21 (pET-28a)菌为阴性对照,经IPTG诱导后,进行SDS-PAGE分析,结果见图6。
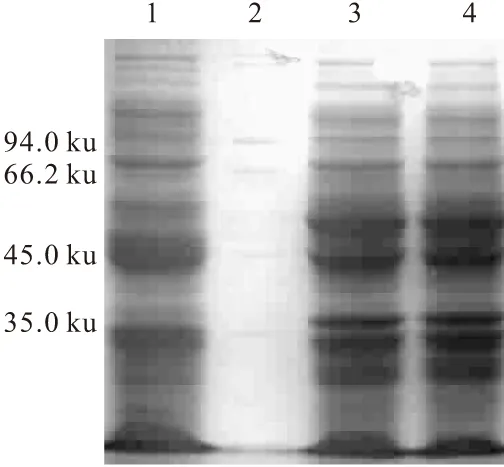
1、3、4分别为IPTG诱导的E.coli BL21(pET-28a)、E.coli BL21(pET-eryKR1)2、E.coli BL21(pET-eryKR1M)的全蛋白 2.蛋白质分子量标准
由图6可知,经诱导的E.coliBL21(pET-eryKR1M)中表达的蛋白质条带和阴性对照的相比,存在明显的差异,重组菌在45.0~62.0 ku之间有明显的突变的酮还原酶域表达条带(相对分子量为57 ku,包括了pET-28a载体表达的His tag片段)。利用BandScan 5.0软件分析目标蛋白的SDS-PAGE扫描图片,其中E.coliBL21 (pET-eryKR1M)重组菌中目标蛋白质的表达量占全菌可溶性蛋白质的5.7%,与异源表达野生型eryKR1基因的E.coliBL21(pET-eryKR1)2中EryKR1酶域的表达量5.89%基本一致。
2.4 E.coli BL21(pET-eryKR1M)催化还原4种羰基底物
E.coliBL21(pET-eryKR1M)和E.coliBL21 (pET-eryKR1)2重组菌以环己酮、2-辛酮、4-氯乙酰乙酸乙酯和苯乙酮分别为底物进行转化,以E.coliBL21(pET-28a)针对这些底物的转化液作为阴性对照进行气相色谱检测,结果见表1。野生型和突变型重组菌以环己酮为底物的转化液的气相色谱见图7。
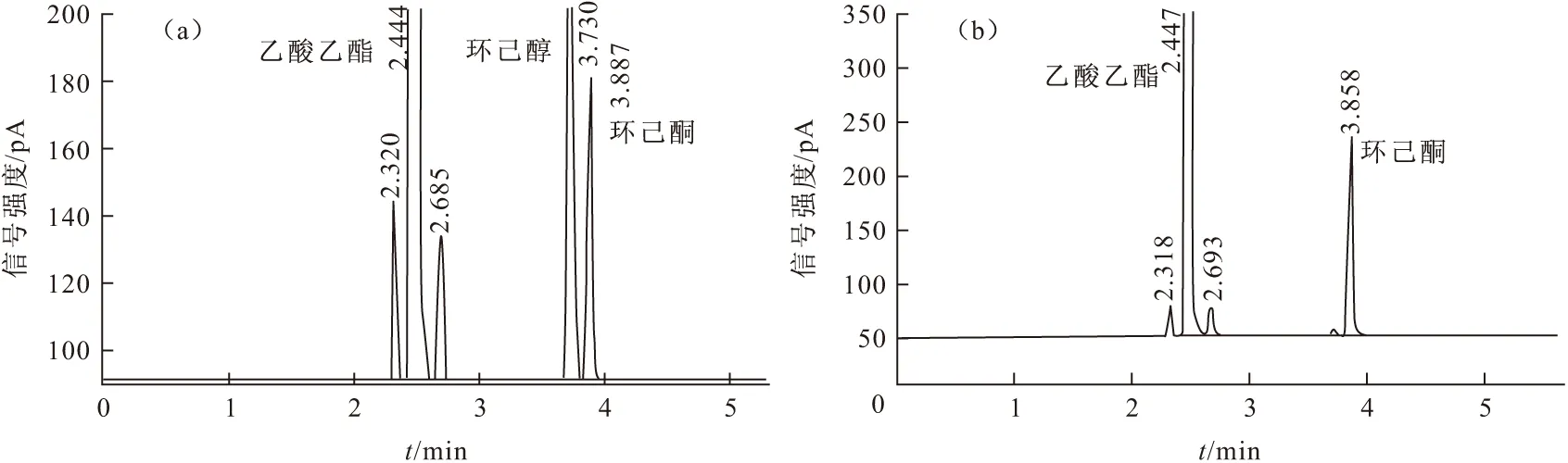
a.E.coli BL21(pET-eryKR1)2还原环己酮转化液 b.E.coli BL21(pET-eryKR1M)还原环己酮转化液
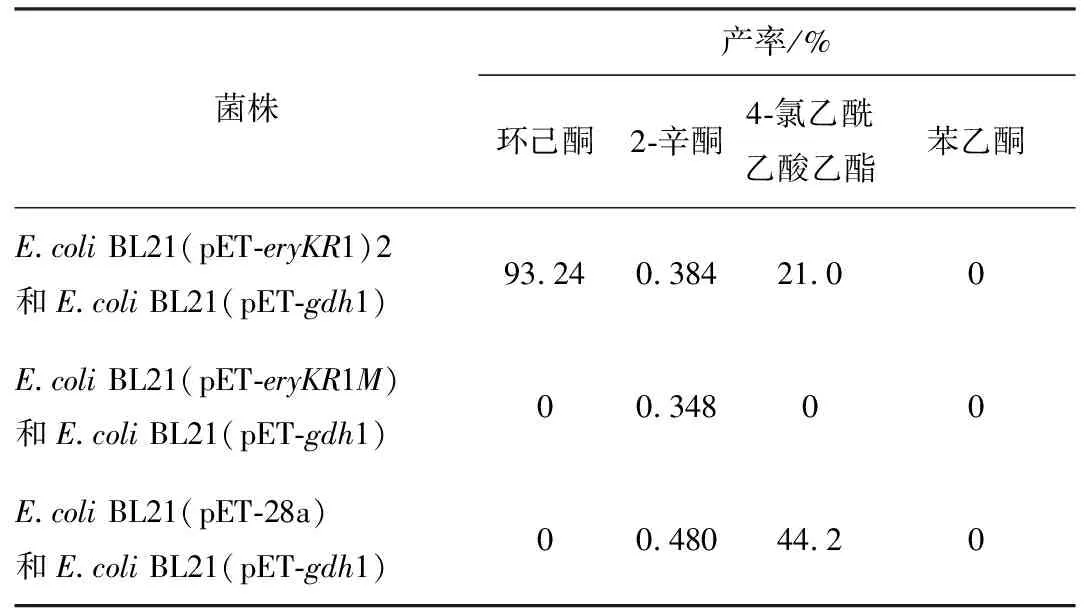
表1 重组菌E.coli BL21(pET-eryKR1M)对4种底物的转化液的气相色谱检测结果
由表1和图7可知,E.coliBL21 (pET-eryKR1M)重组菌不能还原环己酮,而野生型重组菌E.coliBL21(pET-eryKR1)2却可还原环己酮产生环己醇,产率可达93.24%,且两种重组菌对其它底物几乎没有还原作用。这说明EryKR1酶域中α6和α7间的氨基酸定点突变之后,确实会影响酶域与底物环己酮的结合,使得酶域不能催化还原环己酮,但酶域也没有因此突变,而改变对其它底物的催化特异性。
利用Swiss-model对RHGVIEMP残基替换成ActKR对应位点的氨基酸残基EHYSDIWEVS的突变型EryKR1酶域的三维结构进行预测,对比突变前后EryKR1的三维结构(如图8所示),发现EryKR1酶域中α6和α7之间的氨基酸突变后(图8a),与野生型酶域(图8b)相比,该区域伸出了空间之外,这样一个突变,可能造成不同结构的底物不能正常进入到酶活性中心,进而不能发生催化反应。
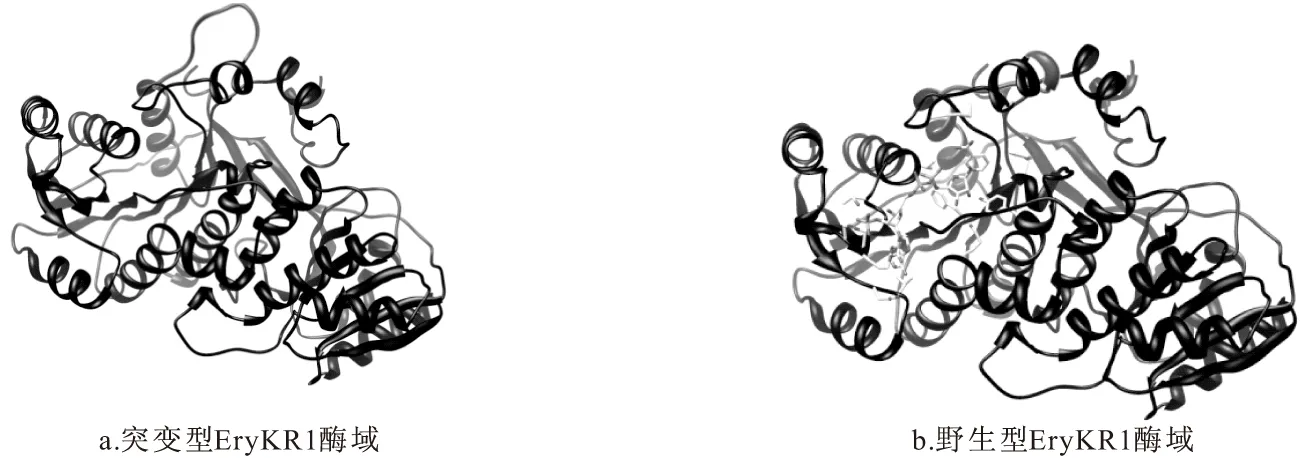
图8 突变型EryKR1酶域与野生型EryKR1的空间结构比对
2.5 讨论
在细胞正常的新陈代谢活动中,大多数的反应如糖代谢、类固醇生物合成等,都能产生醛或酮中间产物。这些含酮和醛的化合物是很强的突变剂,在生物体中,存在很多酶可代谢这些中间产物,如短链脱氢酶/还原酶家族和醛酮还原酶超家族(Aldo-keto reductase,AKR)等,都可以将羰基还原成相应醇[13]。短链脱氢酶/还原酶家族是氧化还原酶超家族之一,该家族大约有3000个成员,作用的底物广泛,包括乙醇、糖、类固醇、芳香族化合物等。SDRs家族成员虽然一级序列只有15%~30%的同源性,但是三级结构却有着高度的相似性[14](除了羧基端有较大的差异),均形成典型的Rossman折叠,具有很多保守的氨基酸序列。放线菌素聚酮还原酶ActKR和糖多孢红霉菌聚酮合成酶的酮还原酶域均属于SDR超家族[4,5]。
2004年,Korman等和Hadfield等提出芳香族聚酮还原酶和其它SDR家族成员最大的差异是α6和α7间的10个氨基酸残基,而这个区域为底物结合口袋的一部分,是SDR家族中最不保守的区域,决定了SDR家族成员的不同的底物特异性[3,4]。本研究根据ActKR和EryKR1的氨基酸序列比对结果和预测的二维结构比较结果,推测EryKR1酶域中的α6和α7之间的氨基酸序列RHGVIEMP也为底物结合口袋中的一部分,决定了该酶域的底物特异性。并利用重叠PCR技术,将eryKR1基因中编码该区域的碱基替换成编码ActKR中对应序列EHYSDIWEVS的相应碱基,并构建了异源表达该突变型eryKR1基因(即eryKR1M基因)的重组菌E.coliBL21(pET-eryKR1M),结果发现与野生型重组菌株E.coliBL21(pET-eryKR1)2相比,突变型重组菌不能还原环己酮,且对于其它类型的羰基底物,也没有因突变而产生还原能力,说明该区域的突变使得原来的底物环己酮不能进入到酶的活性中心,且尽管替换成芳香族酮还原酶的对应位点,也不能因此使芳香族底物苯乙酮进入到酶的活性中心,说明该区域虽然控制底物特异性,但仍需要和其它底物结合位点相互配合,才能使底物进入酶的活性中心,进而发生催化还原反应。由此可见,如果想通过定点突变改变EryKR1的底物特异性,除了该区域的突变,可能还需要其它的底物结合位点的突变进行配合。
3 结论
为验证糖多孢红霉菌聚酮合成酶模块1的酮还原酶域EryKR1中的控制底物特异性位点,以糖多孢红霉菌基因组DNA为模板,用重叠PCR技术扩增出α6和α7之间的氨基酸残基RHGVIEMP对应的核苷酸序列被替换的EryKR1酶域DNA片段eryKR1M,并克隆到表达载体pET-28a上,构建了质粒pET-eryKR1M,转化到EscherichiacoliBL21(DE3)后,获得了重组菌株E.coliBL21(pET-eryKR1M)。经IPTG诱导后,SDS-PAGE电泳分析表明重组大肠杆菌E.coliBL21(pET-eryKR1M)中的重组蛋白质表达量占全菌胞内可溶性蛋白质的5.7%。E.coliBL21 (pET-eryKR1M)和异源表达枯草芽孢杆菌葡萄糖脱氢酶基因的重组菌E.coliBL21 (pET-gdh1)进行双重组菌耦合,对4-氯乙酰乙酸乙酯、苯乙酮、2-辛酮和环己酮4种底物进行转化还原,利用气相色谱分析转化液,结果显示突变型的重组菌E.coliBL21(pET-eryKR1M)失去野生型重组菌E.coliBL21 (pET-eryKR1)2还原环己酮的能力,结合EryKR1酶域与放线菌素聚酮还原酶ActKR的氨基酸序列比对和二维结构比较的结果,推测α6和α7间氨基酸残基RHGVIEMP为EryKR1酶域中的底物结合口袋组成单元,对酶活性保持非常重要。
[1] Castoe T A,Stephens T,Noonan B P,et al.A novel group of type I polyketide synthases (PKS) in animals and the complex phylogenomics of PKSs[J].Gene,2007,392(1-2):47-58.
[2] Siskos A P,Baerga-Oritz A,Bali S,et al.Molecular basis of Celmer′s rules:Stereochemistry of catalysis by isolated ketoreductase domains from modular polyketide synthases [J].Chem Biol,2005,12(10):1145-1153.
[3] Korman T P,Hill J A,Vu T N,et al.Structural analysis of actinorhodin polyketide ketoreductase:Cofactor binding and substrate specificity[J].Biochemistry,2004,43(46):14529-14538.
[4] Hadfield A T,Limpkin C,Teartasin W,et al.The crystal structure of the actⅢ Actinorhodin polyketide reductase:Proposed mechanism for ACP and polyketide binding[J].Structure,2004,12(10):1865-1875.
[5] Keatinge-Clay A T,Stroud R M.The structure of a ketoreductase determines the organization of theβ-carbon processing enzymes of modular polyketide synthases[J].Structure,2006,14(4):737-748.
[6] Bali S,O′Hare H M,Weissman K J.Broad substrate specificity of ketoreductases derived from modular polyketide synthases[J].ChemBioChem,2006,7(3):478-484.
[7] Sam Brook J,Frisch E F,Maniatis T.Molecular Cloning:A Laboratory Manual (2nd ed)[M].New York:Cold Spring Harbor Laboratory Press,1992:1-75,908.
[8] Hopwood D A,Bibb M J,Chater K F,et al.Genetic Manipulation of Streptomyces:A Laboratory Manual[M].Norwich:The John Innes Foundation,1985:239,245,282.
[9] 李凌凌,张部昌,张华,等.糖多孢红霉菌λC3-SRR突变体的构建及其产物鉴定[J].军事医学科学院院刊,2004,28(4):314-318.
[10] 冯超,雷莹,刘永忠.柑橘L-半乳糖-1,4-内酯脱氢酶基因的克隆[J].华中农业大学学报(自然科学版),2009,28(6):731-735.
[11] 李凌凌,吕早生,吴敏,等.重组的葡萄糖脱氢酶催化辅酶的再生性质[J].华中科技大学学报(自然科学版),2010,38(3):112-115.
[12] 李凌凌,吕早生,关海燕,等.糖多孢红霉菌酮还原酶域在羰基还原中的应用[J].华中科技大学学报(自然科学版),2011,39(2):72-75.
[13] Barski O A,Tipparaju S M,Bhatnagar A.The aldo-keto reductase superfamily and its role in drug metabolism and detoxification[J].Drug Metab Rev,2008,40(4):553-624.
[14] Persson B,Bray J E,Bruford E,et al.The SDR (short-chain dehydrogenase/reductase and related enzymes) nomenclature initiative[J].Chem Biol Interact,2009,178(1-3):94-98.
