哈尔滨产乙醇杆菌属AFLP反应体系的建立与优化
2011-03-14郑国香陈忠林郑文玲关正军吴忆宁任南琪
郑国香,陈忠林,郑文玲,关正军,吴忆宁,任南琪
(1.哈尔滨工业大学市政环境工程学院,150090哈尔滨,rnq@hit.edu.cn;2.东北农业大学工程学院,150030哈尔滨)
扩增片段长度多态性(Amplified Fragment Length Polymorphism,AFLP)是结合 RFLP和RAPD优点的基础上发展起来的一项技术.基于其可靠性和PCR技术的高效性,且具有快速、灵敏、稳定、所需DNA量少、多态性检出率高、重复性好、可以在不知道基因组序列特点的情况下进行研究等特点,被认为是一种十分理想的、有效的、先进的分子标记方法,现已广泛用于遗传图谱构建[1-2]、遗传多样性研究[3-5]、基因定位及品质鉴定[6-7]等方面.利用AFLP分子标记的方法探讨产氢突变菌株和野生菌株的基因组DNA之间的遗传多态性差异,将为更深入地研究产氢突变菌株的突变机理提供重要的信息基础.针对哈尔滨产乙醇菌杆菌属的分子标记研究还未见有报道,本研究以发酵产氢细菌作为研究对象,建立和优化适合于哈尔滨产乙醇杆菌属基因组AFLP分析的技术体系,为进行发酵产氢细菌的遗传代谢机理的深入研究提供重要的技术支撑和信息基础.
1 实验
1.1 实验菌株和培养条件
本实验所用的出发菌株分离于CSTR生物制氢反应器的活性污泥,为乙醇型厌氧发酵产氢细菌,哈尔滨产乙醇杆菌Ethanoligenens harbinense ZGX4,以其为出发菌株经过诱变筛选获得突变株Ethanoligenen harbinenseYR-3,以上菌株由哈尔滨工业大学市政环境生物技术中心提供.
1.2 微生物的保存、激活及厌氧培养条件
[8]方法进行.
1.3 DNA的提取
将1 mL已经预先活化的菌液接入10 mL的RZL-2培养基中,37°C进行厌氧过夜培养,然后将2 mL的接种液转接入装有50 mL RZL-2培养基的培养瓶中,在37°C培养24~28 h.菌液的DNA提取利用华舜细菌DNA提取试剂盒,提取的DNA用0.8%琼脂糖电泳进行检测,并用蛋白质核酸分析仪(Beckman Coultor DU800)检测DNA的质量和纯度.
1.4 AFLP指纹分析体系的优化
1.4.1 酶切与连接
1)酶切:37℃下,采用EcoRI和MseI两种酶对细菌DNA的全基因组进行酶切.
2)纯化酶切样品:将1/10体积的醋酸钠和2.5体积的冰醋酸加入酶切样品,-20℃条件下保存2 h;然后在4℃,20 000 r/min离心15 min,弃上清液,加70%乙醇溶解沉淀,20 000 r/min离心15 min,弃上清液,干燥,30 μL TE液溶解,置于-20°C下保存.
3)接头的连接:利用T4 DNA酶连接DNA酶切片段和接头.(酶切+接头)混合样品分别稀释10、20和30倍,做预扩增的模板.
1.4.2 预扩增反应
94℃变性30 s,56℃退火30 s,72℃延伸80 s.取4 μL样在0.8%的琼脂糖凝胶电泳检测,样品稀释10、20和30倍,做选择性扩增的模板.
1.4.3 选择性扩增反应
采用选择性引物对进行特异性选择扩增.程序:30个循环,94℃30 s,65℃→55℃30 s,72℃80 s退火温度每个循环降0.3℃.取PCR样品3 μL进行琼脂糖电泳检测.
2 结果与讨论
扩增片段长度多态性分子标记(AFLP)具有在一次实验中可同时观测大量限制性片段的优点,是目前最有效的分子标记技术.AFLP反应程序中模板DNA质量及酶切片段扩增反应的条件是否恰当都将影响最终结果的分析.因此,利用产氢野生菌株和突变菌株作为研究对象,探讨和优化AFLP程序中的试验条件将为增加产氢发酵细菌的遗传多态性和高效性提供重要的实验基础.
2.1 模板DNA的质量和纯度
AFLP对样本的DNA纯度要求较苛刻,基因组DNA的纯度直接影响AFLP后续的实验结果.使用细菌DNA抽提试剂盒对产氢野生菌株ZGX4和突变菌株YR-3进行了DNA提取.利用紫外分光法测定DNA的质量和纯度,结果表明,DNA质量在50~100 ng时,当 OD260/OD280小于1.8时,DNA样品中存在蛋白质污染现象,见图1的泳道3和4,同时出现明显的弥散现象,即DNA降解迹象.而OD260/OD280大于1.9的DNA样品,存在明显的RNA污染和DNA降解脱尾现象,如图1中的泳道5和6所示;选用OD260/OD280在1.8~1.9之间的DNA样本,经琼脂糖电泳检测结果(图1的泳道1和2)显示,产氢菌株的DNA带型整齐,无RNA污染和DNA降解,完全符合AFLP试验要求,进行PCR扩增可以获得稳定、清晰的带谱,表明DNA质量符合AFLP后续的试验操作要求.
2.2 基因组DNA的酶切和连接
通常情况下,选用单一限制性内切酶对DNA进行酶切,产生的DNA片段较少,体现出来的DNA片段多态性效果较差[4].AFLP所使用的限制性内切酶一般采用两种,一种是内切酶的识别位点是6个碱基,例如EcoRI、PstI和SacI;另一种是4碱基内切酶,如MseI、TaqI和SseI.选用两种酶同时酶切可以产生比较小的酶切片段,经过PCR反应扩增出的产物范围可在100~2 000 bp之间.本实验选用EcoRI/MseI作为产氢细菌YR-3和ZGX4基因组DNA的限制性内切酶.

图1 产氢细菌基因组DNA琼脂糖电泳图
AFLP片段的多态性及丰富性是由酶切的多态性和丰富性决定的,因此,酶切质量是决定AFLP成功与否的关键前提条件之一.酶用量过大,首先是造成浪费,MseI酶是稀有酶,价格昂贵,用量过多还会在相同时间出现酶切过头的现象.用量过少,所需要的时间过长,而且常常会导致酶切不完全.因此,准确把握酶的用量及酶切时间十分重要.
2.2.1 酶切用量
大量实验已经表明,AFLP的两种限制性内切酶的总量不能超过反应体积的1/10[9],本实验针对发酵产氢细菌基因组DNA材料,采用EcoRI (20 U/μL)/MseI(10 U/μL)做双酶切,设定其用量组合分别为 1,EcoRI(0.5μL)/MseI (0.2 μL);2,EcoRI(1.0 μL)/MseI(0.4 μL); 3,EcoRI(1.5 μL)/MseI(0.6 μL);4,EcoRI (2.0 μL)/MseI(0.7 μL);5,EcoRI(2.5 μL)/MseI (1 μL);6,EcoRI(3.0 μL)/MseI(1.5 μL),DNA质量为60 ng,时间为3 h.利用琼脂糖凝胶电泳进行检测,具体的酶切用量对产氢细菌基因组DNA产生的酶切效果见图2所示.
试验结果表明,利用EcoRI(0.5 μL)/MseI (0.2 μL)对哈尔滨产乙醇发酵细菌的DNA进行酶切之后效果很差,如图2的泳道1,DNA酶切片段多分布在800~2 000 bp,基本没有切开;利用EcoRI (1.0 μL)/MseI(0.4 μL)和EcoRI(1.5 μL)/MseI (0.6 μL)组合的酶切效果表明,DNA酶切片段分布的范围在500~2 000 bp,如图2的泳道2和3所示;而通过对比发现,利用 EcoRI(2.0 μL)/MseI (0.7 μL)和EcoRI(2.5 μL)/MseI(1 μL)两个酶切组合的试验结果显示,在100~2 000 bp都有DNA酶切片段出现,见图2的泳道4和5,酶切用量都比较理想,鉴于经济性考虑,在相同酶切效果的前提下,选择EcoRI(2.0 μL)/MseI(0.7 μL)为哈尔滨产乙醇杆菌属的最佳酶切用量;当继续增加酶切用量,即EcoRI(3.0 μL)/MseI(1.5 μL)时,如图2的泳道6所示,出现了小于100 bp的DNA酶切片段.可见,限制性内切酶用量太少,导致酶切不完全,基因组多态性不丰富.酶用量过多,既造成了浪费,又容易出现酶切过头现象,产生低于100 bp的DNA片段,导致接头接不上,PCR反应失败[10].

图2 产氢细菌基因组DNA不同酶切用量的酶切效果
2.2.2 酶切时间
在确定最佳酶切组合和酶切用量后,选择最恰当的酶切时间也是影响酶切成败的必要条件.如果酶切时间过短,酶切不完全,酶切片段不能覆盖整个基因组,影响PCR扩增的多态性[11].从图3可以看出,酶切3 h的效果比较理想(泳道3),酶切片段在100~2 000 bp之间都有分布.而泳道1和2的酶切时间相对较短,分别为1 h和2 h,DNA酶切不充分.泳道4(4 h)和5(5 h)显示酶切时间过长,出现100 bp以下的片段,酶切过头将使接头连接不上,导致PCR反应无结果.因此,通过酶切时间的试验确定利用EcoRI(2.0 μL)/MseI(0.7 μL)酶切哈尔滨产乙醇杆菌属的最佳时间为3 h.将MseI和EcoRI接头配制成终质量浓度为100 μmol/L,T4 DNA酶连接DNA酶切片段和接头,连接时间过夜效果最好.
2.3 预扩增反应条件的选择
AFLP反应程序中另一重要的环节就是酶切连接产物的预扩增反应,该反应起着呈上启下的作用,既反映酶切连接效果的好坏,又决定后续选择性扩增反应成功与否.预扩增的目的是为选择性扩增提供大量的模板,同时对模板起到选择性纯化的作用,以使选择性扩增能产生清晰、稳定、易重复的条带[12-13].本试验酶切和接头的混合样本分别稀释10倍(带1和4)、20倍(带2和5)和30(带3和6)倍进行预扩增.图4显示,样品稀释的倍数对预扩增影响的效果不是很明显,3个梯度的稀释效果都较好,扩增信号强,相对分子质量大小在200~1 000 bp之间出现连续、均匀的弥散带,表明预扩增效果较好,稀释后可为选择性扩增反应提供理想的模板.

图3 产氢细菌基因组DNA不同酶切时间的酶切效果

图4 预扩增反应图谱
2.4 选择性引物组合的选择
选择性引物的筛选很重要,不同的引物组合扩增效果明显不同.而且不同的微生物基因组DNA,同一引物组的扩增效果也大不相同.选择性引物中3'末端选择性核苷酸数目的多少确定了AFLP扩增产物的多少,一般选择性碱基数目越少,扩增的条带数越多;选择性碱基数目越多,扩增的条带数越少.条带太多,不利于分辨;而条带太少,则不利于多态性的检出[14].因此,根据具体的实验材料即基因组DNA的大小确定引物末端所需的选择性核苷酸数目.
哈尔滨产乙醇厌氧发酵产氢细菌的基因组较小,所以,采用的扩增策略是在两种限制性内切酶的核心序列及识别序列基础上分别在3'增加一个选择性碱基(A,C,G,T)进行随机组合,表1显示的是扩增产氢野生菌株ZGX4和突变菌株YR-3的选择性引物的序列.选择稀释10倍的预扩增产物为模板,利用0.8%琼脂糖凝胶电泳对16个限制性酶切引物组合扩增出的DNA片段多态性进行检测.每一个引物组合的试验对象为野生产氢菌株ZGX4(左侧)和突变菌株YR-3(右侧),结果表明,不同引物组合的选择性扩增DNA多态性分布差异较为明显,具体见图5所示.其中以1 (EcoRI-A/MseI-A)、2(EcoRI-A/MseI-C)、5(EcoRI-C/MseI-A)及6(EcoRI-C/MseIC)为选择性扩增引物组合扩增酶切片段的PCR效果表现出的差异性不明显,产氢菌株ZGX4和YR-3的DNA酶切片段的PCR产物集中分布在100~500 bp之间,DNA扩增片段太小,表明基因组DNA片段的多态性丰度不够,不能很好地表现出基因组酶切片段的多态性差异,因此,本试验可以确定(EcoRI-A/MseI-A)、(EcoRI-A/MseIC)、(EcoRI-C/MseI-A)及(EcoRI-C/MseIC)引物组合不适合作为哈尔滨产乙醇杆菌属细菌ZGX4和YR-3的选择性扩增引物.相对而言,利用引物组合4(EcoRI-A/MseI-G)、9(EcoRIG/MseI-A)、10(EcoRI-G/MseI-C)、13 (EcoRI-T/MseI-A)、14(EcoRI-T/MseI-C)、15(EcoRI-T/MseI-G)、16(EcoRI-T/MseIT)对产氢野生菌株ZGX4和突变菌株YR-3的酶切片段进行PCR的扩增结果显示,扩增片段在100~1 800 bp之间呈现出明显的特异性及多态性分布.而引物3(EcoRI-A/MseI-G)、7(EcoRIC/MseI-G)、8(EcoRI-C/MseI-T)和 12 (EcoRI-G/MseI-T)组合的DNA扩增片段在琼脂糖凝胶中表现相似的的分布状态,野生菌株和突变菌株的DNA酶切片段的扩增产物所呈现出的特异性不强,差异微小,因此,不适合作为哈尔滨产乙醇杆菌属AFLP检测技术的选择性引物组合.
①参见北京市第一中级人民法院[2001]一中知初字第185号“白秀娥与国家邮政局、国家邮政局邮票印制局”一案。
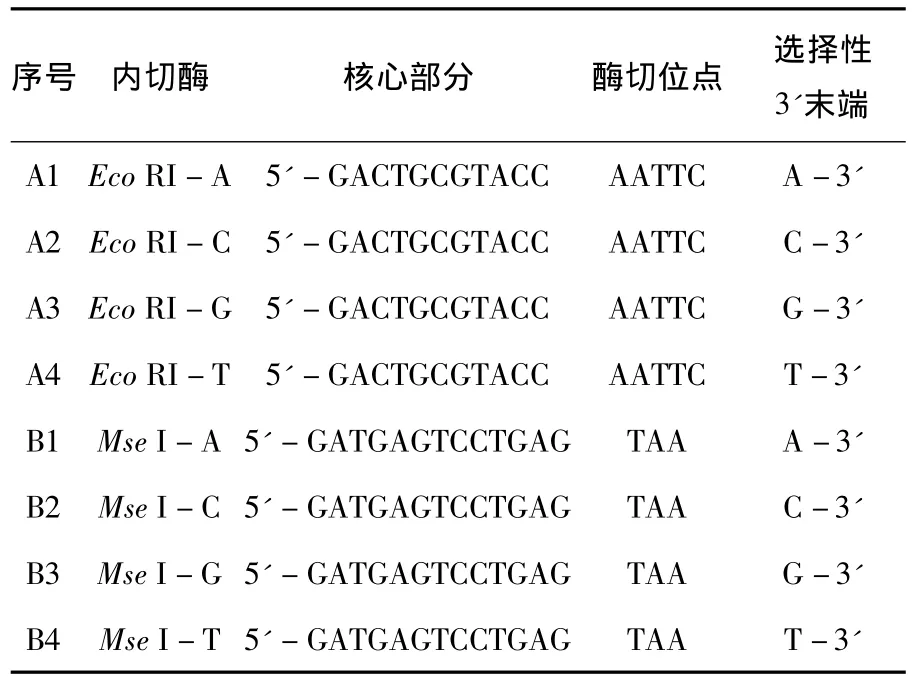
表1 选择性引物的序列

图5 不同引物组合扩增产氢细菌ZGX4和YR-3的PCR产物在琼脂糖凝胶电泳中的多态性
3 结论
1)AFLP的反应体系为 20 μL,利用EcoRI/MseI作为产氢细菌YR-3和ZGX4基因组DNA的限制性内切酶,最适酶切基因组DNA质量和纯度(OD260/OD280比值)范围分别为50~100 ng和1.8~1.9.
2)最佳酶切用量和酶切时间分别为EcoRI (2.0 μL)/MseI(0.7 μL)和3 h;预扩增产物稀释10倍为模板,以引物组合EcoRI-A/MseIG、EcoRI-G/MseI-A、EcoRI-G/MseI-C、EcoRI-T/MseI-A、EcoRI-T/MseI-C、EcoRI-T/MseI-G和EcoRI-T/MseI-T进行选择性扩增,效果较为理想,可以获得较为明显的DNA多态性分布差异.
参考文献:
[1] GANTER P F,BARROS D E,LOPES M.The use of anonymous DNA markers in assessing worldwide relatedness in the yeast species pichia kluyveri bedford and kudrjavzev[J].Can J Microbiol,2000,46(11):967-980.
[2] ZIMNOCH-GUZOWSKA E,MARCZEWSKI W,LEBECKA R,et al.QTL analysis of new sources of resistance to Erwinia carotovo-rassp.atrosepticain potato done by AFLP,RFLP and resistance gene-like markers[J].Crop Sci,2000,40:1156-1167.
[3] MCULIFFE L,KOKOTOVIC B,AYLING R D,et al. Molecular epidemiological analysis of mycoplasma bovis isolates from the United Kingdom shows two genetically distinct clusters[J].J Clin Microbiol,2004,42 (10):4556-4565.
[5] KUMAR V,SHARMA S,KERO S,et al.Assessment of genetic diversity in common bean(Phaseolus vulgaris L.)germ plasm using amplified fragment length polymorphism(AFLP)[J].Scientia Horticu Lturae,2008,116:138-143.
[6] THOMAS C,VOS C,ZABEAN M,et al.Identification of amplified restriction fragment length polymorphism(AFLP)markers lightly linked to the tomatoCf-9 gene for resistence to Cladosporiumfluvum[J].The Plant Jour,1995,8:785-794.
[7] SIMONS G,VANDER L T,DIERGRADE P,et al. AFLP-based fine mapping of the Mlo gene to a 30- kb DNA segment of the barley genome[J].Genetics,1997,44:61-70.
[8] 郑国香,任南琪,李永峰,等.一株高效产氢突变体RF-9的筛选与产氢特性[J].中国环境科学,2007,27(2):184-188.
[9] VOS P R,HOGERS M,BLEEKER M,et al.AFLP: a new concept for DNA fingerprinting[J].Nucleic Acids Res,1995,23:4407-4414.
[10] WANG Z,GAO H W,WU Y Q,et al.Genetic diversity and population structure of Caragana korshinskii revealed by AFLP[J].Crop Science,2007,47:1737-1743.
[11] LARSON S R,JONES T A,JEBSEN K B.Population structure in Pseudoroegneria spicata(Poaceae: Triticeae)modeled by Bayesian clustering of AFLP genotypes[J].American Journal of Botany,2004,91(11):1789-1801.
[12] 宋晓飞,李晓丽,冯志红,等.黄瓜基因组DNA提取及AFLP体系优化研究[J].安徽农业科学,2009,37(29):14035-14037.
[13] 陈旭辉,高玉葆,朱敏杰,等.小叶锦鸡儿基因组DNA的提取及AFLP反应体系的建立[J].植物研究,2009,29(5):529-533.
[14] SHI J,XIN L,YANG Y,et al.Discussion on AFLP molecular markers in Pipermethysticum and pepper[J].Agricultural Science&Technology,2007,8(3/ 4):5-10.
