从养殖贝类和大菱鲆分离的海洋发光弧菌的分类地位分析
2011-03-14叶德赞nterJost黄翔玲郑森林
刘 斌,叶德赞,Günter Jost,林 义,黄翔玲,郑森林
(1.国家海洋局 第三海洋研究所,福建 厦门 361005; 2.国家海洋局海洋 生物遗传资源重点实验室,福建厦门 361005; 3.德国莱布尼兹波罗的海研究所,罗斯托克 D-18119)
弧菌是海洋环境中常见的条件致病菌,在某些环境条件下可引起水产养殖动物的病害。迄今已有分离报道的水产致病性弧菌达10多种,危害的对象包括鱼类、甲壳类和贝类动物等[1]。刘文华等[2]对厦门浔江牡蛎养殖区弧菌的研究表明,水体弧菌密度与牡蛎体的弧菌密度呈密切相关性,是引起养殖区牡蛎严重死亡的原因;Vibrio campbellii可引起鱼、虾和贝类的大量死亡[3]; 发光弧菌V.fischeri可引起大黄鱼体表溃烂症[4]; 而同样具有发光特性的V.harveyi感染对虾幼体后即在幼体体内大量繁殖,致使对虾幼体活力下降,游泳能力变差,体色发白,部分肌肉坏死,是一种在对虾育苗和养殖中都经常发生的细菌性疾病,具有发病急,传播广,感染力强,致死率高和防治困难等特点,给世界养虾业造成了巨大的经济损失[5-6]。
作者采用ARDRA、16S rDNA序列测定、Biolog碳源代谢分析及药敏试验等方法,对从福建省晋江市附近水产养殖场的贝类软组织及波罗地海大菱鲆肠道内含物中分离得到的 16株发光细菌的分类地位进行了分析,也为防治海水养殖中潜在的由发光弧菌引发的病害提供了帮助。
1 材料与方法
1.1 培养基及主要试剂和仪器
发光菌培养基:蛋白胨5.0g,酵母膏1.0g,甘油3 mL,陈海水1000 mL,琼脂15 g,pH 7.0; 引物由上海英骏生物技术有限公司合成,DNA Marker、Ex Taq酶及限制性内切酶AfaⅠ、MspⅠ均购自大连Takara公司; Biolog微生物自动鉴定系统由美国Biolog公司生产; 药敏纸片购自杭州天和微生物试剂有限公司。
1.2 菌株来源
将2008年5月采集自福建省晋江市附近营前、白沙及江崎水产养殖场正常生长状态下的花蛤及牡蛎样品去除贝壳后研磨,取5 g经充分研磨的样品置于45 mL内置玻璃珠的无菌生理盐水中,振荡混匀,取上清液稀释为 10-1,10-2两个梯度,各梯度分别吸取 100 μL,用涂布棒均匀涂布于发光菌培养基上,25℃恒温培养24 h,于暗室中挑取发出荧光的菌株。经分离纯化,共得到12株发光细菌。采取同样的方法于2009年7月在购自市场的波罗地海大菱鲆肠道内含物中分离得到4株发光细菌。
1.3 细菌基因组DNA的提取
细菌基因组DNA的提取参照《精编分子生物学实验指南》[7]中的细菌基因组DNA的小量制备方法,并将提取的DNA模板储存于-20℃冰箱中。
1.4 细菌16S rDNA的PCR扩增
所用引物为细菌16S rDNA通用引物,扩增正向引物 27F:5'-AGAGTTTGATCCTGGCTCA-3′; 反向引物 1492R:5′-GGTTACCTTGTTACGACTT-3′。PCR反应条件为:95 ℃预变性5 min; 94 ℃变性40 s,56℃退火40 s,72℃延伸2 min,30个循环后,72℃最终延伸10 min。PCR产物用1%琼脂糖凝胶电泳检测。
1.5 16S rDNA PCR产物的ARDRA分析
PCR扩增产物用限制性内切酶AfaⅠ、MspⅠ进行双酶切,酶切反应体系为 20μL,其中 PCR 产物10 μL,内切酶各 0.5 μL,相应的 10×Buffer 2 μL,0.1%BSA 2 μL,去离子水 5 μL。37 ℃恒温 3 h。酶切产物用3%琼脂糖凝胶电泳检测。
1.6 16S rDNA PCR产物测序及系统发育树的构建
PCR产物经纯化后送上海英骏生物技术有限公司测序。用EzTaxon Server version 2.1对所得到的16S rDNA序列进行分析,并用Mega 4.1软件包采用Neighbor-Joining算法自展 1000次进行系统发育树的构建。
1.7 Biolog碳源代谢分析
按照 Biolog微生物鉴定操作步骤,调整接种菌悬液浊度为28%T,接种至96孔GN2微孔鉴定板中,于25 ℃培养至第24小时 、48小时 、72 小时,结合“人工”及“自动”两种读板模式,分别测定3次。采用 NTSYS-pc 2.10e软件对读取结果进行聚类分析。
1.8 药敏试验
药敏试验采用 K-B纸片琼脂扩散法进行,于25℃恒温培养24 h后取出,记录各药敏纸片的抑菌圈直径(包括药敏纸片直径7 mm)。按CLSI抗菌药物敏感性试验执行标准(2009年版)判断药敏结果。
2 结果与分析
2.1 发光细菌的分离、形态观察及染色
经分离纯化得到16株发光细菌(表1),经革兰氏染色及KOH拉丝法验证,这些菌株均为革兰氏阴性菌; 在发光菌甘油培养基上的菌落均为圆形,浅黄色,表面光滑湿润,边缘整齐或呈锯齿状,隆起形状分扁平和微凸; 油镜下观察,呈长杆状、短杆状或弧状; 在黑暗中均发射浅绿色荧光。
2.2 ARDRA结果分析
研究表明[8],每一个菌种的16S rDNA都有一个独特的酶切图谱,每种代表一个操作分类单位(即OTU),用16S rDNA序列的ARDRA分析可以区分出菌种。本研究选用限制性内切酶AfaⅠ和MspⅠ对16株发光菌16S rDNA的PCR扩增产物进行双酶切分析,其结果见图 1。根据图 1中的限制性酶切图谱,可将 16株菌分为 3组,即 3个不同的 OTUs,其中OTU2包含菌株FY-6和FJ-2,属于OTU3的仅有菌株FJ-1,其余13个菌株则均属于OTU1。

表1 16株发光细菌来源Tab.1 The sample sources of 16 luminous bacteria
2.3 16S rDNA序列测定及系统发育分析
根据发光细菌菌落和细胞形态、16S rDNA序列的ARDRA指纹图谱及样品来源的不同,选取其中5个菌株进行 16S rDNA序列测定并进行系统发育分析。所有测序菌株均与数据库中已知标准菌株的16S rDNA序列具有较高的相似性(表 2)。结合实验菌株16S rDNA序列的最高相似性及在系统发育树(图 2)中所处的分支可知,所有5株发光菌株均属于Vibrio属,分别与V.campbellii,V.orientalis,V.fischeri及V.azureus具有最近的亲缘关系,其中菌株 FY-1和FB-1虽同属OTU1,鉴定结果却有所不同,但两株菌在系统发育树中的位置较为接近。
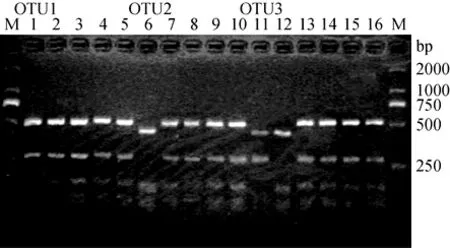
图1 限制性内切酶AfaⅠ和MspⅠ双酶切16S rDNA的琼脂糖凝胶电泳图Fig.1 Agarose gel electrophoresis of AfaⅠ+ MspⅠdigestion of PCR-amplified 16S rDNAs
2.4 Biolog碳源代谢分析
微生物对碳源的利用能力是表征微生物生长情况的主要指标[9]。本研究中 5株海洋发光弧菌的Biolog碳源代谢分析结果与数据库中标准菌株的相似性较低,无法鉴定至种。5株菌对 95种碳源的利用率及聚类分析结果见图3,各株菌的碳源利用图谱均有所不同,其中FY-1可利用57种碳源,FB-1可利用54种,FJ-1则可利用34种,而FJ-2与FY-6的碳源利用谱较窄,分别可利用18种和14种碳源,但各株菌均可利用吐温 40、N-乙酰基-D-葡萄糖胺、D-纤维二糖、D-果糖、α-D-葡萄糖、L-天冬酰胺酸、L-谷氨酸、甘氨酰-L-天门冬氨酸、肌苷和尿苷等10种碳源。由图3中的碳源代谢聚类分析结果可知,菌株FY-6与FJ-2碳源利用情况虽有所差异,但处于同一分支,相似度较高,而其他菌株的碳源利用情况差异较大。
2.5 药敏试验
选用复方新诺明、氟哌酸等11种药物进行了药敏性试验。依据2009年版CLSI抗菌药物敏感性试验执行标准判断药敏结果(表 3)。试验结果表明,各株菌对同一药物的敏感性多有不同,但系统发育分析中亲缘关系较近的FY-6及FJ-2也具有较为相似的药物敏感性,且所有菌株均对复方新诺名和庆大霉素等敏感。
3 讨论
近些年来,分子生物学手段发展迅速,一些新的基于细菌基因组序列的分子生物学方法,如ARDRA、rep-PCR、RAPD等,为细菌的分离鉴定和多样性研究提供了有力的工具。ARDRA方法可在16S rDNA序列水平上区分细菌种的差异。但研究中也发现ARDRA存在着一定的局限性:用ARDRA区分不同的 16S rDNA片段的精确性有赖于所选用的限制性内切酶的种类和数量,选用不同的酶可能会划分出不同的OTUs,尤其是要区分同一个属内的不同种时需要用几种不同的酶消化,以综合分析得到的电泳图谱[10]。本研究中,通过AfaⅠ和MspⅠ两种酶的ARDRA方法筛选菌株,16株发光细菌可分为3个OTUs,但随后的序列测定和系统发育分析结果表明,两种酶的 ARDRA方法可在属水平上对细菌做有效甄别,也可以区分同一属内亲缘关系较远的种,但难以区分同一属内的近缘种。

表2 5株发光细菌的16S rDNA序列相似性Tab.2 Similarity of 16S rDNA sequences from the five luminous bacteria

图2 基于16S rDNA序列的系统发育树Fig.2 Phylogenetic tree based on 16S rDNA sequences of the five luminous bacteria and relating species
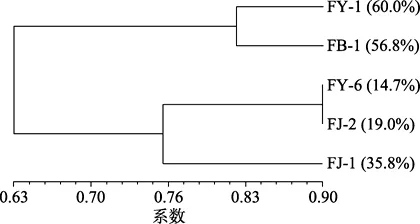
图3 发光弧菌对 95种有机碳源的利用率及聚类分析结果Fig.3 Cluster analysis of the utilizing rates of 95 C-Sources by the 5 strains
16S rDNA序列测定及系统发育分析表明,选取的5株发光菌均属于Vibrio属,其中菌株FJ-1的近缘种为发光细菌V.fischeri; 来源不同的菌株FY-6及FJ-2与具有发光特性的V.orientalis以100%的置信度处于同一分支,亲缘关系最近;V.azureus为最近报道的一发光新种[11],作者自波罗地海分离得到的发光菌株FB-1与其具有最大的16S rDNA序列相似性达98.613%; 而发光菌株FY-1与V.campbellii序列相似性最高(99.926%),但未见有关于V.campbellii发光的正式报道。谢文阳等[12]从台湾沿岸浅水区域分离出来 4株表型特性非常相似的发光菌株,鉴定为V.harveyi类似种,但也大多与V.campbellii而非V.harveyi,具有最高的16S rDNA序列相似性。

表3 11种药物对5株发光弧菌的抑菌圈直径及判断结果Tab.3 Sensitivity of the five luminous bacteria to 11 drugs
Biolog系统是一种测定微生物对95种不同单一碳源利用能力的快速、简便方法,适用于陆地环境、临床细菌鉴定[13]。本研究中利用Biolog系统得到的5株海洋发光弧菌的碳源利用图谱均有所不同,但无法鉴定至种,主要是因为海洋细菌的生长条件和陆地细菌有较大区别,该系统的某些实验条件不适合海洋细菌,并且目前该鉴定系统收集的标准菌株数据库尚无足够的海洋细菌的特征资料,可鉴定的种类有限[14]。药敏试验也属于菌种鉴定的生化指标范围,其结果可为海水养殖中潜在的由发光弧菌引起的病害的防治提供帮助。两种方法得到的菌株 FY-6和 FJ-2的实验结果较为相似,与 ARDRA及 16S rDNA序列测定方法一样,可将其划分至同一群体,推测为V.orientalis的不同亚种。
一般来说,基于16S rDNA序列的分析可将细菌鉴定到属的水平,常规生理生化特征研究方法依然是将细菌鉴定到种的最精确的方法。结合ARDRA、16S rDNA序列测定、Biolog碳源代谢分析及药敏试验等方法,虽无法鉴定海洋细菌至种,但可将细菌区分为属内分类地位相一致的不同群体,为后续更精确的在生理生化方面鉴定细菌提供参考依据。
[1]李亚晨,包永明,吕建发,等.海洋水产动物弧菌病的生物防治[J].水产科学,2004,23(2):35-38.
[2]刘文华,叶德赞,倪纯治,等.厦门浔江湾牡蛎养殖区弧菌研究[J ].台湾海峡,1996,15(4):412-415.
[3]沈晓盛,蔡友琼,房文红,等.养殖牡蛎体内检出坎氏弧菌的鉴定[J].微生物学报,2005,45(2):177-180.
[4]张庆华,瞿小英,郑岳夫,等.大黄鱼体表溃烂症病原菌的鉴定[J].上海水产大学学报,2003,12(3):233-237.
[5]黄万红,陈月忠.对虾发光病生物防治技术的初步研究[J].福建水产,2007,2:8-14.
[6]梁华芳,庞雄明.南美对虾和斑节对虾育苗中发光病的综合防治[J].水产养殖,2003,24(4):31-32.
[7]奥斯伯 F M,布伦特 R,金斯顿 E,等.精编分子生物学实验指南[M].北京:科学出版社,2008:39.
[8]Dunbar J,White S,Forney L.Genetic diversity through the looking glass:effect of enrichment bias [J].Applied and Environmental Microbiology,1997,63(4):1326-1331.
[9]Kennedy A C,Smith K L.Microbial diversity and sustainability of agricultural soils [J].Plant Soil,1995,23(2):69-79.
[10]Priemé A,Braker G,Tiedje J M.Diversity of nitrite reductase (nirK andnirS ) gene fragments in forested upland and wetland soils [J].Applied and Environmental Microbiology,2002,68:1893-1900.
[11]Yoshizawa S,Wada M,Kita-Tsukamoto K,et al.Vibrio azureus sp.nov.,a luminous marine bacterium isolated from seawater [J].International Journal of Systematic and Evolutionary Microbiology,2009,59:1645-1649.
[12]谢文阳,邱秀慧.海洋弧菌多样性[J].世界科技研究进展,2005,27(2):34-41.
[13]谢家仪,王永力.Biolog 细菌自动鉴定系统的应用与研究[J].微生物学通报,1996,23(5):264-267.
[14]莫照兰,茅云翔,陈师勇,等.养殖牙鲆鱼苗腹水症病原菌的鉴定及系统发育分析[J].海洋与湖沼,2003,34(2):131-141.
