大肠埃希菌耐药谱型、基因型和生物膜表型关系研究*
2011-03-07陈朝喜朱恒乾廖晓萍刘雅红
陈朝喜,朱恒乾,廖晓萍,刘雅红*
(1.华南农业大学兽医学院兽医药理与毒理学教研室,广东广州510642;2.西南民族大学生命科学与技术学院兽医药理学教研室,四川成都610041)
在抗生素压力下,大肠埃希菌(Escherichia coli,E.coli)能够形成生物被膜得以生存,导致对临床抗菌药物的敏感性降低[1]。同时生物被膜作为大肠埃希菌的毒力因子之一,常常引起持续性感染的迁延不愈[2-5]。因此,从研究耐药谱型、基因型和生物被膜表型之间相互关系的角度探究生物被膜的耐药机制,阐述抗菌药物和生物被膜形成之间的相互作用,探讨在不同抗生素压力下生物被膜形成能力的变化,对大肠埃希菌流行病学研究和大肠埃希菌生物被膜感染疾病的防治药物筛选具有一定的临床意义。
1 材料与方法
1.1 材料
1.1.1 大肠埃希菌样本来源 249株大肠埃希菌采自广东省养殖场(禽类养殖场和养猪场,包括养殖场周围的土壤和水塘样本)和宠物医院,大部分的样本从患病动物的粪便、肠道内容物和脏器分离。大肠埃希菌标准株ATCC 25922为华南农业大学兽医药理教研室保存。
1.1.2 培养基 麦康凯琼脂培养基,MH肉汤,蛋白胨大豆肉汤等为广州环凯生物科技有限公司产品;分子生物学试剂为宝生物工程(大连)有限公司产品;ERIC-PCR通用引物由上海英骏生物工程有限公司合成(ER-F:5-ATGTAAGCTCCTGGGGATTCAC-3′;ER-R:5′-AAGTAAGTGACTGGGGTGAGCG-3′)。
1.1.3 抗菌药物 恩诺沙星、多西环素、氟苯尼考、阿莫西林、利福平、氨苄西林、安普霉素、阿米卡星、庆大霉素、头孢噻呋、链霉素、新霉素和卡那霉素由华南农业大学实验药厂惠赠,临用前配成5 120 μ g/mL备用。
1.2 方法
1.2.1 药敏试验 实验步骤及结果判定按照美国临床实验室标准化委员会(NCCLS)推荐的微量肉汤稀释法操作程序进行[6]。
1.2.2 249株临床分离大肠埃希菌生物被膜表型分析 参照Peeters和Stepanovic等的微孔板法进行[7-8],具体方法如下:在无菌96孔细胞培养板的加入200 μ L经1∶100稀释的菌液,阴性对照孔只加入相应量的TSB肉汤,37℃培养24 h,然后弃去每孔内培养液并用250 μ L灭菌PBS溶液漂洗3次以彻底弃去孔内未黏附的浮游菌,孔壁上黏附的细菌用200 μ L 990 mL/L甲醇固定15 min后,弃去甲醇,室温下自然风干。200 μ L 20 g/L结晶紫染色5 min,然后将96孔微量培养板浸没在流动的自来水下冲洗干净多余的染料。待微孔板过夜自然风干后,结合在孔内的染料用160 μ L 330 g/L冰乙酸溶解,用酶标仪测定各孔的OD570值。每个细菌接种4孔,每个菌3次重复。根据测试孔与阴性对照孔OD570比值的不同,将所分离大肠埃希菌的生物被膜表型分为4个表型,即无成膜能力(—),弱成膜能力(+),中等成膜能力(++)和强成膜能力(+++)。
1.2.3 249株临床分离大肠埃希菌耐药性分析及其与生物被膜表型的关系 采用Whonet5.4软件对临床分离的249株大肠埃希菌的试验数据进行统计处理,按照耐药谱型的不同进行分析汇总。结合1.2.2结果分析耐药谱型与生物被膜表型之间关系。
1.2.4 大肠埃希菌生物被膜表型和基因型的关系
1.2.4.1 大肠埃希菌基因组DNA提取采用陶文琴等方法进行基因组DNA的抽提[9]。
1.2.4.2 ERIC-PCR反应体系及反应条件 采用肠杆菌科基因间重复一致序列引物进行扩增[10-11],ERIC-PCR的25 μ L PCR反应体系包括两条引物(50 pmol)各1 μ L、DNA溶液2 μ L、dNTP混合物2 μ L、Ex Taq(5 U/μ L)0.25 μ L及2.5 μ L缓冲液,各组分加入后补充灭菌双蒸水至25 μ L后充分混匀,按照下面的反应条件进行:94℃预变性5 min,然后接着35个循环,循环条件为:94℃1 min,53℃1 min,72℃4 min,最后72℃10 min。PCR产物经20 g/L琼脂糖凝胶电泳,经溴化乙锭染色后用凝胶成像系统进行图像采集。
1.2.4.3 ERIC-PCR指纹图谱聚类分析 采用NTSYS pc 2.1软件对ERIC-PCR指纹图谱进行分析,得到Jaccard's遗传相似性系数矩阵,采用非加权配对法做遗传分析,绘制聚类分析树状图谱。
2 结果
2.1 249株大肠埃希菌耐药谱分布规律与成膜能力表型分析
249株不同成膜能力表型的大肠埃希菌的耐药谱型分析汇总结果显示,除了强成膜能力表型组的耐药谱型比较分散外,其余3种表型的耐药谱型主要集中在5~10耐之间(图1)。强成膜能力组的耐药谱型主要分布在4耐和7耐,分别占强成膜能力表型组的25.0%和37.5%。中等成膜能力组的耐药谱型主要集中在7~10耐,占65.96%,其中7~10各耐药谱分别占该表型的17.02%、19.15%、12.77%、17.02%。弱表型能力组的耐药谱型在5~10耐的比例相差不大,集中了该耐药谱型的81.39%,而5~10耐药谱型分别占弱表型能力组的13.95%、13.18%、12.40%、13.18%、14.73%、13.95%。无成膜能力组的耐药表型也主要分布在5~10耐之间,占84.62%,除了9耐和10耐占的比例比较大(分别为21.54%和23.08%)外,其余几个耐表型菌低于15.0%。

图1 249株不同成膜能力大肠埃希菌耐药谱分布规律Fig.1 Drug-resistance spectrum of 249 E.coli strains in different biofilm-forming ability
2.2 大肠埃希菌临床分离株ERIC-PCR指纹图谱的聚类分析
64株临床分离大肠埃希菌PCR产物15 g/L琼脂糖凝胶电泳(图2),利用UVI Photo MW软件包对ERIC-PCR的电泳图谱进行处理,图谱中每一条带(DNA片段)均作为一个分子标记,并代表一个结合位点,根据各条带对应的峰图进行相关参数(峰体积,峰高度和峰面积)的比较。然后根据各分子标记的迁移速率,将同一水平线上,即同一分子质量大小,有带的样品读为“1”,无带或者条带模糊且很淡的样品读为“0”。所得的位点按分子质量从大到小排列。读出并记录3条以上不同的带,将读出来的带(用0和1来表示)转换成Excel表格文件,制成0~1表之后,转换为纯文本文件用于数据处理。
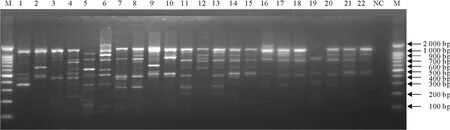
图2 ERIC-PCR琼脂糖凝胶电泳图Fig.2 Agarose gel electrophoresis of ERIC-PCR products
应用NTSYS pc2.1软件对64株临床分离大肠埃希菌ERIC-PCR指纹图谱进行分析,得到大肠埃希菌指纹图谱的Jaccard's遗传相似性系数矩阵,结果以非加权算术平均数配对进行聚类分析,自动生成相似系数和树状图。
64株大肠埃希菌的聚类分析分为A,B,C和D 4个型,从表1可以看出,不同成膜能力在各个型中均有分布,而中等成膜能力组主要分布在A和C型,成膜能力弱的组主要分布在B、C和D,无成膜能力组的菌株则主要分布在D型。ERIC-PCR基因分型技术能够实现从表型和基因水平探究大肠埃希菌菌株生物被膜的形成机理,并提供可靠的理论依据,对大肠埃希菌的流行病学研究和生物被膜感染防治药物的筛选具有一定的实际意义。

表1 临床分离株大肠埃希菌生物被膜表型和ERIC-PCR基因分型关系Table 1 Relationship between E.coli biofilm phenotype and ERIC-PCR genotype
3 讨论
3.1 耐药谱型与生物被膜表型的关系
分析耐药谱型和生物被膜表型之间的关系,有助于阐述在形成生物被膜的过程中,抗菌药物和生物被膜形成能力之间相互作用,为进一步探讨生物被膜形成对抗菌药物的耐药机制奠定基础。本研究结果表明,在临床分离的249株大肠埃希菌中,不同成膜能力表型的大肠埃希菌对实验中常用抗菌药物的耐药谱型主要集中在5~10耐之间,耐药谱型的多样性是造成生物被膜能力差异的主要原因,且在耐药谱型中耐药抗菌药物种类过多和过少对生物被膜的表型能力的影响不显著。
3.2 ERIC-PCR基因指纹图谱分析与成膜能力表型关系研究
本研究通过优化反应条件对64株临床分离的大肠埃希菌进行ERIC-PCR分析并建立基因指纹图谱,对不同生物被膜表型大肠埃希菌临床菌株进行分型,在表型水平和基因水平证实大肠埃希菌生物被膜表型与其基因型关系密切:64株大肠埃希菌临床菌株分为A、B、C、D 4个基因型且生物被膜形成能力较中等和弱的菌株大都属于B型和C型,而不能形成被膜的菌株大都属于D型,与Casarez和Wei等的研究结果相近。
3.3 ERIC-PCR分型技术探讨
相关文献报道利用ERIC-PCR对肠杆菌科进行分型研究,扩增引物的选择存在两种模式:即单引物扩增[12-13]和双引物扩增[14-15]。Yang W Q等[17]在对铜绿假单胞菌进行分型时采用ERIC-PCR单引物进行扩增,这可能是随机扩增的原理,其扩增的片段大小分布在100 bp~2 500 bp之间。而我们的研究中采用双引物进行扩增,结果扩增的条带基本和Yang W Q的报道一致,扩增引物选择的具体目的和优缺点还有待进一步研究和分析。
[1] Naves P,Delprado G,Hueives L,et al.Correlation between virulence factors and in vitro biofilm formation by Escherichia coli strains[J].Microb Pathog,2008,45:86-91.
[2] Rijavec M,Müller-Premru M,Zakotnik B,et al.Virulence factors and biofilm production amongEscherichia colistrains causing bacteraemia of urinary tract origin[J].J Med Microbiol,2008,57(11):1329-1334.
[3] 成大荣,黄维嘉,张迎春,等.不同毒力基因型大肠杆菌的毒力测定[J].动物医学展,2005,26(12):78-80.
[4] Rijavec M,Müller-Premru M,Zakotnik B,et al.Virulence factors and biofilm production among Escherichia coli strains causing bacteraemia of urinary tract origin[J].J Med Microbiol,2008,57(11):1329-1334.
[5] 刘 华,祁克宗,涂 健,等.鸡源致病性大肠埃希菌菌毛基因研究进展[J].动物医学进展,2006,27(5):16-19.
[6] National Committee for Clinical Laboratory Standards.Methods for dilution antimicrobial susceptibility tests for bacteria that grow aerobically,6th ed.Approved standard M7-A6[S].National Committee for Clinical Laboratory Standards:Wayne P A,2003:M7-A6.
[7] Peeters E,Nelis H J,Coenye T.Comparison of multiple methods for quantification of microbial biofilms grown in microtiter plates[J].J Microbiol Methods,2008,72(2):157-165.
[8] Stepanovic S,Cirkovic I,Ranin L,et al.Biofilm formation by Salmonella spp.and Listeria monocytogenes on plastic surface[J].Let in Appl Microbiol,2004,38:428-432.
[9] 陶文琴.耐氟喹诺酮类大肠杆菌基因突变及检测方法的研究[D].广东广州:华南农业大学,2004.
[10] Casarez E A,Pillai S D,Di Giouanni G D.Genotype diversity of Escherichia coli isolates in natural waters determined by PFGE and ERIC-PCR[J].Water Res,2007,41:3643-3648.
[11] Wei G F,Pan L,Du H M,Chen J Y,et al.ERIC-PCR fingerprinting-based community DNA hybridization to pinpoint genome-specific fragments as molecular markers to identify and track populations common to healthy human guts[J].J Microbiol Meth,2004,59:91-108.
[12] 晏 群,陈丽华,刘文恩,等.分子生物学分型技术ERIC-PCR的建立[J].实用医学杂志,2003,19(6):688-689.
[13] 余方友,胡龙华,贾坤茹,等.应用ERIC-PCR对多重耐药大肠埃希氏菌进行基因分型[J].江西医学检验,2005,23(6):513-515.
[14] Coquet L,Cosette P,Quillet L,et al.Occurrence and phenotypic characterization of Yersinia ruckeri strains with biofilmforming capacity in a rainbow trout farm[J].Appl Environ Microbiol,2002,68(2):470-475.
[15] 魏 华,孙立国,朱 晶,等.不同的DNA提取法对两种常见医院感染菌ERIC-PCR鉴定的影响研究[J].中华医院感染学杂志,2007,17(9):1044-1047.
[16] Yang W Q,Shi L,Jia W X,et al.Evaluation of biofilm-forming ability and genetic typing for clinical isolat es of Pseudomonas aeruginosa by enterobacterial repetitive intergenic consensus-PCR[J].Microbiol immunol,2005,49(2):1057-1061.
