CYP725A4分子序列分析与结构建模*
2011-01-24蒙海林汪建峰王勇张嗣良王小宁
蒙海林 汪建峰 王勇 张嗣良 王小宁
(1.华南理工大学生物科学与工程学院,广东广州510006;2.中国科学院上海生命科学研究院合成生物学重点实验室,上海200032;3.华东理工大学生物反应器工程国家重点实验室,上海200237)
植物来源的细胞色素P450酶在大肠杆菌等原核宿主中很难实现功能表达,从而给异源合成紫杉醇等高附加值天然产物带来困难[1-2].紫杉醇生物合成途径中涉及到多步P450酶催化的羟基化反应,其中P450725A4(由CYP725A4)催化的第1步羟基化反应已经成为制约紫杉醇异源合成研究的瓶颈[2-4].目前这步反应在异源宿主中得到的基本上是副产物[5],这可能与酶分子结构改变所引起的催化活性变化有关.因为疏水性很强的P450膜蛋白在缺乏内质网的原核宿主中实现功能表达通常需要切除其疏水的跨膜端,可能会对蛋白的最终构象产生一定影响.因此,寻求一种能有效去除P450蛋白疏水特性而又不影响其催化活性的分子改造方案是在原核宿主中实现植物P450蛋白功能表达并最终获取目标产物的有效途径,而该方案的工作基础是已知P450蛋白的结构数据.但遗憾的是,由于实验技术所限,目前通过实验测定所得到的蛋白质数据库(PDB,http:∥www.rcsb.org/pdb/home/home.do)中膜蛋白所占比例极小,而属于P450超家族的结构数据更为缺乏,因而目前尚不能基于目标P450蛋白的真实结构进行理性的分子设计从而更好地实现其功能;但是,在少数已知蛋白结构的基础上可以通过同源结构建模而得到未知的P450蛋白结构模型.这些模型在现阶段的研究工作中是具有指导意义的.然而,目前有关CYP725A4分子结构建模的研究鲜见报道.本研究以CYP725A4为例,通过序列分析和结构建模工作,在P450蛋白结构建模方面进行初步探索,为后续的紫杉醇等萜类化合物异源合成研究中的P450酶分子改造和功能表达奠定基础.
1 材料与方法
1.1 材料
CYP725A4的原始基因和蛋白序列在美国国家生物信息技术中心(NCBI,http:∥www.ncbi.nlm.nih.gov/)中检索获得,同源模板的结构信息通过检索PDB数据库获得.
1.2 方法
1.2.1 酶分子的序列分析
采用 ProtScale工具(http:∥www.expasy.ch/tools/prot scale.html)中的 Kyte and Doolittle算法对CYP725A4进行疏水性分析.分析图中正值越大表示疏水性越强,而负值越大表示亲水性越强.疏水值介于-0.5~+0.5之间的为两性氨基酸.
分别用工具 HMMTOP(http:∥www.enzim.hu/hmmtop/html/submit.html)和 TMHMM Server v.2.0(http:∥www.cbs.dtu.dk/services/TMHMM/)来在线预测CYP725A4的跨膜区[6-7].
在NCBI上进行protein blast(选择nr数据库)得到相似序列,进行序列比对并构建进化树.
1.2.2 酶分子的三级结构建模与验证
在 expasy 网站(http:∥au.expasy.Org/tools)上选择swiss-model工具的Automated模式进行同源建模[8],用 Pymol(http:∥ pymol.org/)查看结构模型[9].对建模结果用 PROCHECK程序进行合理性验证[10].
2 结果与分析
2.1 CYP725A4序列的生物信息学分析
2.1.1 疏水性分析
CYP725A4疏水性分析的结果如图1所示.由图1可见,29~40区具有很强的疏水性,很可能是CYP725A4的跨膜区.其余区域的疏水性较弱或亲水性较强,可能是折叠区.

图1 CYP725A4的疏水性分析Fig.1 Hydrophobic profile analysis of CYP725A4
2.1.2 跨膜区预测
首先用HMMTOP工具在线进行CYP725A4的跨膜区预测,结果显示CYP725A4在N端有两个跨膜螺旋,分别位于25~42区和57~74区.然后用TMHMM工具在线进行预测,预测结果如图2(a)所示.结果表明,CYP 725A4只有一个跨膜区(20~42),在此之前的区域(1~19)位于膜内侧,之后的区域(43~499)位于膜外侧,与HMMTOP所预测的两个跨膜区有所不同.TMHMM所得结果与图1所示的疏水区分析结果颇为吻合,预示着前40位左右的区域属于跨膜区,在原核宿主中表达时可将其切掉.但为保险起见,也可按照HMMTOP的预测结果尽可能地切掉跨膜端(去掉前74个氨基酸残基甚至更多)以实现可溶表达.

图2 由TMHMM方法预测的蛋白跨膜区Fig.2 Protein transmembrane regions predicted by TMHMM method
2.1.3 进化树构建
在NCBI上检索CYP725A4的相似序列并构建进化树(见图3).由图3可见,CYP725A4与紫杉醇途径上其它P450酶分子的相似性最高.这是因为它们都在执行非常相似的功能——对紫杉烷骨架上的不同位点进行羟基化.它们可能具有十分相似的空间结构.因此,对CYP725A4的结构进行建模也有助于研究该途径上其它P450酶分子的结构和功能.

图3 CYP725A4的相似序列进化树Fig.3 Phylogenetic tree of similar sequences of CYP725A4
2.2 CYP725A4的三级结构建模
在PDB结构数据库中进行CYP725A4同源模板的筛选,发现只有3个蛋白与CYP725A4的同源性超过30%,最高的也只有36%(CYP120A1).
用swiss-model在线进行CYP725A4的结构建模.在Automated模式下将自动选择同源性最高的CYP120A1为模板.图4(a)为swiss-model所建立的CYP725A4天然态三级结构模型图.
用PROCHECK程序对CYP725A4天然态三级结构模型进行Ramachandran分析,见图5(a),图中Ψ表示N-Cα-C-N原子间的二面角,Φ表示C-N-Cα-C原子间的二面角,A、B、L 为最合理区域,a、b、l、p 为较合理区域,~a、~b、~l、~p为次合理区域.由图5(a)可知,有89.1%的氨基酸残基二面角位于合理区域,说明模型具有较好的可靠性.
在P450中,半胱氨酸以十分保守的方式与亚铁血红素(HEM)中的铁元素形成硫醇盐键,从而形成P450的活性中心[11].天然态 CYP725A4共有4个半胱氨酸残基(Cys20、Cys105、Cys328、Cys445),根据前面的跨膜区分析结果,推测Cys20位于跨膜区内,其余3个位于折叠区组成活性中心,负责催化紫杉烷5α位碳氢键的羟基化.

图4 CYP725A4的三级结构建模结果Fig.4 Tertiary structure modeling results of CYP725A4
此外,为构建可溶表达蛋白,可按照前面的预测结果把疏水N端跨膜区切掉.分别考虑切除74个氨基酸残基(74 aa)和42个氨基酸残基(42 aa)两种情况,并按照文献[12]加上小牛P45017α的前8个氨基酸残基(MALLLAVF)从而得到两个新序列,其长度分别为433 aa(记为 CYP725A4-M1)和465 aa(记为CYP725A4-M2).用swiss-model工具的Automated模式对新设计的两个序列进行同源建模,所得结构如图4(b)和(c)所示.结果表明,两种切除方式所得到的蛋白折叠结构基本维持了天然态CYP725A4的高级结构,对蛋白催化活性的影响似乎不大.而CYP725A4-M2比CYP725A4-M1更接近于天然态CYP725A4的结构,可能是由于它被切除的N端长度更短的缘故.
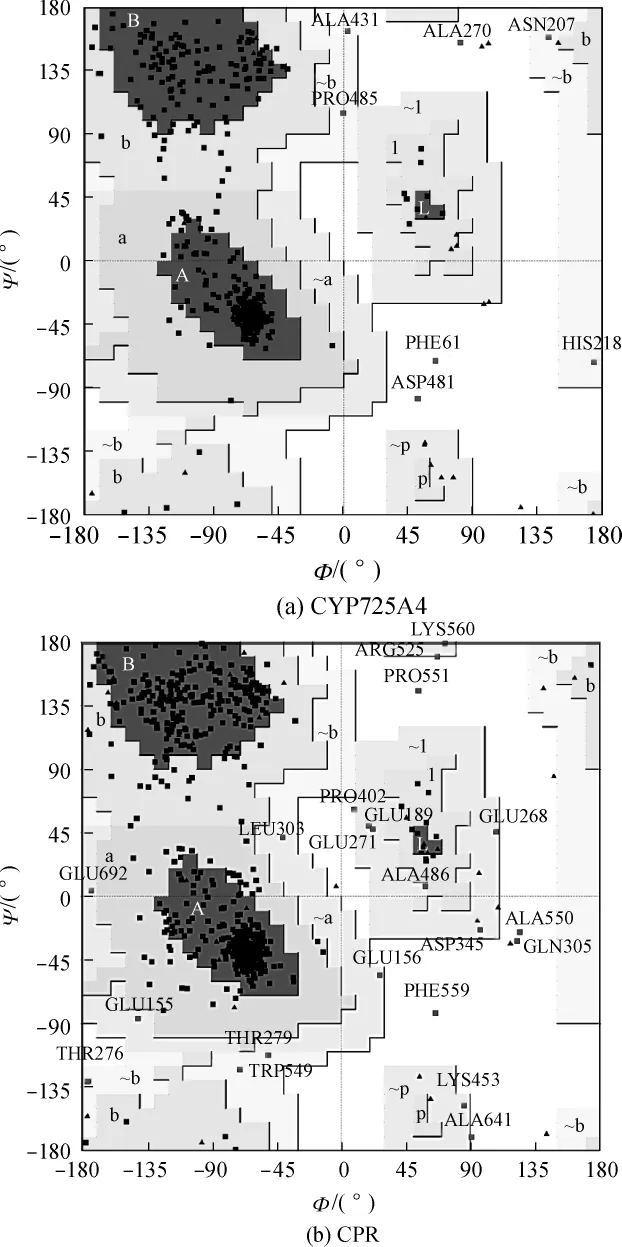
图5 天然态CYP725A4和CPR结构模型的Ramachandran图Fig.5 Ramachandran plots of structure models of native CYP725A4 and CPR
2.3 细胞色素P450还原酶的三级结构建模
由于大肠杆菌缺乏P450蛋白催化所必需的细胞色素P450还原酶(CPR),在表达P450蛋白的同时必须一起表达CPR才能实现功能表达.因此本研究还构建了CPR的结构模型.
首先用TMHMM方法预测了CPR的跨膜区,(见图2(b)).预测结果显示,CPR有两个跨膜区,分别为26~48区和55~74区.其余的区域中,49~54区位于膜内侧,1~25区和75~717区位于膜外侧.参考文献[12],本研究将N端的前74个氨基酸残基切掉并加上小牛P45017α前8个氨基酸残基(MALLLAVF)而构建一个新序列.对CPR原序列(天然序列)和新设计序列用swiss-model分别进行三级结构建模(见图6),并用PROCHECK对天然态结构模型进行验证(见图5(b)).
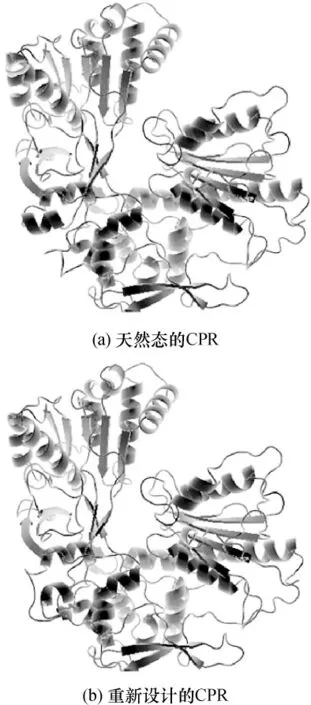
图6 CPR的三级结构建模结果Fig.6 Tertiary structure modeling results of CPR
由图6可见,切除N端跨膜区似乎对CPR的高级结构影响不大.据此可以用重新设计的CPR与上述重新设计的CYP725A4-M1或CYP725A4-M2联合构建一个融合表达蛋白,以实现异源可溶表达.
3 讨论
由于蛋白质的序列、结构以及功能之间的关系极其复杂,目前还没有一种可靠的算法能由序列预测出完全正确的结构.二级结构预测的平均准确度通常在75%~80%之间,而三级结构预测的准确度更低[13].由同源建模方法预测的三级结构与模板的选择有很大的关系[14],通常目标序列与模板之间的同源性越高其预测准确度就越高.遗憾的是,目前已知三级结构的蛋白中与CYP725A4同源性最高的仅为36%.这是由于实验技术难以测定膜蛋白结构而造成PDB数据库中P450结构数据严重不足的缘故.因此,尽管本研究所建立的CYP725A4三级结构模型与实际情况相比有一定的偏差,但在实验难以获取CYP725A4真实结构的情况下,分子建模结果对CYP725A4乃至更多P450蛋白在大肠杆菌等异源宿主中更好地实现功能表达仍然是有指导意义的.后续的工作可在此基础上进行分子改造模拟与实验,以实现蛋白分子的再设计.
此外,在大肠杆菌等原核宿主中进行植物细胞色素P450蛋白表达时需要考虑到宿主自身并没有任何P450蛋白的相关基因,因而缺乏P450蛋白催化所必需的辅助还原酶CPR.为解决这个问题,通常需要实现P450和CPR在大肠杆菌中的共表达.而由于CPR本身也是一个跨膜蛋白,因此应先将其跨膜区切除,或者再加上小牛P45017α的前8个密码子以增强可溶表达效果[12].此外,尽可能地减少5'端和3'端非翻译序列对5'末端的修饰,尽量选择大肠杆菌偏好的密码子,并通过同义突变增加5'端A、T的含量以降低与核糖体结合或翻译时mRNA二级结构的形成,将有助于P450及其CPR更好地实现功能性表达,获取下游的中间产物或终产物.
4 结语
本研究对紫杉醇生物合成途径上的首个P450酶分子CYP725A4(CYP725A4)及其还原酶CPR进行了序列特性分析以及三级结构建模,并提出了实现功能表达的一些策略.在现阶段实验数据缺乏的情况下,这些工作可为CYP725A4等P450蛋白在大肠杆菌等异源宿主中更好地实现功能表达提供指导.
[1] Chau M,Croteau R.Molecular cloning and characterization of a cytochrome P450 taxoid 2α-hydroxylase involved in taxol biosynthesis[J].Archives of Biochemistry and Biophysics,2004,427(1):48-57.
[2] Chau M,Jennewein S,Walker K,et al.Taxol biosynthesis:molecular cloning and characterization of a cytochrome P450 taxoid 7 beta-hydroxylase[J].Biochemistry& Biology,2004,11(5):663-672.
[3] Jennewein S,Croteau R.Taxol:biosynthesis,molecular genetics,and biotechnological applications[J].Applied Microbiology and Biotechnology,2001,57(1/2):13-19.
[4] Jennewein S,Long R M,Williams R M,et al.Cytochrome P450 taxadiene 5α-hydroxylase,a mechanistically unusual monooxygenase catalyzing the first oxygenation step of taxol biosynthesis[J].Biochemistry & Biology,2004,11(3):379-387.
[5] Rontein D,Onillon S,Herbette G,et al.CYP725A4 from yew catalyzes complex structural rearrangement of taxa-4(5),11(12)-diene into the cyclic ether 5(12)-oxa-3(11)-cyclotaxane[J].Journal of Biological Chemistry,2008,283(10):6067-6075.
[6] Tusnady G E,Simon I.The HMMTOP transmembrane topology prediction server[J].Bioinformatics,2001,17(9):849-850.
[7] Chen Y J,Yu P,Luo J C,et al.Secreted protein prediction system combining CJ-SPHMM,TMHMM,and PSORT[J].Mammalian Genome,2003,14(12):859-865.
[8] Schwede T,Kopp J,Guex N,et al.SWISS-MODEL:an automated protein homology-modeling server[J].Nucleic Acids Research,2003,31(13):3381-3385.
[9] DeLano W L,Lam J W.PyMOL:a communications tool for computational models[J].Abstracts of Papers of the American Chemical Society,2005,230(1):1371-1372.
[10] Laskowski R A,Macarthur M W,Moss D S,et al.PROCHECK:a program to check the stereochemical quality of protein structures[J].Journal of Applied Crystallography,1993,26(1):283-291.
[11] Yoshioka S,Takahashi S,Hori H,et al.Proximal cysteine residue is essential for the enzymatic activities of cytochrome P450(cam)[J].European Journal of Biochemistry,2001,268(2):252-259.
[12] Chang M C,Eachus R A,Trieu W,et al.Engineering Escherichia coli for production of functionalized terpenoids using plant P450s[J].Nature Chemical Biology,2007,3(5):274-277.
[13] Floudas C A.Computational methods in protein structure prediction[J].Biotechnology and Bioengineering,2007,97(2):207-213.
[14] 熊盛,王一飞,钱垂文,等.抗HBsAg单链抗体的等电点测定和结构模拟[J].华南理工大学学报:自然科学版,2003,31(11):73-76.Xiong Sheng,Wang Yi-fei,Qian Chui-wen,et al.Isoelectric point determination and structure modeling of a single chain Fv against HBsAg[J].Journal of South China University of Technology:Natural Science Edition,2003,31(11):73-76.
