香港巨牡蛎ISSR-PCR反应体系的建立及优化
2011-01-11黎小正童桂香韦信贤吴祥庆
黎小正, 童桂香, 韦信贤, 吴祥庆, 陈 康
(1. 广西渔业病害防治环境监测和质量检验中心, 广西 南宁 530021; 2. 钦州市钦南区水产畜牧局, 广西 钦州535000)
香港巨牡蛎ISSR-PCR反应体系的建立及优化
黎小正1, 童桂香1, 韦信贤1, 吴祥庆1, 陈 康2
(1. 广西渔业病害防治环境监测和质量检验中心, 广西 南宁 530021; 2. 钦州市钦南区水产畜牧局, 广西 钦州535000)
以香港巨牡蛎(Crassostrea hongkongensis)基因组DNA为模板, 采用正交设计及单因素比较试验,分别对Mg2+、dNTPs、引物、TaqDNA聚合酶和DNA模板浓度的PCR原料进行优化, 并通过温度梯度PCR, 筛选适宜的退火温度。确立了香港巨牡蛎的最适ISSR-PCR反应体系: 25 μL反应体系中含PCR 1×Buffer, 2.5 mmol/L Mg2+、0.2 mmol/L dNTPs、0.2 μmol/L 引物、1.2 UTaqDNA 聚合酶、20 ng DNA模板。反应程序为: 94℃预变性5 min; 94℃变性1 min, 48~54℃(随引物而确定)退火1 min, 72℃延伸1.5 min, 35个循环; 72℃延伸10 min。利用所建立的 ISSR-PCR反应体系对香港巨牡蛎基因组DNA进行扩增, 获得了清晰、重复性好、多态性高的DNA谱带, 为进一步利用ISSR分子标记研究香港巨牡蛎的遗传多样性奠定了基础。
香港巨牡蛎(Crassostrea hongkongensis); ISSR-PCR; 体系优化
香港巨牡蛎 (Crassostrea hongkongensis), 属于双壳纲(Bivalvia), 珍珠贝目(Pteriodae), 牡蛎科(Ostreidae)[1], 俗称大蚝, 是暖水性双壳类软体动物,以滤食海水浮游生物为主, 其肉质鲜美、营养丰富,素有“海底牛奶”之称, 是中国南方沿海主要的经济贝类和出口海产品, 也是作为海洋重金属等污染的重要指示生物[2]。钦州湾海岸曲折、咸淡水交汇、滩涂广阔、十分有利于牡蛎的繁殖和生长, 茅岭海域也因产上乘品质的牡蛎而被农业部授予“中国牡蛎之乡”的荣誉称号。钦州市的牡蛎养殖逐年增多, 苗种采集及成品产量大幅增长, 随着人工开发和自然环境的变化, 香港巨牡蛎的生长发生了一些变化, 甚至出现种质退化和变异现象[3]。因此, 研究钦州香港巨牡蛎种质资源的遗传多样性和群体遗传结构变异,对充分开发利用及保护资源具有重要意义。目前, 对于研究牡蛎遗传多样性的分子标记方法主要有RAPD分析、线粒体DNA基因片段分析、微卫星标记及同工酶带型分析等[4-6]。简单重复序列间扩增(inter simple sequence repeat, ISSR )是近年来在微卫星技术的基础上发展起来的一种新型分子标记技术,具有操作简单、重复性好, 多态性高、适合大样本基因组DNA的检测等优点[7], 且ISSR为显性标记, 具有孟德尔遗传特征, 因此被广泛地应用于各种生物的遗传多样性分析、品种和种质鉴定以及物种和种群亲缘关系等方面研究。但由于 ISSR标记是基于PCR的反应, 其扩增结果易受Mg2+、TaqDNA聚合酶、dNTPs、引物、模板 DNA等因素的影响, 且最佳扩增条件因被测物种不同而各有不同。本研究采用正交设计及单因素比较试验对香港巨牡蛎ISSR-PCR反应体系进行优化, 建立香港巨牡蛎的最适ISSR-PCR反应体系, 为钦州牡蛎主要养殖品种香港巨牡蛎的种质资源鉴定及遗传多样性分析提供分子标记方法。
1 材料与方法
1.1 材料
香港巨牡蛎采自钦州茅尾海域, 活体低温运回实验室解剖取闭壳肌, -80℃保存。
1.2 主要试剂
ISSR引物参考加拿大哥伦比亚大学(University of British Columbia, UBC)公布的 100条引物序列,参照文献[8-10]的研究结果选取其中的 25条引物进行试验, 编号分别为 810, 811、812、813、814、815、822、823、824、828、829、830、835、840、841、842、843、844、845、852、853、854、855、856、857, 各引物均为二核苷酸重复碱基。PCRPremix Taq、Mg2+、dNTPs、TaqDNA聚合酶购自大连宝生物工程有限公司。
1.3 DNA提取
每个牡蛎样取闭壳肌50 mg剪碎, 加600 µL细胞裂解液振荡混匀; 加蛋白酶 K消化过夜, 依次用酚、氯仿抽提, 最后加无水乙醇沉淀 DNA, 干燥后加超纯水100 µL溶解。紫外分光光度计测DNA浓度和纯度, 稀释成20 µg/mL, -20℃保存备用。
1.4 引物初次筛选
用PCRPremix Taq反应液将25条引物初步筛选,参照相关文献[11], ISSR-PCR反应体系为: 25 µL中含 2×PCR Mix 12.5 µL, 引物(10 µmol/L)1 µL, 模板20 ng, 加水补足25 µL。PCR反应程序为94℃预变性5 min; 94℃变性1 min, 52℃退火1 min, 72℃延伸1.5 min, 进行35个循环; 72℃延伸10 min。15 g/L琼脂糖凝胶电泳检测结果, 选取扩增条带清晰、多态性好的引物作下一步优化实验。从25条引物中筛选,扩增较理想的是823(TCT CTC TCT CTC TCT CC)。
1.5 筛选引物最佳退火温度的确定
将扩增条带清晰的引物823作温度梯度PCR试验, 反应体系参照1.4, 退火温度分别设为48、50、52、54、56、58℃, 电泳检测结果, 确定该引物的最佳退火温度。经检测, 823的最佳退火温度为50℃。
1.6 ISSR-PCR反应体系的优化
参照已发表文献中的ISSR- PCR反应体系[12-13],用引物823进行香港巨牡蛎ISSR- PCR体系的优化,对 Mg2+浓度(1.0、1.5、2.0、2.5、3.0 mmol/L)、dNTPs(0.1、0.14、0.20、0.26、0.30 mmol/L)、引物(0.1、0.14、0.20、0.26、0.30 µmol/L)、Taq酶(0.5、1.0、1.5、2.0、2.5 U)、模板(10、20、30、40、50 ng)5个因素水平用正交设计L25(55)进行试验(表1), 设置25个不同组合, 反应体系均为25 µL, 按表1各反应因子终浓度加样; PCR反应程序参照 1.4, 结束后经15 g/L的琼脂糖凝胶电泳观察条带数目及清晰度得到理想的浓度组合, 再逐一进行单个因素试验。
1.7 各单个因子浓度梯度试验
在利用引物 823确定了最佳组合的基础上, 分别对5种因子的不同浓度水平进行ISSR-PCR, 设置Mg2+浓度分别为 0.5、1.0、1.5、2.0、2.5、3.0、3.5、4.0、4.5 mmol/L 9个梯度; dNTPs浓度分别为0.10、0.12、0.14、0.16、0.18、0.20、0.22、0.24、0.26、0.28 mmol/L 10个梯度; 引物浓度分别为0.10、0.12、0.14、0.16、0.18、0.20、0.22、0.24、0.26、0.28、0.30 µmol/L 11 个梯度;Taq酶分别为 0.5、0.6、0.8、1.0、1.2、1.4、1.6、1.8、2.0、2.2、2.4、2.6、2.8U 13个梯度; 模板DNA含量分别为10、15、20、25、30、35、40、45、50、55、60 ng 共11个梯度。比较各因素在不同浓度时的扩增效果, 获得最佳的ISSR-PCR反应体系。
1.8 其余引物筛选
取香港巨牡蛎DNA样品用优化后的反应体系对其余24条引物进行筛选, 并通过温度梯度PCR(退火温度为48、50、52、54、56、58℃)探索每条引物的最佳退火温度, 最终筛选到扩增条带清晰, 多态性好的引物及其适宜的退火温度。
1.9 优化体系的应用
随机选取 24份香港巨牡蛎闭壳肌提取基因组DNA, 利用优化建立的体系进行 ISSR-PCR, 电泳检测结果, 验证 ISSR-PCR体系对香港巨牡蛎的适用性。
2 试验结果
2.1 初次引物筛选结果
将25条引物初步筛选, 结果引物823扩增条带较理想(图1), 可以扩增到5条较清晰、便于观察的条带, 因此, 选择823进行ISSR-PCR反应体系的优化。
2.2 引物823的最佳退火温度
经温度梯度 PCR试验, 823的最佳退火温度是50℃。
2.3 ISSR- PCR正交试验结果
Mg2+、dNTPs、引物、DNA模板浓度及TaqDNA聚合酶量等各反应因素的综合作用影响PCR的扩增,正交设计的ISSR-PCR试验结果(图2)显示各组合的扩增结果差异明显, 通过比较可筛选到较理想的各反应因子浓度组合。电泳显示第8组合效果最好, 其TaqDNA聚合酶量高, 扩增条带清晰; 第1、4、5、22、23号组合都没有扩增, 2、3、9号显示微弱扩增
带。因此选择 8号组合(含有 1.5 mmol/L Mg2+、0.20 μmol/L dNTPs、0.26 μmol/L引物、2.5 UTaqDNA聚合酶、10 ng DNA模板)为香港巨牡蛎ISSR-PCR的较适反应体系组合。

表1 香港巨牡蛎ISSR-PCR反应L25(55)正交试验设计Tab. 1 Orthogonal design L25(55)of ISSR-PCR in Crassostrea hongkongensis

图1 25条ISSR引物的PCR结果Fig. 1 ISSR-PCR results of 25 primers

图2 香港巨牡蛎ISSR-PCR反应L25(55)正交试验结果Fig. 2 Orthogonal design L25(55)results of ISSR-PCR in Crassostrea hongkongensis
2.4 各单因子对扩增结果影响
2.4.1 Mg2+浓度
当 Mg2+终浓度依次为 0.5、1.0、1.5、2.0、2.5、3.0、3.5、4.0、4.5 mmol/L时均有扩增(图 3), 浓度为 0.5mmol/L时只有两条扩增带, 高于 2.5 mmol/L时大片段的非特异性带较多, 而浓度为 2.5 mmol/L时扩增条带数目适中、清晰, 因此确定 Mg2+的终浓度为2.5mmol/L。
2.4.2 dNTPs浓度
dNTPs浓度分别为 0.1、0.12、0.16、0.18、0.20、0.22、0.24 mmol/L时都有扩增, 浓度为0.26 mmol/L只有微弱的扩增带, 浓度为 0.14 mmol/L 和0.28 mmol/L无扩增带(图4)。由图可知, dNTPs终浓度为0.20 mmol/L时效果最好。

图3 Mg2+浓度对ISSR-PCR影响Fig. 3 Effect of different concentrations of Mg2+
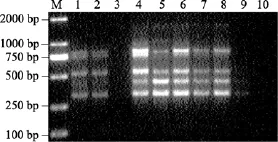
图4 dNTPs浓度对ISSR-PCR影响Fig. 4 Effect of different concentrations of dNTPs
2.4.3 引物浓度
引物浓度在 0.12、0.14、0.16、0.18、0.20、0.22、0.24、0.26、0.28、0.30 µmol/L 10个梯度时都有扩增,引物浓度过低(0.1 µmol/L)无扩增产物, 引物浓度过高(0.28、0.30 µmol/L)扩增效率低, 引物浓度为0.20 µmol/L时可得到较理想的结果(图5)。
2.4.4 Taq酶
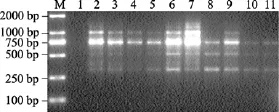
图5 引物浓度对ISSR-PCR影响Fig. 5 Effect of different concentrations of primer
TaqDNA聚合酶共设置了0.5、0.6、0.8、1.0、1.2、1.4、1.6、1.8、2.0、2.2、2.4、2.6、2.8 U 的 13个梯度, 每个浓度均可得到较清晰的扩增产物, 但低浓度时效率相对低, 扩增条带弱, 随着酶的增加,扩增效率提高, 但酶的浓度过高使扩增的检测结果背景较重, 增加了成本, 当TaqDNA聚合酶为1.2 U时即可获得较好效果(图6)。
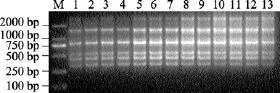
图6 Taq DNA聚合酶对ISSR-PCR影响Fig. 6 Effect of different concentrations of Taq DNA polymerase
2.4.5 模板浓度影响
DNA 模板分别为 10、15、20、25、30、35、40、45、50、55、60 ng 时均有扩增(图 7), 低浓度 DNA(10、15 ng)时扩增效率很低, 不易观察; 20 ng DNA即可得到较好效果, 将 DNA浓度增大, 其扩增产物无明显差异, 因此, 本反应体系应加入20ng模板。
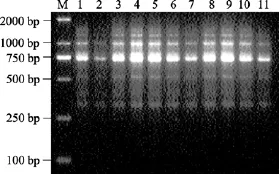
图7 模板浓度对ISSR-PCR影响Fig. 7 Effect of different concentrations of DNA
2.5 香港巨牡蛎ISSR-PCR的最佳反应体系
经过正交试验设计及各单因子浓度探索, 确定香港巨牡蛎 ISSR-PCR分析技术的最佳反应体系为:25 µL的反应体系含有Mg2+浓度2.5 mmol/L、dNTPs浓度0.20 mmol/L、引物浓度0.20 µmol/L、TaqDNA聚合酶1.2 U、模板DNA 20 ng; 扩增程序为: 94℃5 min; 94℃ 1 min, 退火(48~56℃)1 min, 72℃延伸l.5 min, 35个循环; 72℃延伸10 min, 4℃结束反应。
2.6 引物筛选结果
按ISSR-PCR最佳反应体系对引物进行筛选, 结果从25条引物中筛选到9条扩增条带清晰、重复性较好, 显带强的引物(表 2)。根据各引物的解链温度(Tm), 对每一个引物在(Tm±5)℃的范围内进行温度梯度 PCR, 引物的退火温度对扩增结果影响不大,优化后得到9个引物的最佳退火温度值, 因Tm值不同, 其最佳退火温度也不尽相同, 在48~54℃之间。
2.7 优化体系的应用
利用优化建立的体系将筛选的引物应用于香港巨牡蛎基因组ISSR-PCR, 验证该体系的适用性。图8是其中一条引物对随机选取的24份香港巨牡蛎闭壳肌DNA扩增结果, 电泳显示该反应具有较清晰的扩增带。
3 讨论
ISSR-PCR对不同引物、不同材料的扩增条件存在较大差异, 并受诸多因素影响, 如 DNA模板的浓度与纯度、dNTPs、Mg2+的浓度、TaqDNA聚合酶用量、退火温度和反应程序等[14], 并且各反应因子的相互影响直接关系到ISSR-PCR结果。正交试验是研究多因素多水平的一种方法, 具有均衡分散和整齐可比的特点, 在ISSR-PCR反应的各因素评价中正交试验显示了其优越性[15]。本试验以香港巨牡蛎基因组 DNA为模板, 利用正交试验设计方法, 得到了香港巨牡蛎的 ISSR-PCR较佳反应体系组合; 并以此为基础, 对各反应因子逐个优化, 探索各因子的最佳反应浓度。
影响ISSR-PCR反应的因素多, Mg2+浓度影响反应的特异性和扩增效率, 通常 Mg2+终浓度在 1.0~2.5 mmol/L之间可得到较好的结果, 高浓度的Mg2+能提高扩增效率, 但是 Mg2+过高电泳带会有明显的背景且会降低反应的特异性[16], 因此, 在兼顾扩增效率情况下应适当降低 Mg2+浓度。本试验中 Mg2+终浓度设置了 0.5~4.5 mmol/L的梯度, 各浓度均有扩增带, 但浓度为 0.5 mmol/L时只有两条扩增带,显然低浓度 Mg2+影响了整个反应的扩增效果, 而高于2.5 mmol/L时大片段的非特异性带较多, Mg2+浓度为2.5 mmol/L时扩增条带数目适中、清晰。dNTPs浓度一般在0.02~0.20mmol/L之间, 其浓度过高会导致聚合酶错误的掺入, 浓度过低又会影响合成效率,甚至会因 dNTPs过早耗尽而使产物单链化, 影响扩增效果; 本试验中dNTPs浓度为0.2 mmol/L时效果最佳, dNTPs浓度过高反应无扩增, 可能是由于高浓度dNTPs分子中的磷酸基团能与Mg2+结合, 使有效反应的Mg2+减少, 反应受到抑制。引物浓度对 PCR反应起重要作用, 浓度过低时结合几率小而扩增效率降低, 浓度过高可能导致错配, 特异性降低, 并可能导致引物二聚体形成; 本试验中引物 0.1 µmol/L无扩增产物, 表明过低的引物浓度无法得到扩增产物, 而过高的引物浓度(0.28、0.30 µmol/L)时扩增产物带不清晰、效率低, 可能是由于高浓度的引物抑制了反应体系中其他因子的有效作用, 当引物浓度为0.20 µmol/L时可得到较理想的结果。高浓度TaqDNA聚合酶增加成本和非特异扩增产物, 低浓度可能使酶催化能力不够强而影响产物的合成效率,Taq酶的用量一般在0.5~5 U之间, 此试验中随着Taq酶量的增加, 扩增效率提高的同时伴有带型变得模糊、大片段的非特异性产物增多, 故 25 µL体系中使用1.2 U即可。ISSR-PCR反应对模板浓度要求不严格[17],但是浓度过高和过低都会影响到扩增反应的效率和特异性。从本试验模板浓度的影响结果看, DNA浓度对反应的影响较其他因子小, 在10~60 ng均有扩增,低浓度DNA影响PCR效率, 扩增带少, 随着DNA浓度的增加, PCR结果差异不大, 25 µL反应体系中DNA含量20 ng即可获得较好效果。此外, 本实验根据筛选到的各引物的解链温度(Tm), 在(Tm±5)℃的范围内对各引物的最佳退火温度进行确定, 因各引物的Tm值不同, 其最佳退火温度也不尽相同, 在48~54℃之间, 一般在52℃即可获得较好的效果。
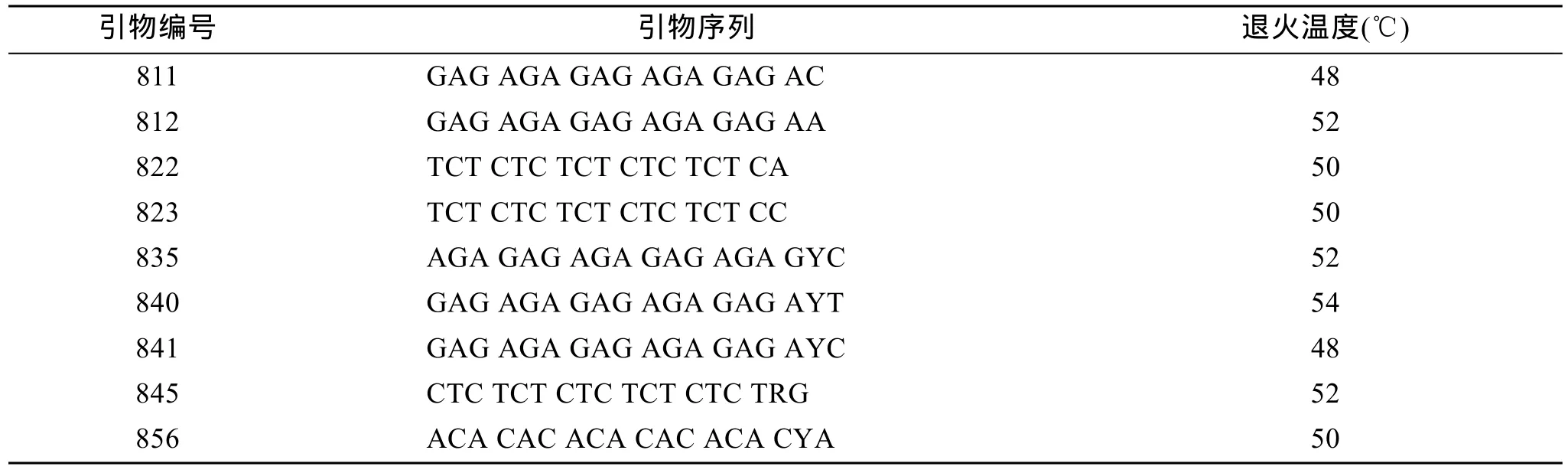
表2 从25条引物中筛选的9条ISSR-PCR引物Tab. 2 Nine satisfactory ISSR-PCR primers to Crassostrea hongkongensis selected out of 25 candidates
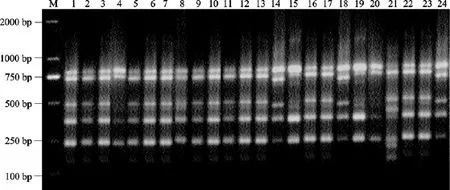
图8 引物823在优化后的体系下对24份香港巨牡蛎DNA样品的扩增结果Fig. 8 ISSR-PCR results of 24 Crassostrea hongkongensis DNA amplified by the optimized reaction system
综合以上试验, 筛选出适合香港巨牡蛎的ISSR-PCR最佳反应体系为: 25 μL的反应体积中含10×Buffer 2.5 µL, 2.5 mmol/L Mg2+、0.20 mmol/L dNTPs、0. 20 μmol/L引物、1.2 UTaqDNA聚合酶、20 ng DNA模板; 反应程序为: 94℃预变性 5 min;94℃变性 1 min, 48~54℃退火 1 min, 72℃延伸 1.5 min, 35个循环; 72℃延伸 10 min, 4℃结束反应。试验表明, 利用所建立的 ISSR反应体系对香港巨牡蛎基因组DNA进行PCR扩增, 可获得清晰、重复性好、多态性高的 DNA谱带, 为进一步利用ISSR分子标记技术进行香港巨牡蛎遗传多样性分析和种质资源鉴定等研究奠定了基础。
[1]王海艳, 郭希明, 刘晓, 等. 中国近海“近江牡蛎”的分类和订名[J]. 海洋科学, 2007, 31(9): 85-86.
[2]贾晓平, 蔡文贵, 林钦, 等. 广东沿海香港巨牡蛎体砷含量水平、地理分布特点和变化趋势[J]. 中国水产科学, 1999, 6 (2): 97-100.
[3]李咏梅, 陈秀荔, 赵永贞, 等. 钦州湾牡蛎线粒体16S rRNA和 COⅠ基因片段的序列变异分析[J]. 广东海洋大学学报, 2009, 29(3): 11-18.
[4]Carlsson J, Morrison C L, Reece K S. Wild and aquaculture populations of the eastern oyster compared using microsatellites[J]. J Hered, 2006, 97(6): 595-598.
[5]Taris N, Boudry P, Bonhomme F, et al. Mitochondrial and nuclear DNA analysis of genetic heterogeneity among recruitment cohorts of the European flat oyster Ostrea edulis[J]. Bio Bull, 2009, 217(3): 233-241.
[6]黄荣莲, 杜晓东, 张运银. 香港巨牡蛎 3个种群生化遗传变异的研究[J]. 湛江海洋大学学报, 2006, 26(3): 3-7.
[7]黎小正, 童桂香, 韦信贤, 等.ISSR-PCR技术及其在水产生物遗传多样性研究中的应用[J]. 西南农业学报, 2010, 23(3): 956-964.
[8]Bouilly K, Chaves R, Leitao A,et al.Chromosomal organization of simple sequence repeats in the Pacific oyster(Crassostrea gigas): (GGAT)(4), (GT)(7) and (TA)(10)chromosome patterns[J]. J Genet, 2008, 87(2): 119-125.
[9]姜因萍, 何毛贤, 黄良民, 等. 两个大珠母贝群体遗传多样性的ISSR分析[J]. 热带海洋学报, 2008, 27(3):61-65.
[10]王婷, 丁君, 丁佳平, 等. 利用ISSR技术研究虾夷扇贝不同地理种群的遗传多样性及其分化[J]. 烟台大学学报(自然科学与工程版), 2009, 22(1): 35-41.
[11]孙始威, 孙振兴, 陈燕妮, 等. 扁玉螺 ISSR-PCR 反应体系的建立及其优化[J]. 湖北农业科学, 2008,47(5): 575-577.
[12]张波, 孟学平, 陈建安. 西施舌 ISSR-PCR扩增条件优化[J]. 海洋湖沼通报, 2008(1): 46-53.
[13]张志伟, 陈大鹏, 张曹进, 等. 半滑舌鳎基因组的提取及ISSR反应体系的建立[J]. 生物技术, 2006, 16(6):44-47.
[14]余艳, 陈海山, 葛学军. 简单重复序列区间(ISSR)引物反应条件优化与筛选[J]. 热带亚热带植物学报,2003, 11(1): 15-19.
[15]郁建锋, 鲍峰, 韩晓磊, 等. 松江鲈鱼 ISSR-PCR 反应体系的建立及优化研究[J]. 安徽农业科学, 2008,36(33): 14420-14421.
[16]Abbot P. Individual and Population variation in invertebrates revealed by Inter-simple sequence repeats(ISSRs) [J]. Insect Science, 2001, 1(8): 1-3.
[17]杨浩, 陈喜, 陈欣, 等. 黄鳝 ISSR-PCR 反应体系的建立及条件优化[J]. 生物技术通报, 2009, (7): 113-116.
Establishment and optimization of an ISSR-PCR reaction system forCrassostrea hongkongensis
LI Xiao-zheng1, TONG Gui-xiang1, WEI Xin-xian1, WU Xiang-qing1, Chen Kang2
(1. Center of Disease Prevention and Cure, Environmental Monitoring and Quality Monitoring of Guangxi Fishery,Nanning 530021, China; 2. Qinnan Fisheries and Husbandry Bureau of Qinzhou, Qinzhou 535000,China)
Dec., 23, 2010
Crassostrea hongkongensis; ISSR-PCR; reaction system optimization
To establish an ISSR-PCR reaction system inCrassostrea hongkongensis, the factors influencing ISSR-PCR system were explored with the orthogonal design and main parameters comparison test, based on the genomic DNA ofC. hongkongensis. The optimized ISSR-PCR system (25 μL) was determined as follows:1×Buffer, 2.5 mmol/L Mg2+, 0.2 mmol/L dNTPs, 0. 2 μmol/L primer, 1.2 UTaqDNA polymerase, and 20 ng DNA template. The reaction program included an initial denaturation for 5 minutes at 94℃, 35 cycles of denaturation for 1 minutes at 94℃, annealing for 1 minutes at 48~54℃, extension for 1.5 minutes at 72℃, and a final extension of 10 minutes at 72℃. The clear, reproducible, polymorphic products were obtained by the established ISSR-PCR reaction system. The results lay a foundation for the analysis of genetic diversity ofC.hongkongensisby ISSR marker.
S968
A
1000-3096(2011)08-0018-07
2010-12-23;
2011-03-15
广西科技攻关项目(0992015-1)
黎小正(1962-), 男, 广东阳春人, 高级工程师, 硕士, 主要从事水产品的检验检疫和渔业环境保护等方面的研究, E-mail:lixz@tom.com; 韦信贤, 通讯作者, E-mail: Byang15@126.com
梁德海)
