枯草芽胞杆菌基因组混组方法
2010-10-11杨俊杰范文超肖晗关春鸿曹传增邵海峰姜卫红杨晟
杨俊杰,范文超,肖晗,关春鸿,曹传增,,邵海峰,姜卫红,杨晟,
1 中国科学院上海生命科学研究院植物生理生态研究所 合成生物学重点实验室,上海 200032
2 上海工业生物技术研发中心,上海 201201
3 中国科学院上海生命科学研究院湖州工业生物技术中心,湖州 313000
4 浙江顺风海德尔有限公司,东阳 322100
细胞工厂的构建技术
枯草芽胞杆菌基因组混组方法
杨俊杰1,2,范文超3,肖晗1,关春鸿1,曹传增1,3,邵海峰4,姜卫红1,杨晟1,2,3
1 中国科学院上海生命科学研究院植物生理生态研究所 合成生物学重点实验室,上海 200032
2 上海工业生物技术研发中心,上海 201201
3 中国科学院上海生命科学研究院湖州工业生物技术中心,湖州 313000
4 浙江顺风海德尔有限公司,东阳 322100
基因组混组作为一种育种方法,通过循环原生质体融合等手段,使得不同菌株来源的基因组能够得到充分重组,增加将正向突变整合到同一重组子中的机会。使用4株带有4种不同标记的枯草芽胞杆菌亲本为初始菌株,通过循环转化、循环转导或循环原生质体融合的手段进行基因组混组,统计后代中非亲本类型占整个群体的比例,以衡量基因组混组的效果。分别经过 5轮循环原生质体融合、循环转化或者循环转导,结果显示,重组程度较高者在后代群体中的比例较低,带有4种标记的后代未出现,带有3种标记的后代最高分别为4.53×10−4、1.64×10−4、4.47×10−3,明显低于文献报道的天蓝色链霉菌中同样实验的结果:带4种和3种标记的后代分别占2.5%、17%。对比上述实验的结果和文献报道的天蓝色链霉菌、乳杆菌基因组混组的结果,并结合计算机模拟循环融合过程,分析后认为:要达到较充分的基因组混组,需要有能够实现微生物细胞间高频重组的操作技术作为基础,重组频率应该不低于10−3~10−2数量级。
基因组混组,枯草芽胞杆菌,分子育种,原生质体融合,转化,转导,计算机模拟
Abstract:Genome shuffling methods were explored forBacillus subtilisstrain molecular breeding. Recycling protoplast fusion,recycling transformation and recycling universal transduction were used for genome shuffling inB. subtilis. Four strains with different nutrition-deficiency markers were used as initial strains. After five rounds protoplast fusion, transformation or transduction,the descendant with 4 markers had not been detected, and the rate of descendant with 3 markers were 4.53×10−4, 1.64×10−4,4.47×10−3, respectively. A computer program was made to simulate the recycling fusion process. Based on simulation result andcomparing the genome shuffling result ofB. subtilisin this experiment and that ofStreptomyces coelicolorreported in references,effective genome shuffling needs a high recombination rate of at least between 10−3and 10−2.
Keywords:genome shuffling,Bacillus subtilis, molecular breeding, protoplast fusion, transformation, transduction,in silicosimulation
优良菌种选育是发酵工程的核心技术。传统的手段有诱变育种和杂交育种。常规的诱变育种方法是以紫外线、辐射或化学诱变剂处理微生物,产生大量突变,再以所需指标进行筛选。由于负突变的比例非常高,一般要筛选 105以上突变体才能得到一个正突变[1]。经过长期诱变获得的菌株在特定性状有所改善的同时,一般积累很多有害突变,比如生长条件要求变苛刻、生存能力变差、细胞变“脆弱”等。杂交育种选用已知性状的供体和受体菌种作为亲本,结果大多可以获得优于亲本菌株的后代菌株。利用杂交育种往往还可消除诱变获得的菌株所带的缺陷[1]。
基因组混组 (Genome shuf fl ing) 是在杂交育种的基础上产生的新育种方法。首先通过经典的诱变育种等方法获得多个得到改良的菌株,而后通过循环原生质体融合等手段,使得不同菌株来源的基因组能够得到充分重组,增加将正向突变整合到同一重组子中的机会,同时,积累的有害突变可被其他菌株的野生型序列所代替。希望最终可以获得一批性能改良的重组子。这种思路最初是从 DNA 混组(DNA shuf fl ing) 中的退火步骤得到启发,对原生质体融合育种技术作了改进,将融合后的菌株不经筛选就进行下一轮融合,经过这样反复多次的操作以后才进行筛选,从而使得再生菌落的基因组重组程度大幅度提高。
基因组混组的用于微生物育种过程,其具体操作过程分为 3个步骤[2]。第一步是建立用于混组的初始群体。如果初始群体不好,那么无论后面的步骤做得如何,最终也将难以获得具有预期性状的后代。初始群体的获得,可以使用传统育种的方法,比如通过传统诱变筛选等技术获得一批性状有所改善的初始菌株。野生型菌株或者代谢工程改造获得的菌株,也可能作为混组初始群体的成员[3]。第二步,使用原生质体融合等技术手段,使初始群体中多个亲本基因组之间发生充分的交换和重组。常规单轮原生质体融合用于 2个亲本间的基因组交换重组较为有效,但是融合过程中多个细胞碰撞到一起融合的概率较低。为了实现多亲本基因组之间的交换重组,引入了循环原生质体融合的手段。即多个初始群体中多种类型的亲本细胞同时制备原生质体,混合后进行一轮融合;而后收集融合后获得的后代再生细胞,作为一个群体进行培养。而后制备原生质体,进行新一轮融合。融合过程重复多轮。第三步,使用特定的筛选方法将具有期望性状的后代细胞筛选出来。这一步需要根据育种具体目的采取相应的方案。循环融合获得的后代细胞库可能非常大,此时需要有相应的高通量筛选手段以便从中筛选到具有更好性状者[1]。
2002年Zhang等首次应用基因组混组技术提高弗氏链霉菌Streptomyces fradiae的泰乐菌素产量。经1次诱变育种加上2轮基因组混组,历时1年,得到了2株高产菌株GS1和GS2,其生产能力和通过传统诱变技术获得的高产菌 SF21的生产能力相当。而SF21的获得历经20轮诱变、历时20年[4]。同一年,Patnaik等通过基因组混组提高乳杆菌Lactobacillussp.耐酸性,经5轮循环融合后,筛选得到的新菌株可以在出发株不能生长的酸性环境pH 3.5条件下生长。同时,在pH 4.0的条件下该菌株的乳酸产量比野生型菌株提高了3倍[5]。如文献[2]所综述,近几年来又有了较多的关于此种技术应用的报道。
基因组混组在芽胞杆菌中的应用有以下一些报道。梁惠仪等以具有纤溶酶活性的枯草芽胞杆菌DC-12为初始菌株,首先通过进行紫外诱变和亚硝酸诱变。而后选取 4株诱变菌株作为直接亲本,采用电融合的方法进行 2次多亲本的基因组混组,共筛选出 2株酶活大大提高并能稳定遗传的菌株,酶活提高了 4~5倍[6]。陈亮等首先对枯草芽胞杆菌BS14进行诱变,而后使用获得3株菌UV22-5、HN8-7和HN18-3为初始菌株进行了3轮基因组混组,获得了1株稳定菌株F35。F35对真菌尖孢镰刀菌甜瓜专化型Fusarium oxysporumf. sp.melonis的抗性比 HN8-7和 BS14分别提高了 34.52%和65.48%[7]。
在芽胞杆菌中,尚没有报道对于多轮融合后基因组混组的效果进行较系统的研究。本研究以枯草芽胞杆菌为材料,研究了多轮融合过程中基因组混组程度的改变。除此之外,也将循环转化和循环转导这2种手段引入基因组混组的工作,探索了多轮转化或转导过程中,基因组混组程度的改变。根据这些实验结果以及通过计算机模拟循环融合过程,推断要达到高效的基因组混组,需要有能够实现微生物细胞以较高频率获得重组的操作技术作为基础。
1 材料与方法
1.1 材料与试剂
培养基使用 LB、TB、SOC培养基。抗生素浓度分别为氨苄青霉素100 mg/L、壮观霉素50 mg/L、卡那霉素50 mg/L、氯霉素5 mg/L。所用的其他材料为常规材料。
1.2 菌株、质粒和噬菌体
本研究中构建和使用的质粒见表1。
构建的枯草芽胞杆菌菌株和噬菌体见表 2。本研究中部分菌株构建过程如图1所示,具体如下:

表1 本研究中使用或构建的质粒Table 1 Plasmids used in this study

表2 本研究中使用或构建的菌株和噬菌体株Table 2 Strains used in this study
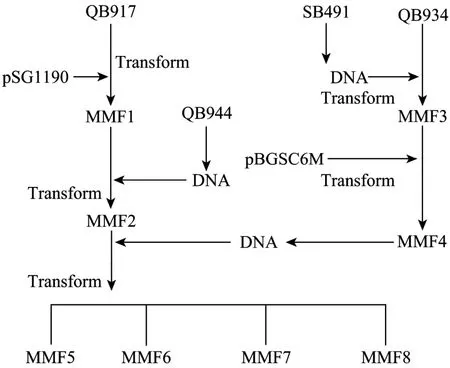
图1 本研究中枯草芽胞杆菌菌株构建过程Fig.1 Constructinon ofBacillus subtilisstrains.
用整合载体pSG1190转化QB917 (hisA1,thr5,trpC2),在 QB917基因组上的 amyE基因的位置上插入一个壮观霉素抗性基因,得到 MMF1(hisA1,thr5,trpC2,SpcR)。以 QB944(purA16,cysA14,trpC2)作为 DNA供体,MMF1作为受体,获得 MMF2(purA16,thr5,trpC2,SpcR)。
以原养型 SB491作为 DNA供体,以 QB934(tre12,metC3,glyB133,trpC2) 作为受体菌,转化得到 MMF3 (metC3)。用 PCR方法把 MMF3中的metC3基因紧邻下游的基因扩增出来,克隆到整合载体pBGSC6中,得到pBGSC6M。用pBGSC6M转化MMF3,得到MMF4 (metC3,CmR)。以 MMF4作为DNA供体,MMF2作为受体菌,分别以不同条件筛选转化子,并对获得的转化子进行鉴定。最终获得 4株菌株,分别是:MMF5 (purA16,thr5,trpC2),MMF6 (purA16,metC3,trpC2),MMF7 (thr5,metC3,trpC2),MMF8 (purA16,thr5,metC3)。
1.3 循环融合
取MMF5、MMF6、MMF7、MMF8的过夜培养物,等比例混合作为初始群体,计作为F0。
将F0群体制备原生质体,加入PEG 以介导融合,涂布于非选择性的再生平板。将再生平板上获得的全部菌落以无菌水洗下,获得的菌体悬液为F1。同样的,以F1群体制备原生质体并进行融合,获得的下一代群体定义为F2。以后各代依次为F3、F4、F5。具体过程如图2所示。
取各代梯度稀释后涂布于添加不同氨基酸或碱基的基本培养基,计数各代中各种基因型细胞的数目并计算它们在群体中所占的比例。
原生质体的制备和PEG介导的原生质体融合的具体实验方法参照文献[13]报道,高渗溶液使用SMMP + BSA,再生培养基使用DM3培养基。
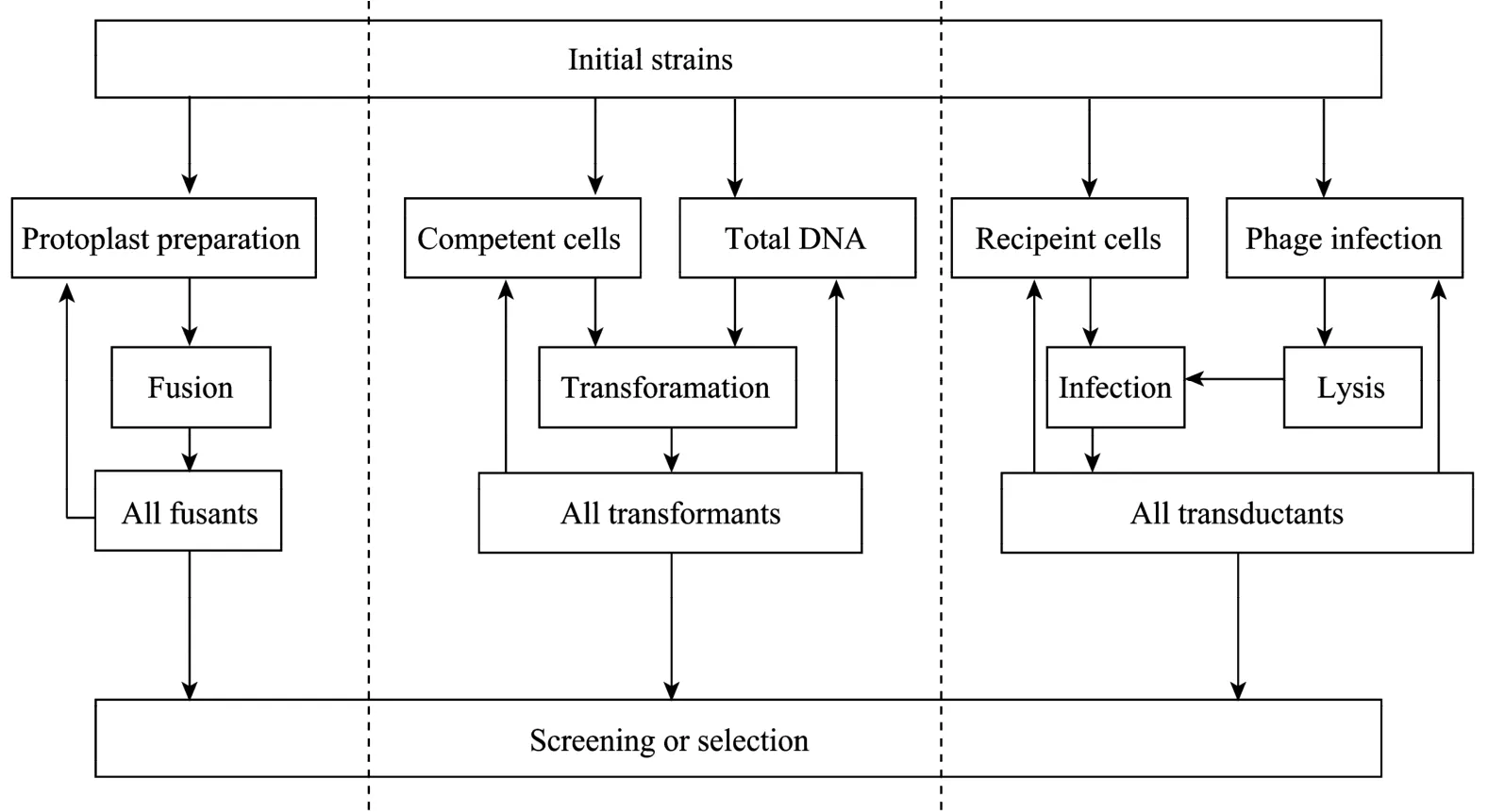
图2 3种技术手段进行基因组混组示意图Fig.2 Perform genome shuffling by recycling protoplast fusion, transformation or transduction.
1.4 循环转化
和 1.3节一样,取MMF5、MMF6、MMF7、MMF8的过夜培养物,等比例混合作为初始群体,计作为F0。抽提F0群体的基因组,并且转化F0群体的感受态细胞,获得的下一代群体定义为F1。同样,基因组转化F1,获得的下一代群体定义为F2。以后各代的依次为 F3、F4、F5。具体过程如图 2所示。
取各代细胞梯度稀释后涂布于添加不同氨基酸或碱基的基本培养基,计数各代中各种基因型细胞的数目并计算在群体中所占的比例。
为了确定每一批的感受态转化效率与基因组制备质量,引入了辅助菌株 FW7E。每一代进行过程中,FW7E基因组转化Fn (Fn代表F0、F1……),用于检验感受态效率;同时,使用 Fn 基因组转化FW7E,用于检验基因组抽提质量。
抽提基因组与制备感受态的具体实验方法参照文献[13]报道。
1.5 循环转导
和1.3节一样,取 MMF5、MMF6、MMF7、MMF8的过夜培养物,等比例混合作为初始群体,计作为F0。
将F0群体的一部分用噬菌体PBS1感染,制备裂解液。裂解液感染F0群体,获得的下一代群体定义为 F1。同样地,以 F1群体制备噬菌体裂解液感染 F1群体,获得的下一代群体定义为 F2。以后各代的依次为F3、F4、F5。具体过程如图2所示。
取各代梯度稀释后涂布于添加不同氨基酸或碱基的基本培养基,计数各代中各种基因型细胞的数目并计算它们在整个群体中所占的比例。
转导的具体实验方法参照文献[13]报道。
1.6 循环融合过程的电子模拟
编制程序,模拟含有 108个细胞的群体,以不同的重组频率,经过 5轮融合后,后代中各种基因型的所占比例的变化。
模拟过程使用穷举算法。以重组频率10%为例对算法进行具体说明:首先取出一个细胞,根据初始比例得到其基因型。有10%可能性这个细胞参加了融合和重组,则根据亲本基因型确定融合子基因型,计算产生下一代的细胞。剩余90%可能性,这个细胞在本轮融合过程中没有参加重组,即可能没有参加融合,保持亲本类型;也可能细胞暂时融合后,形成异核体,而后基因组没有进行重组而迅速分离成亲本类型,那么又按照其基因型不变,产生下一代的细胞。每一轮融合模拟计算 108个细胞,产生108个后代细胞。共模拟计算5轮。
FORTRAN95程序源代码如附录。使用ThinkPad T400 硬件系统,UBUNTU8.10、GFORTRAN 7.4软件系统,不同的重组频率参数下,分别重复运行 4次,取平均值。记录模拟的结果,并与实验结果以及文献报道进行对比分析。
2 结果
2.1 进行基因组混组的初始菌株
本实验首先获得的4株带有3缺陷的菌株,即MMF5、MMF6、MMF7、MMF8。对于purA16、thr5、metC3、trpC2 这4个位点,上述每一菌株分别带有1个原养型标记、3个缺陷型标记:MMF5 (purA16,thr5,trpC2,MetC3+)、MMF6 (purA16,metC3,trpC2,Thr5+)、MMF7 (thr5,metC3,trpC2,PurA16+)、MMF8 (purA16,thr5,metC3,TrpC2+),其中粗体且首字母大写的为原养型正标记。
这些标记在基因组上的位置如图 3所示;从上面的正标记的位置图可以看出来,4个标记在基因组上分布均匀。通过鉴定这4个菌株的后代群体中,带有2个或2个以上原养型正标记的个体的数量和所占的比例,可以比较基因组混组的效率。
2.2 原生质体的制备、再生与融合
经过实验条件摸索,确定了原生质体的制备、再生与融合的具体方案和参数。维持原生质体稳定的等渗溶液使用 SMMP (+BSA)。主要稳定剂是蔗糖,缓冲体系是磷酸盐和顺丁烯二酸盐,BSA用于提高再生率。溶菌酶浓度为2 mg/mL,37℃~39℃处理1~2 h,镜检观察至没有杆菌残留时停止溶菌酶处理。再生培养基选用DM3培养基,主要稳定剂和缓冲体系是丁二酸盐。融合方法使用PEG法,处理条件为40% PEG6000 冰浴1~2 min。
经多次重复,原生质体形成率,即原生质体数/(原生质体数+剩余杆菌数),一般大于99.9%;原生质体再生率,即 (再生菌数−剩余杆菌数)/(细胞总数−剩余杆菌数),为10%左右;单次融合过程中重组频率,即后代群体中重组体总数/后代群体总数,在10−5左右。这些都符合文献[14]报道。
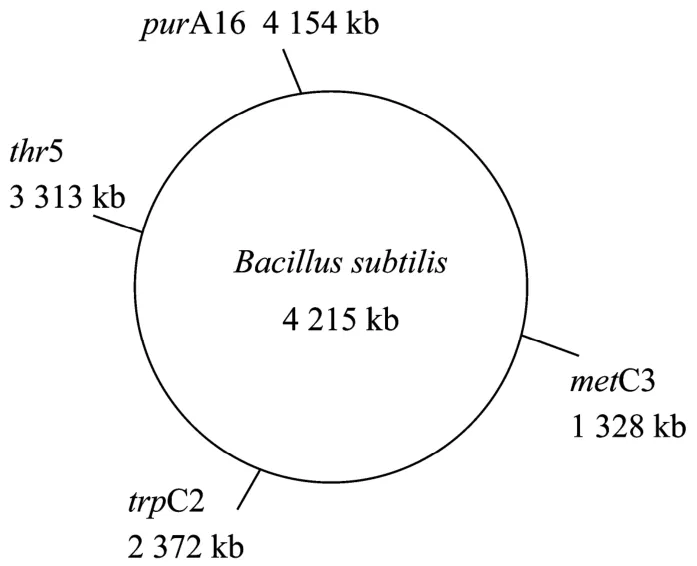
图3purA16、thr5、metC3、trpC2 4个基因枯草芽胞杆菌在染色体上的位置Fig.3 Positions ofpurA16,thr5,metC3 andtrpC2onBacillus subtilischromosome.
2.3 循环融合实验的结果
循环融合实验的结果见表 3。从中可以看出,经过5轮转化,其中同时带有4个标记的融合子没有稳定出现。带有3个和2个标记的后代,总体上有增加的趋势。
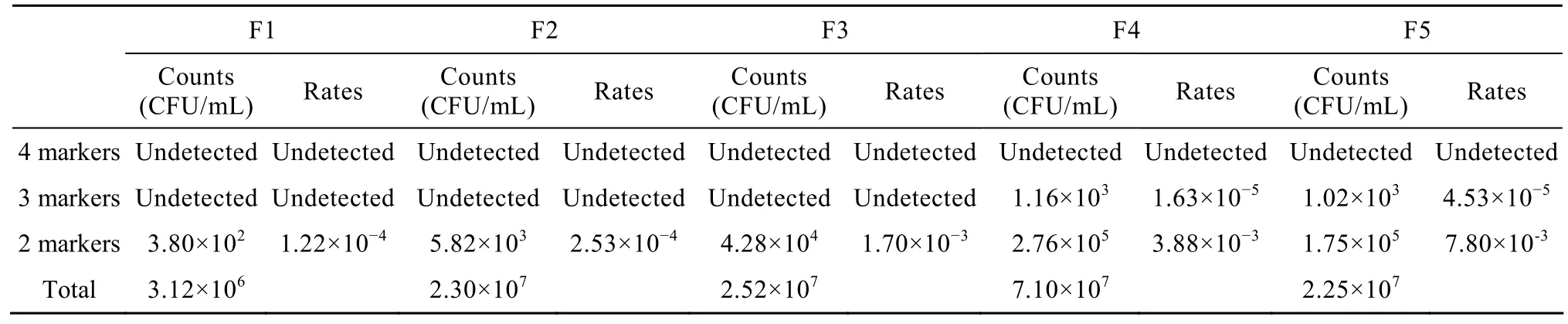
表3 五轮循环融合过程中各类型枯草芽胞杆菌后代的数量以及在群体中所占的比例Table 3 Counts and rates of descendant with 2, 3 and 4 markers in five rounds protoplast fusion
2.4 电子模拟循环融合过程的结果
分别把重组频率设定为 10−5、10−4、10−3、10−2、10−1作为参数,运行模拟程序。模拟 5轮融合后,含有多个标记的细胞在后代群体中所占的比例如表4所示。
对比实验结果和模拟结果。天蓝色链霉菌的实验结果和重组频率 10−1的模拟结果相互对应,带 4个标记的后代4到五轮融合后达到10−2数量级。枯草芽胞杆菌的实验结果和重组频率10−4左右的模拟结果相互对应。带4个标记的后代5轮融合后没有出现,带有 3个标记出现但比例不高。考虑到生长过程中带有更多个原养型标记基因的后代会略有优势。电子模拟过程基本上符合了实际的情况。

表 4 模拟不同重组频率下五轮融合后各类型枯草芽胞杆菌后代在群体中所占的比例Table 4 Rates of descendant with 2,3 and 4 markers after five rounds simulation protoplast fusion

表 5 天蓝色链霉菌和枯草芽胞杆菌循环融合实验结果比较Table 5 Comparison of the result of cycling protoplast fusion ofB. subitilisandS. colicolor
2.5 转化效率与转化频率
经过多次重复实验中,单次转化过程中重组频率基本可以保证在10−3数量级,也符合文献[15]报道。
2.6 循环转化实验的结果
循环转化实验的结果见表 6。从中可以看出,经过5轮转化,其中同时带有4个标记的转化子没有稳定出现。带有3个和2个标记的后代,总体上处于增加的趋势。
2.7 噬菌体效价和转导频率
裂解液效价在108PFU/mL以上。单次转导过程中重组频率 7.0×10−7~2.0×10−6之间,符合文献[15]报道。
2.8 循环转导实验的结果
循环转化实验的结果见表 7。从中可以看出,经过五轮转导,其中同时带有4个标记的转导子没有稳定出现。带有3个和2个标记的后代,总体上处于增加的趋势。
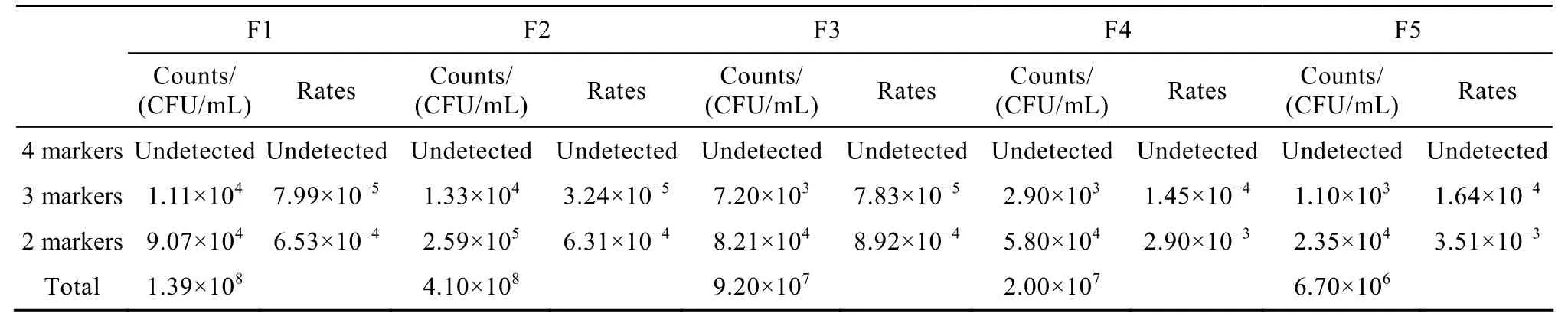
表6 五轮循环转化过程中各类型枯草芽胞杆菌后代的数量以及在群体中所占的比例Table 6 Counts and Rates of descendant with 2, 3 and 4 markers in five founds transformation

表7 五轮循环转导过程中各类型枯草芽胞杆菌后代在群体中所占的比例Table 7 Counts and rates of descendant with 2, 3 and 4 markers in five rounds transduction
3 讨论
据报道,使用4株带有4个不同标记基因的天蓝色链霉菌S. coelicolor为实验材料进行基因组混组实验,经过4轮原生质体融合,带有4个标记基因的后代在群体中的比例达到了 2.5%,带有 3个和2个遗传标记的后代所占的比例分别到达17%和60%[4]。根据本研究的结果,使用枯草芽胞杆菌做相同的实验,经过5轮原生质体融合,带有4个标记的后代没有出现。在3轮融合后带有3个遗传标记的后代开始出现。5轮后带有 3个遗传标记的后代最终比例是 10−5,带有 2个遗传标记的后代所占的比例也在10−3数量级。
究其原因,在于各种微生物细胞重组频率差别很大。天蓝色链霉菌融合过程中重组频率在10−1数量级,最高达到17%。链霉菌循环融合实验中,第1轮融合后,重组子比率达到8.4%[4]。这样,前一轮融合获得的重组子,有很大机会参加第 2轮融合而产生新的重组子。因此,4轮融合后的群体中,高达80% 的成员是经过基因重组的。其中2.5%是充分重组的后代,即带有来自4个不同亲本的4个不同的遗传标记。另外的报道中,使用通过基因组混组的方法提高了乳杆菌的耐酸性能,其每轮融合的重组频率为0.1%~1%[5]。而枯草芽胞杆菌原生质体融合过程中重组频率在 10−5的数量级,明显低于上述二者。常规单轮融合后,重组的后代在后代群体中所占的比例很少,群体中的绝大多数成员仍然是亲本类型。多轮融合过程中,重组的后代参加后一次融合获得再次重组的机会很低。
循环融合的电子模拟结果同样印证了这一点。单轮的重组频率差一个数量级,带有3个或者4个标记的后代在多轮融合后所占的比例,就会差 2~3个数量级。因此,要达到充分的基因组混组,需要一种有能够实现微生物细胞间高频重组的操作技术作为基础:循环融合中,不低于10−3~10−2的重组频率应该是进行有效混组的技术基础。否则重组频率较高的后代在群体中所占的频率将会很低,从中筛到具有优良性状的后代会较为困难。
同时,本研究中引入了循环融合和循环转导这两种技术手段进行基因组混组。从循环转化的结果来看,单轮转化后除了产生带有 2个遗传标记的后代外,同时也产生了带有 3个遗传标记的后代。解释为只要发生共转化事件,即可实现 3个遗传标记集中于一个后代细胞。经过多轮转化后,重组细胞有所增加,3个遗传标记的后代在10−4数量级。循环转导的结果和循环转化的结果类似,单轮转导即可产生了带有3个遗传标记的后代。多轮转导后,3个遗传标记的后代也在 10−3数量级。其中,转导结果后代所占比例总体上显示增加的趋势,但有一定的波动,分析可能的原因是部分细胞被噬菌体裂解影响到了总菌落计数。总结起来,循环融合和循环转导这两种技术手段同样无法实现高效的基因组混组。根据目前获得的结果,尚无法对枯草芽胞杆菌 3种技术手段进行基因组混组的优劣作出明确的结论。
如果使用转化或者转导的技术手段进行有效基因组混组,考虑其单轮引入的片段更短,所以对于转化频率或转导频率要求会比循环融合更高一些,也不应低于 10−3~10−2数量级。在关于多元自动化基因组工程 (Mupltiplexautomated genome engineering,MAGE) 技术的报道中,每一轮操作中可以使群体中30%以上的细胞获得了新的遗传修饰。由此推而广之,包括多元自动化基因组工程、基因组混组在内的多种基因组水平的菌种改造手段,可能都需要有一种技术手段,保证每一次操作后,群体中获得修饰的成员在整体中所占的比例达到一定的水平,最终经过多轮操作后才能得到多样性较为丰富的突变库,最终更容易从中筛选到性状优良的成员。
退一步讲,根据本研究的结果,多轮实验操作后,虽然重组子比例较低、基因组混组不充分,但也有一定程度的混组,已经出现了带有 3个筛选标记的后代。如果扩大混组群体的规模,或者增加循环数,无筛选的条件下仍然有可能获得充分混组、带有 4个标记的后代。如果后续有高效筛选方法,可以将群体中较低频率出现的一些重组子有效地筛选出来。应用于育种的过程,仍然有可能获得性能提高的菌株。
如果在枯草杆菌的实验技术上有突破,比如重组频率可以大幅提高,那么多轮操作后基因组混组的效果同样可以得到提高。原生质体灭活法可以有效地提高筛选到优良融合子的机率,并且用于紫杉醇生产菌树状多节孢菌Nodulisporium sylviform的基因组混组[16]。原生质体融合前,首先以不同的方式灭活。灭活的过程中对原生质体的某些方面造成了一定的损害,使其无法再生。不同的方法造成的损害是不同的,比如热灭活主要是蛋白质变性,紫外灭活主要是DNA损伤。因此,不同方法灭活后的原生质体融合后可以相互互补损害,理论上只有融合子才可以再生。这样减少了一些筛选工作,提高了筛选到优良融合子的机率。新的融合技术的引入也可能解决重组频率低的问题。李寰宇[17]和 Gong等[18]使用飞秒激光诱导红法夫酵母Phaf fi a rhodozyma细胞融合,重组频率达到80%以上。另外,一种新报道的融合方法是使用微观流体设备直接捕获原生质体用于融合[19]。此种方法中用于融合的细胞是有导向的选择,因此比其他方法有更高的融合效率。
REFERENCES
[1] Patnaik R. Engineering complex phenotypes in industrial strains.BiotechnolProg, 2008, 24(1): 38−47.
[2] Gong JX, Zheng HJ, Wu ZJ,et al. Genome shuf fl ing:progress and applications for phenotype improvement.Biotechnol Adv, 2009, 27(6): 996−1005.
[3] Chen T, Wang JY, Zhou SQ,et al. Trait improvement of riboflavin producingBacillus subtilisby genome shuffling and metabolic flux analysis.J Indust Engin Chem(China),2004, 55(11): 1842−1848.
陈涛, 王靖宇, 周世奇, 等. 基因组改组及代谢通量分析在产核黄素Bacillus subtilis性能改进中的应用. 化工学报, 2004, 55(11): 1842−1848.
[4] Zhang YX, Perry K, Vinci VA,et al. Genome shuf fl ing leads to rapid phenotypic improvement in bacteria.Nature, 2002, 415(6872): 644−646.
[5] Patnaik R, Louie S, Gavrilovic V,et al. Genome shuf fl ing ofLactobacillusfor improved acid tolerance.Nat Biotechnol, 2002,20(7): 707−712.
[6] Liang HY, Guo Y. Whole Genome shuffling to enhance activity of fibrinolytic enzyme-producing strains.China Biotechnol, 2007, 27(10): 43.
梁惠仪, 郭勇. 全基因组重排育种技术提高豆豉纤溶酶菌产酶量. 中国生物工程杂志, 2007, 27(10): 43.
[7] Chen L, Chen WL. Genome shuf fl ing enhanced antagonistic activityagainstFusarium oxysporumf. sp.melonisand tolerance to chemical fungicidesinBacillus subtilisbs14.J Food Agri Environ, 2009, 7(2): 856−860.
[8] Hartl H, Wehrl W, Wiegert T,et al. Development of a new integration site within theBacillus subtilischromosome and constructionof compatible expression cassettes.J Bacteriol, 2001, 183(8): 2696−2699.
[9] Feucht A, Lewis PJ. Improved plasmid vectors for the production of multiple fl uorescent protein fusions inBacillus subtilis. Gene, 2001, 264(2): 289−297.
[10] Ziegler DR.BacillusGenetic Stock Center Catalog of Strains.7th ed. Columbus: Ohio State University Press, 2002.
[11] Zeigler DR, Prágai Z, Rodriguez S,et al. The origins of 168, w23, and otherBacillus subtilislegacy strains.J Bacteriol, 2008, 190(21): 6983−6995.
[12] Dedonder RA, Lepesant JA, Lepesant-Kejzlarová J,et al.Construction of a kit of reference strains for rapid genetic mapping inBacillus subtilis168.Appl Environ Microbiol,1977, 33(4): 989−993.
[13] Cutting SM, Vander Horn PB. Molecular biological methods forBacillus//Genetic Analysis. West Sussex:John Wiley, 1990: 27−74.
[14] Shi QQ, Wu SG. Industrial Microbial Breeding Science.3rd ed. Beijing: Science Press, 2009.
施巧琴, 吴松刚. 工业微生物育种学. 3版. 北京: 科学出版社, 2009.
[15] Carlton BC. Transformation mapping of the genes controlling tryptophan biosynthesis inBacillus subtilis.J Bacteriol, 1967, 94(3): 660−665.
[16] Zhao K, Ping WX, Zhang LN,et al.Screening and breeding of high taxol producing fungi by genome shuffling.Sci China C: Life Sci, 2008, 51(3): 222−231.
[17] Li HY, Gong JX, Xing QR,et al.Studies on femtosecond laser induced cell fusion.Chin J Lasers,2006, 33(12):1642.
李寰宇, 巩继贤, 邢岐荣, 等. 飞秒激光诱导细胞融合技术的实验研究. 中国激光, 2006, 33(12): 1642.
[18] Gong JX, Zhao XM, Xing QR,et al. Femtosecond laser-induced cell fusion.Applied Physics Lett, 2008,92(9): 093901-093901-3.
[19] Skelley AM, Kirak O, Suh HK. Micro fl uidic control of cell pairing and fusion.Nat Methods, 2009, 6(2): 147−152.
Genome shuffling method ofBacillus subtilis
Junjie Yang1,2, Wenchao Fan3, Han Xiao1, Chunhong Guan1, Chuanzeng Cao1,3, Haifeng Shao4,Weihong Jiang1, and Sheng Yang1,2,3
1Key Laboratory of Synthetic Biology,Institute of Plant Physiology and Ecology,Shanghai Institutes for Biological Sciences,Chinese Academy of Sciences,Shanghai200032,China
2Shanghai Research and Development Center of Industrial Biotechnology,Shanghai201201,China
3Huzhou Research Center of Industrial Biotechnology,Shanghai Institutes for Biological Sciences,Chinese Academy of Sciences,Huzhou
313000,China
4Zhejiang Shunfeng Haider Co. Ltd.,Dongyang322100,China
Received:March 20, 2010;Accepted:June 7, 2010
Supported by:National Natural Science Foundation of China (Nos. 30370022, 30570028), National Basic Research Program of China (973 Program)(No. 2007CB707803).
Corresponding author:Sheng Yang. +86-21-54924173; Fax: +86-21-54924015; E-mail: syang@sibs.ac.cn
国家自然科学基金 (Nos. 30370022, 30570028),国家重点基础研究发展计划 (973计划) (No. 2007CB707803) 资助。
