山羊群体遗传多样性的微卫星分析
2010-10-11许丽梅刘丑生张利平王志刚韩旭李晓霞常爽
许丽梅,刘丑生,张利平,王志刚,韩旭,李晓霞,常爽
1 甘肃农业大学,兰州 730070
2 全国畜牧总站 畜禽牧草种质资源保存利用中心,北京 100193
3 河南农业大学,郑州 450002
动物及兽医生物技术
山羊群体遗传多样性的微卫星分析
许丽梅1,刘丑生2,张利平1,王志刚2,韩旭2,李晓霞1,常爽3
1 甘肃农业大学,兰州 730070
2 全国畜牧总站 畜禽牧草种质资源保存利用中心,北京 100193
3 河南农业大学,郑州 450002
为了保护和合理利用我国地方山羊品种遗传资源提供理论基础,本研究利用国际农粮组织和国际家畜研究所推荐的10对微卫星引物,结合荧光标记PCR,检测了中国9个地方山羊品种和1个引进山羊品种的遗传多样性。所研究的10个品种中7个呈现出高度多态,3个呈现出中度多态。并共检测到119个等位基因,有效等位基因数在1.4641~9.2911之间,座位平均杂合度在0.2618~0.7672之间,品种平均杂合度在0.5196~0.7024之间,其中SRCRSP23位点和河西绒山羊 (HXR) 平均杂合度最高。聚类关系 (NJ和UPGMA) 和主成分分析结果与其起源、育成历史及地理分布基本一致。
山羊,微卫星,遗传多样性
Abstract:Fluorescence PCR was applied to investigate the genetic diversities of 9 indigenous Chinese goat breeds and 1 exotic breed with 10 microsatellite DNA markers recommended by the Food and Agriculture Organization of the United Nations and the International Livestock Research Institute of Animal Genetics, which provide data for the preservation and utilization of indigenous goat breeds genetic resource. We found that the 7 breeds were high polymorphic while 3 breeds were moderate polymorphic. We also detected 119 alleles, and the effective allele number ranged from 1.4641 to 9.2911. The average heterozygosity of loci and breeds respectively varied from 0.2618 to 0.7672 and from 0.5196 to 0.7024. As well as SRCRSP23 site and Hexi cashmere goat had the highest average heterozygosity. Then we analyzed the phylogenetic trees (NJ and UPGMA), and found both of them were generally in accordance with their original breeding history and localities.
Keywords:goat, microsatellite, genetic diversity
山羊为分布范围广、产品类型多、适应性较强的物种,因此其表现出了丰富的遗传多样性。我国地方山羊品种资源十分丰富,根据2004年《中国畜禽遗传资源状况》,我国山羊品种为 50个,地方品种有43个[1]。但近年来随着国外优良品种的大量引入、生态环境的逐渐恶化,我国许多地方固有品种处于濒危或濒临灭绝状态。对于山羊这种几千年来持续驯养的家畜物种,其所携带的优良基因一旦消失,无论是从种质资源保护的深远意义,还是从社会生产的经济效益考虑,都将是无法挽回的损失[2-4]。本研究利用10个微卫星位点,结合荧光引物PCR技术,利用ABI3130XL自动测序仪进行基因型判定,对10个山羊品种进行遗传学检测,探讨品种内和品种间的遗传多样性和遗传分化关系,旨在为我国地方山羊种质资源的保护和合理利用提供理论依据。
1 材料与方法
1.1 试验样本
10个山羊品种共计447个个体,样品具体信息见表 1。本实验试验样本均由农业部全国畜牧兽医总站畜禽牧草种质资源保存利用中心提供。
1.2 试剂及器材
pBR322/MspⅠ购自北京华美生物工程公司;普通下游引物、rTaq酶购自大连宝生物技术有限公司;全血基因组提取试剂盒购自 TIANGEN公司;ABI3130XL测序仪、荧光引物购自美国应用生物系统公司 (ABI),引物具体信息见表2。
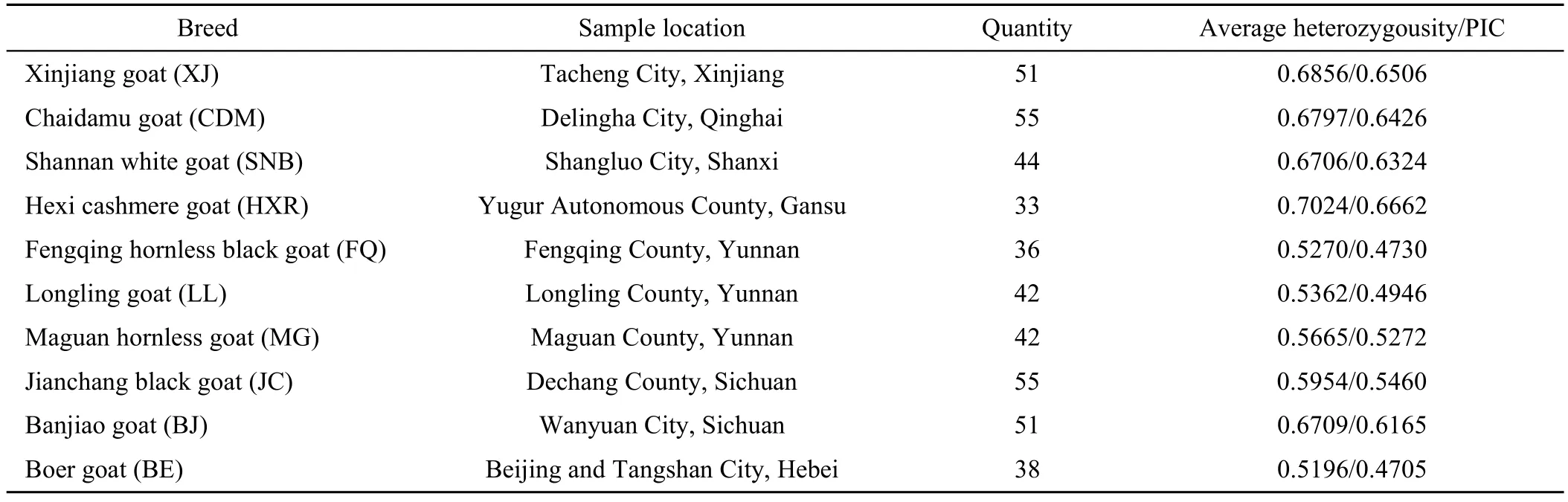
表1 样品采集信息Table 1 Sample information

表2 10个微卫星座位信息Table 2 Information of 10 microsatellite loci
1.3 血样处理及基因组DNA提取
将冷冻的血样在20℃水浴中解冻,用全血基因组提取试剂盒提取 DNA,具体步骤参照试剂盒说明书。
1.4 PCR体系及条件
PCR 反应体系为:10×buffer 1.5 μL,Mg2+1.2~1.5 μL,引物 0.2~0.5 μL,dNTPs 1.2~1.5 μL,rTaq酶0.3 μL,ddH2O补足至15 μL。反应条件:95℃预变性5 min;94℃变性1 min,50℃~61℃退火1 min,72℃延伸1 min,30~36个循环;72℃延伸60 min;4℃保存。
1.5 PCR产物的检测
PCR 扩增后,先取7 μL产物进行8%非变性聚丙烯酰胺凝胶电泳,银染法检测。然后再用ABI3130XL 测序仪进行分析。
1.6 统计分析
利用Microsatellite Toolkitve version 3.1进行数据格式的转化;利用 GENEPOP软件进行 Hardy-Weinberg平衡检验;利用POPGENE软件计算等位基因数、有效等位基因数等;利用 DISPAN软件构建NJ和UPGMA系统发生树[5-6];利用MVSP软件进行群体分化成分分析。
2 结果
2.1 Hardy-Weinberg平衡检验
微卫星DNA本身是选择中性的,不受选择的压力,因此在一个理想群体中各等位基因在群体中的分布频率应该是稳定的[7]。利用 Hardy-Weinberg平衡检验结果见表3、4。

表3 Hardy-Weinberg平衡检验各座位P值Table 3 ThePvalue of every locus in Hardy-Weinberg
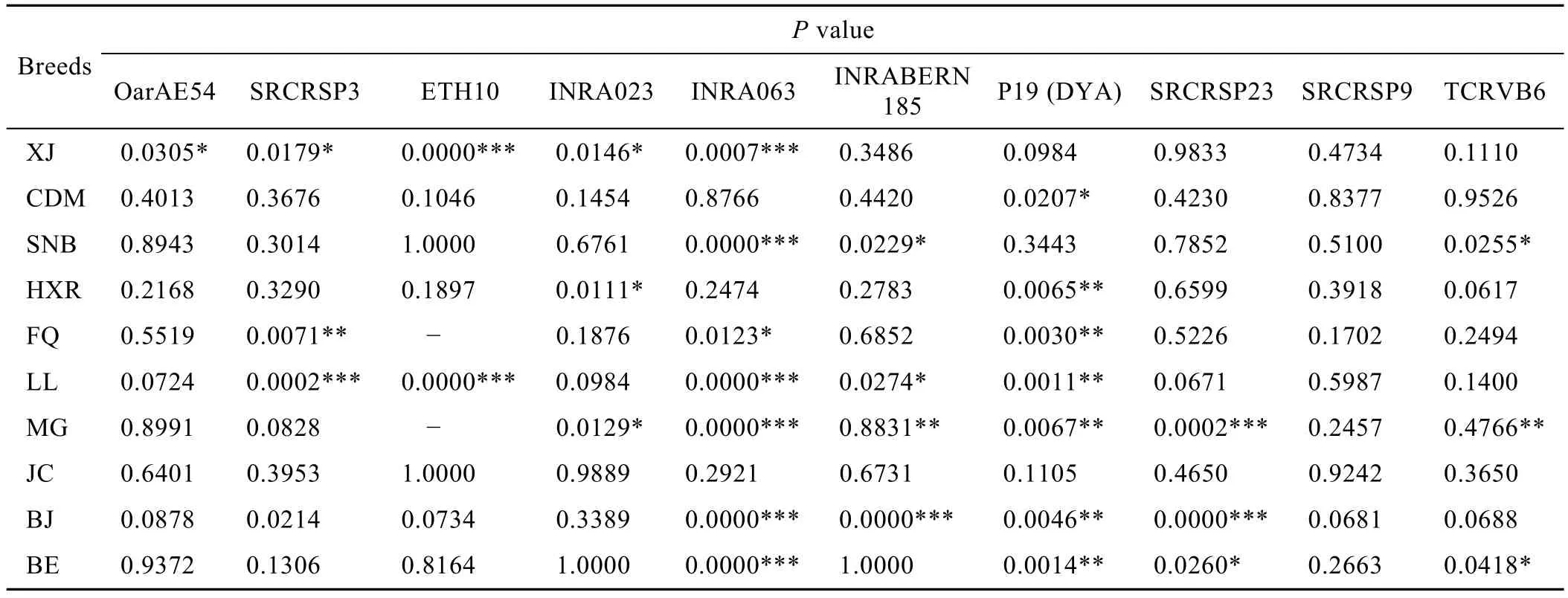
表4 Hardy-Weinberg平衡检验各群体P值Table 4 ThePvalue of every population in Hardy-Weinberg
2.2 等位基因的检测及其平均杂合度和平均多态信息含量
本实验研究10个山羊群体的10个微卫星等位基因数据都是通过 ABI3130XL测序仪分析获得(具体座位信息见表2),数据用GeneMapper软件分析。ABI3130XL检测的部分微卫星座位结果见图1(A、B)。
利用 Nei[8-9]公式,根据各微卫星座位等位基因频率,计算各位点的等位基因数、有效等位基因数、期望杂合度和平均杂合度 (表5)。
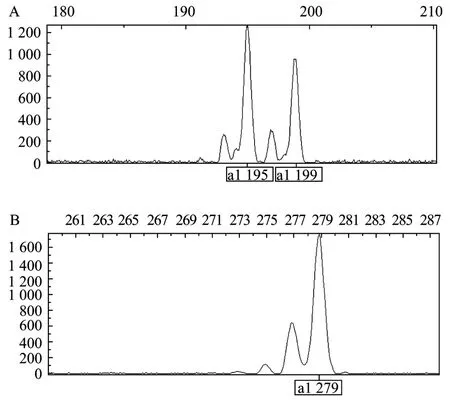
图1 座位INRA023杂合子 (A) 和座位INRABERN 185纯合子 (B) 的ABI3130XL检测结果Fig.1 Detection of loci INRA023 He (A) and loci INRABERN185 Ho (B) on ABI313OXL.

表5 各位点的等位基因数、有效等位基因数、期望杂合度、平均杂合度Table 5 Na/Ne/Exp-He/Average He of all loci
2.3 群体间的遗传距离
用Dispan软件计算出各品种间的遗传距离,见表6。
2.4 10个山羊品种的聚类分析
利用Dispan软件构建Nj和UPGMA系统发生树,结果如图2A、2B所示。依图2A所示,XJ和HXR聚到一起,再和CDM聚到一起;SNB和FQ先聚到一起,然后再和CDM、HXR、XJ聚为一类;LL和JC先聚到一起,然后和MG聚到一起,再和BJ聚为一类;最后是对照品种BE。依图2B所示,XJ和HXR聚到一起,再和CDM聚到一起,随后依次和 SNB、BJ、FQ先聚为一类;LL和 MG先聚到一起,然后再和 JC聚到一起;最后是对照品种 BE。两种聚类分析方法得到的聚类结果基本一致。

表6 10个山羊品种间的遗传距离Table 6 Genetic distance between ten goat breeds
2.5 群体间遗传分化的主成分分析
群体间遗传分化是研究多个群体间在多个标记座位上的差异性,具有多变量的特征,因而通过主成分分析可以揭示群体间遗传分化程度并反映群体间的遗传关系[10-12]。利用MVSP软件对10个山羊群体进行主成分分析,结果如图3所示,其中第1主成分、第2主成分、第3主成分分别占总变异的31.42%、25.52%和11.77%。

图2 10个山羊品种的DA/NJ聚类图 (A) 和DA/UPGMA聚类图 (B)Fig.2 DA/NJ tree (A) and DA/UPGMA tree (B) among 10 goat breeds.

图3 10个山羊品种群体间遗传分化的第1主成分、第2主成分、第3主成分散点图Fig.3 Scatter plot of showing the first, the second and the third principal components of genetic differentiation among 10 goat breeds.
3 结论
3.1 Hardy-Weinberg平衡
根据Hardy-Weinberg定律,在一个没有选择、突变和迁移的随机大群体中,基因频率和基因型频率在世代之间保持不变[1]。群体遗传不平衡是生物体在长期自然进化过程中形成的群体特征,是遗传资源评价、经济性状标记等研究中的重要内容。在生态系统中,基因的流动遵循Hardy-Weinberg定律,但由于基因突变、选择和遗传漂变等多种因素的作用,Hardy-Weinberg平衡往往被打破,造成基因频率和基因型频率的非随机分布 (即遗传不平衡),其中选择在这一过程中发挥主要作用[13]。选择包括人工选择和自然选择,同时由于动物个体的体质差异,自由交配不等于随机交配。本研究中的大部分群体处于不平衡状态,利用P值的无偏估测对各群体和各座进行检测,发现10个群体只在座位OARAE54(P=0.2161) 和 SRCRSP9 (P=0.4196) 上处于平衡状态,而在其他座位都不同程度地偏离平衡;除CDM(P=0.2415)、JC (P=0.8236) 处于平衡状态,其他群体均处于哈温不平衡。出现这种情况的原因首先可能与样品的采集有关,主要是由于人工不能作到随机选择。其次可能与样本含量有关,样本含量是误差的决定因素。要把取样误差降低到统计学所允许的范围之内,必须要有一个足够大的样本。Barker等[14]提出每个品种应分析25个个体样品,如果样本含量达到50个,则能够弥补可能的失误。在本实验中平均每个群体有40个左右,基本满足以上条件,研究结果可以代表整个群体的全部遗传信息,故可以排除这点影响。最后可能与群体的繁育方式有关,由于地方品种公畜数量有限和地域的限制,长时间的自由交配导致群体遗传不平衡。
3.2 群体遗传多样性
多态信息含量和平均杂合度是群体内遗传变异的量度,可以用来描述微卫星座位的变异程度[15-16]。本实验的10个山羊群体均表现出了多态性,平均多态信息含量大于0.5,其中HXR多态信息含量高达0.666,然后依次是XJ、CDM、BJ、SNB、JC、BJ、LL、FQ,最低的为 BE多态信息含量为 0.471,平均杂合度的结果与多态信息含量一致。10个位点中TCRVB6等位基因数最多为19个,等位基因数目最少的座位是ETH10,检测到6个。10个群体平均有效等位基因数在 4.3~2.4之间,HXR平均有效等位基因数最多为4.3个。
本实验的群体遗传多样性量度的范围与国内其他研究结果稍有差异,狄冉[3]报道新疆山羊平均杂合度为 0.7049,多态信息含量为0.6584,HXR平均杂合度为 0.6052,多态信息含量为 0.5587;王杰等[17]用10个微卫星检测JC平均杂合度为0.7739,多态信息含量为 0.7352。这是由于本实验所采用的微卫星位点与其他研究不同造成的。
在本实验中新疆山羊和河西绒山羊都表现了丰富的遗传多样性,这可能是由于其产区的地理环境复杂,长期不定向培育和不断引进其他品种杂交造成的结果[18]。新疆山羊在新疆各地均有分布,是肉、毛、绒、乳兼用的地方品种,也有可能成就其丰富的遗传多样性。而且新疆和河西走廊这些地方皆位于古丝绸之路,中亚地区的山羊品种被引入是可能的。波尔山羊的遗传多样性较低,主要是由于采集数量较少;另外波尔山羊繁育历史悠久,是世界上最优秀的肉用山羊品种,是优良公羊的重要品种来源,作为终端父本能显著提高杂交后代的生长速度和产肉性能,国内繁育的波尔山羊数量有限,近交现象比较严重。
3.3 遗传距离和聚类分析
遗传距离的度量对品种间遗传变异提供了一个最佳的有效而客观的描述[19]。本研究采用 DA、DS分别构建NJ树、UPGMA树和计算群体间遗传距离,并采用主成分分析进行聚类分析。Takezaki等[20]认为在物种内群体间的遗传变异 DA是获得准确系统发生树的最有效方法;而 DS用于估计群体分化时间上为最优。狄冉[3]也证明了 DA用来构建进化树的准确性。另外,本研究中NJ聚类图自检值相对较低,与 UPGMA聚类图略有差异。由 DA构建的UPGMA聚类图较符合样本的地理分布与育种历史。从表 6中可以看出:XJ和 HXR的遗传距离最近(DA=0.0297),XJ和 CDM 距离次之 (DA=0.0457),LL和BE之间的遗传距离最远 (DA=1.1549)。品种间的亲缘关系不仅要从地理位置和起源上来确定,还应考虑到迁移、突变、环境效应以及微卫星座位数目等其他因素的影响[21]。
根据聚类分析可以看出XJ和HXR聚为一类,再和 CDM 聚到一起,结果与山羊群体的地理分布一致[22-23]。FQ明显有别于其他云南地方品种,这与地理位置不一致。云南省地形复杂多样,自然气候和生态环境类型甚多,确有研究证实凤庆无角黑山羊有别于云南其他山羊品种。凤庆无角黑山羊是云岭山羊和临沧长毛山羊经多年繁育杂交和后代选育形成的品种[24],极可能受近交的影响。李彦屏等[25]认为凤庆无角黑山羊从体尺、体重、生长速度、产肉性能等均有别于马关无角山羊。此外因云南省与四川省接壤,部分地区的自然条件极为相似,也可能是造成建昌山羊、龙陵山羊和马关无角山羊 3个遗传距离相近的原因;也有可能是位点数和取样数不足的原因,因为当遗传距离期望值较低时,相对测定误差较大,因此要准确确立较相似品种间的遗传关系,还应在位点数和取样个体数上多加考虑,使变异系数控制在较低水平上,进而准确区分其品种。
3.4 主成分分析
从散点图可以看出,XJ、HXR、CDM、SNB聚为一类;JC、LL、MG聚为一类;BJ和FQ位于两类之间;BE与其它山羊群体明显分开。这与聚类分析结果一致,足以证明结果的可靠性。
REFERENCES
[1] Qi JF, Jia YL, He XT,et al. Report on Domestic Animal Genetic Resources in China. Beijing: China Agriculture Press, 2004: 12−13.
齐景发, 贾幼陵, 何新天, 等. 中国畜禽遗传资源状况.北京: 中国农业出版社, 2004: 12−13.
[2] Liu CS, Wang ZG, Li N. Current condition and countermeasure of genetic resources of endangered domestic animal and poultry in China.China Anim Husband Bull, 2004, 1: 28−31.
刘丑生, 王志刚, 李宁. 我国濒危畜禽遗传资源保护的现状与对策. 中国牧业通讯, 2004, 21: 28−31.
[3] Di R. The microsatellite and SNPs study of Chinese cashmere goats [D]. Beijing: Chinese Academy of Agricultural Science, 2008.
狄冉. 中国产绒山羊微卫星和单核苷酸多态性研究[D].北京: 中国农业科学院, 2008.
[4] Simianer H, Chairman of the group. Report of the ISAG/FAO advisory group on animal genetic diversity.FAO Report, 2007.
[5] Li MH, Tapio I, Vilkki J,et al. The genetic structure of cattle populations (Bos taurus) in northern Eurasia and the neighboring Near Eastern regions: implications for breeding strategies and conservation.Mol Ecol, 2007, 16:3839−3853.
[6] Kumar S, Dixit SP, Verma NK,et al. Genetic diversity analysis of the gohilwari breed of Indian goat (Capra hircus) using microsatellite markers.Am J Anim Vet Sci,2009, 4(3): 49−57.
[7] Sheng ZL, Wu CX. Quantitative Genetics. Beijing: China Agriculture Press, 1999: 7−71.
盛志廉, 吴常信. 数量遗传学. 北京: 中国农业出版社,1999: 7−71.
[8] Nei M. Genetic distance between populations.Am Nat,1972, 106: 283−292.
[9] Nei M, Tajima F, Tateno Y, Accuracy of estimated phylogenetic trees from molecular data II. gene frequency data.J Mol Evol, 1983, 19: 153−170.
[10] Li XL, Valentini A. Genetic diversity of Chinese indigenous goat breeds based on microsatellite markers.J Anim Breed Genet, 2004, 121: 350−355.
[11] Pritchard JK, Stephens M, Donnell YP. Inference of population structure using multilocus genotype data.Genetics, 2000, 155(2): 945−959.
[12] Saitbekova N, Gaillard C, Ruff GO,et al. Genetic diversity in Swiss goat breeds based on microsatellite analysis.Anim Genet,1999, 30: 36−41.
[13] Pan QJ, Li XL, Min LJ,et al. Genetic co-adaptability among structural genes under the condition of genetic disequilibrium.Hereditas, 2007, 29(5): 643−648.
潘庆杰, 李晓林, 闵令江, 等. 群体遗传不平衡条件下的遗传共适应现象及其遗传分析. 遗传, 2007, 29(5):643−648.
[14] Barker JS, Bradleyd G, Fries R,et al. An integrated global program to establish the genetic relationships among the breeds of each domestic animal species.FAO Report,1993.
[15] Wang J, Liu CS, Zhang LP,et al. Individual identification and paternity testing of bulls using microsatellite.Hereditas, 2009, 31(3): 285−289.
王静, 刘丑生, 张利平, 等. 微卫星在种公牛个体识别与亲缘鉴定方面的应用. 遗传, 2009, 31(3): 285−289.
[16] Zhong T, Ma YH, Guan WJ,et al. Genetic diversity of microsatellite DNA among ten sheep breeds.Chin J AnimVet Sci, 2008, 39(5): 555−561.
仲涛, 马月辉, 关伟军, 等. 10个绵羊品种的微卫星DNA多态性研究. 畜牧兽医学报, 2008, 39(5): 555−561.
[17] Wang J, Chen MH, Huatai CR,et al. Polymorphism research of microsatellite DNA in nine Sichuan black goat breeds (populations).Chin J Anim Vet Sci, 2006, 37(11):1124−1129.
王杰, 陈明华, 华太才让, 等. 四川 9 个黑山羊品种(群体) 微卫星DNA多态性研究. 畜牧兽医学报, 2006,37(11): 1124−1129.
[18] Li MH, Zhao SH, Bian C,et al. Genetic relationships among twelve Chinese indigenous goat populations based on microsatellite analysis. Genet Sel Evol, 2002, 34:729−744.
[19] Zhao YH, He XH, Guan WJ,et al. Analysis of genetic diversity of chinese six goat breeds by microsatellite markers.Chin J Anim Vet Sci, 2007, 38(1): 20−24.
赵艳红, 何晓红, 关伟军, 等. 中国 6个山羊群体微卫星标记的遗传多样性分析. 畜牧兽医学报, 2007, 38(1):20−24.
[20] Takezaki N, Nei M. Genetic distances and restruction or phylogenetic trees from microsatellite DNA.Genetic,1996, 144: 389−399.
[21] Zhang GX, Wang ZG, Chen WS,et al. Genetic diversity and population structure of indigenous yellow cattle breeds of China using 30 microsatellite markers.Anim Genet, 2007, 38: 550−559.
[22] Yang L, Zhao SH, Li K,et al. Determination of genetic relationships among five indigenous Chinese goat breeds with six microsatellite markers.Anim Genet, 1999, 30:452−455.
[23] Zhang XF, Li L, Shen W,et al. Genetic and phylogeny status of Chaidamu goat population.Hereditas, 2005,27(1): 75−79.
张西锋, 李兰, 沈伟, 等. 柴达木山羊群体遗传结构及系统地位的研究. 遗传, 2005, 27(1): 75−79.
[24] Zhang Y, Guo CY, Gao ZY. Discussion of Yunnan province local goat breeds on germplasm.J Anim Sci Vet Med, 2008, 35(11): 163−165.
张莹, 郭成裕, 高子尧. 云南山羊地方品种种质资源初探. 中国畜牧兽医, 2008, 35(11): 163−165.
[25] Li YP, Zhang CL, Li WZ,et al. Investigation report of Fengqing hornless black goat on germplasm//Proceedings of the national animal genetic resources conservation and utilization symposium, 2005: 502−505.
李彦屏, 张春利, 李文章, 等. 凤庆无角黑山羊种质资源调查报告//全国畜禽遗传资源保护与利用学术研讨会论文集, 2005: 502−505.
Genetic diversity in goat breeds based on microsatellite analysis
Limei Xu1, Chousheng Liu2, Liping Zhang1, Zhigang Wang2, Xu Han2, Xiaoxia Li1,and Shuang Chang3
1Gansu Agricultural University,Lanzhou730070,China
2National Animal Husbandry & Veterinary Service of the Ministry of Agriculture (MOA),Beijing100193,China
3Henan Agricultural University,Zhengzhou450002,China
Received:January 19, 2010;Accepted:March 17, 2010
Supported by:Ministry of Agriculture 2009 Protection Project of Animal Germplasm Resources (No. [2009]99).
Corresponding author:Chousheng Liu. Tel: +86-10-62814021; E-mail: liuchousheng@sina.com
农业部2009年畜禽种质资源保护项目 (农财发[2009]99号) 资助。
