黑足鲍COⅠ基因序列在遗传分析中的初步研究
2010-09-24高海霞侯旭光
高海霞, 侯旭光, 祝 茜,2
(1. 山东大学 威海分校 海洋学院, 山东 威海 264209; 2. 国家海洋局 第三海洋研究所, 福建 厦门 361005)
黑足鲍COⅠ基因序列在遗传分析中的初步研究
高海霞1, 侯旭光1, 祝 茜1,2
(1. 山东大学 威海分校 海洋学院, 山东 威海 264209; 2. 国家海洋局 第三海洋研究所, 福建 厦门 361005)
对引自新西兰的黑足鲍(Haliotis iris)的线粒体DNA 13个蛋白基因之一细胞色素C氧化酶亚基基因进行了特异性扩增和测序, 并分析了524 bp的碱基序列。结果显示, 在37个野生个体的基因序列中检测到3个变异位点, 定义了3个单倍型, 个体间序列差异很小。单倍型多样性(Hd)和核苷酸多样性(Pi)分别为0.249和0.000 79, 遗传多样性水平很低。在以贻贝(Mytilus edulis)为外群构建的系统发育树上, 黑足鲍处于相对适中的位置。本次研究以期为了解黑足鲍的遗传学背景提供科学依据,同时为鲍COⅠ基因的研究提供资料。
黑足鲍(Haliotis iris); COⅠ; 遗传多样性; 系统发育
黑足鲍(Haliotis iris)属软体动物门(Mollusca),腹足纲(Gastropoda), 前鳃亚纲(Prosobranchia), 原始腹足目(Archaeogastropoda), 鲍科(Haliotidae)。分布于太平洋西南部大洋洲沿岸, 主要分布于新西兰的部分岛礁周围海域。外壳常被作为漂亮的工艺品出售, 并在国际市场上很受欢迎[1], 是一种具有很高经济价值的鲍。继红鲍(H. rufescens)、绿鲍(H. fulgens)、西式鲍(H. sieboldii)的引进之后[2], 黑足鲍亦被引入国内养殖。各种鲍的相继引进虽然丰富了我国鲍的养殖品种, 但也存在对本地鲍种质资源造成遗传污染等危险。目前国内外关于黑足鲍分子遗传学水平上的研究很少, 且大部分研究集中于种群评估、病理学和生长发育方面[3~8]。因此, 运用分子生物学技术,选取合适的遗传标记, 对其野生种的遗传背景进行了解显得尤为必要。
线粒体基因(mtDNA)的蛋白基因细胞色素 C氧化酶亚基Ⅰ(COⅠ)基因进化速度适中, 具有较强的种间解析和鉴别能力, 因而在动物的种类鉴定、系统发生及进化遗传学等研究领域中作为分子标记而得到广泛应用。
作者对黑足鲍的 COⅠ基因在遗传分析中的作用作了初步研究。以期为了解其遗传学背景提供资料, 也为水产养殖引种、遗传育种、种质资源鉴定以及苗种放流增殖效果的监测等提供科学依据。
1 材料和方法
1.1 实验材料
2007年从新西兰南岛中部引进的野生黑足鲍,共37个个体, 于−20℃冰冻保存。实验所用组织为黑足鲍腹足肌肉。
1.2 实验方法
1.2.1 基因组DNA的提取
黑足鲍组织消化采用Hye-Suck等[9]的研究方法,并稍加改进。基因组抽提采用酚、氯仿/异戊醇和改进的CTAB法混合(CTAB抽提液: 1mol/L NaCl, 2%CTAB)的抽提方法[10]。
1.2.2 PCR扩增
引物序列为: COⅠF: 5′TGATCCGGCTTAGTCGGACTGC3′, COⅠR: 5′GATGTCTT GAAATTACGGTCGGT3′[9,11~12]。反应总体积为 25 µL, 其中 10~100 ng模板DNA, PCR Mix(由广州东盛生物科技有限公司提供), 0.6 µmol/L引物。94 ℃预变性 5 min, 94 ℃变性50 s, 46 ℃复性45s, 72 ℃延伸45s, 30次循环后72 ℃ 延伸 10 min。
1.2.3 测序
所有样本均在上海生工生物工程技术服务有限公司用ABI-377基因分析仪进行双向测序。
1.2.4 数据处理与分析
用BioEdit软件对双向测序后得到的序列进行剪切和拼接、核对; 同时对照测序的峰形图对序列进行人工核对。利用两端的引物和 Metz等在 GenBank上提交的黑足鲍COⅠ序列(登录号: AF060854)对所得序列进行最后整理。采用 ClustalX软件进行多序列比对, 作基因片段的碱基组成、核苷酸变异等初步分析。利用分析软件DnaSP Version 4.10.9计算种群的遗传多样性, 包括核苷酸多样性和单倍型多样性。同时, 运用分析软件MEGA3.1, 以GenBank上提交的贻贝为外群(登录号: AY377727), 构建 Neighbor-Joining树和Maximum Parsimony树。
2 结果与分析
引物COⅠF和COⅠR具有很高的特异性, 目的基因片段经 PCR扩增后得到清晰的电泳谱带, 分子质量大小在580 bp左右, 扩增产物凝胶电泳谱带见图1。
图1 mtDNA COⅠ序列扩增结果Fig. 1 The Amplification results of mtDNA COⅠ sequence
2.1 碱基组成分析
经序列测定, 获得一段长约 550 bp的特异性COⅠ基因片段, 参照GenBank中的黑足鲍COⅠ序列AF060854, 每段序列剪接到524 bp。经分析得到37条序列中4种核苷酸A、T、G、C的含量(表1)。个体之间 4种碱基的含量非常相近, 而且所有个体A+T的含量相等, 都为53.8%, 高于GC含量。较高的A+T含量符合mtDNA碱基组成的特点。
2.2 核苷酸变异及遗传多样性分析
在37个黑足鲍样本的序列中共检测出3个变异位点, 是这段长度的0.57%(3/524), 其中一个为颠换,另两个为转换, 没有发现插入或缺失突变的核苷酸位点; 这3个变异位点定义了3个单倍型(表2)。其中, 32个样本为单倍型 HapA, 2个样本为单倍型HapB, 3个样本为单倍型HapC。实验中黑足鲍的单倍型特别少, 不同单倍型之间也只有 1~2个碱基的差异。同时这 3个单倍型在个体中的分布明显不均衡, 单倍型HapA出现的频率远远高于单倍型HapB与HapC。
经分析得到核苷酸多样性指数为0.000 79、单倍型多样性指数为0.249、平均核苷酸差异数为0.411,可见黑足鲍的遗传多样性低。
2.3 系统发育关系
以贻贝为外群构建的NJ和MP系统发育树显示结果相似, 此处只列出NJ树(图2)。黑足鲍处于相对适中的位置。属内, 黑足鲍与弗吉尼亚鲍(H.virginea)亲缘关系最近。种内, 本实验获得的单倍型HapC与GenBank上提交的单倍型AF060854最近。
3 讨论
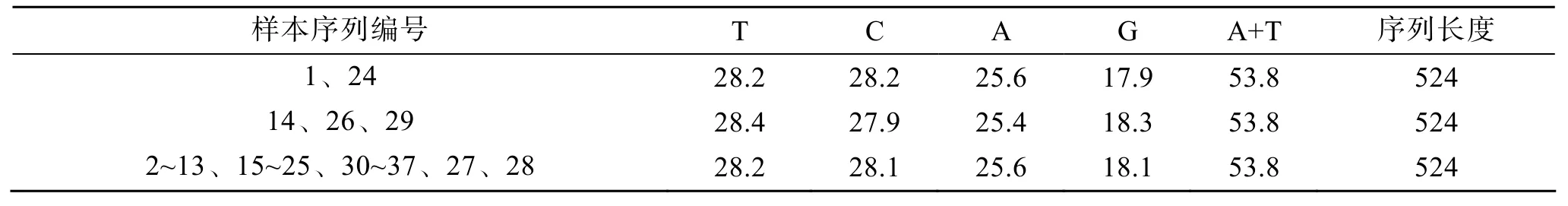
表1 37个黑足鲍个体的COⅠ基因片段的碱基组成(%)Tab. 1 Base composition(%) of mtDNA COⅠ fragment sequences in 37 Blackfoot abalones
基因测序与RAPD、RFLP 等其他分子遗传方法相比, 能提供更加准确的遗传信息。不同的基因对同一物种具有不同的解析能力, 同一基因在不同的物种间解析能力也有差异[2]。Mather等[13]发现, 同一地理种群的罗氏沼虾 (Macrobrachium rosenbergii),COⅠ基因的平均序列差异可达到 15%~16%。冯建彬等[14]在对我国五大淡水湖日本沼虾(Macrobrachium nipponense)的COⅠ基因序列研究中, 100个野生个体的578 bp核苷酸片段中有49个变异位点, 得到了35个单倍型, 且各种群都具有较高的单倍型多态性和核苷酸多态性。
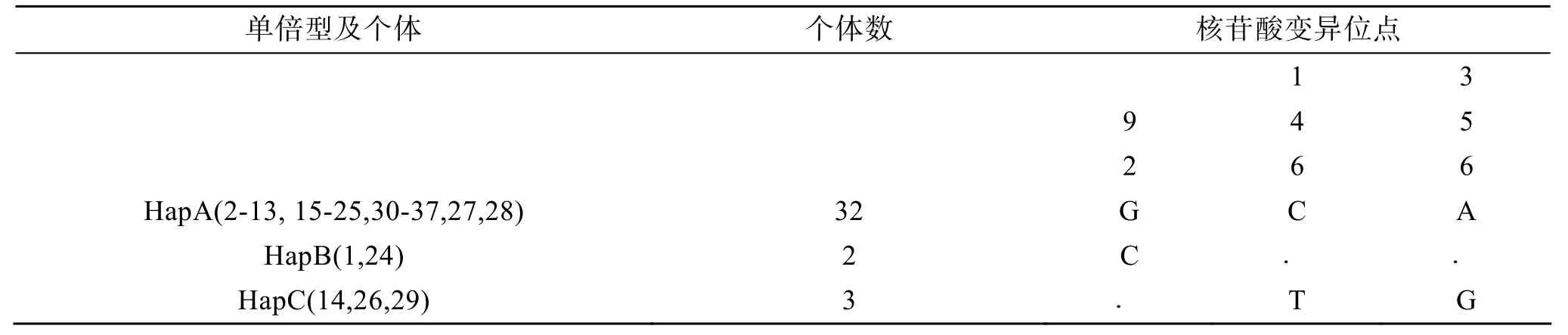
表2 黑足鲍COⅠ基因片段的核苷酸变异位点及单倍型频率Tab. 2 Variable sites within the sequences of the Blackfoot abalone and the distributions of haplotypes among different individuals
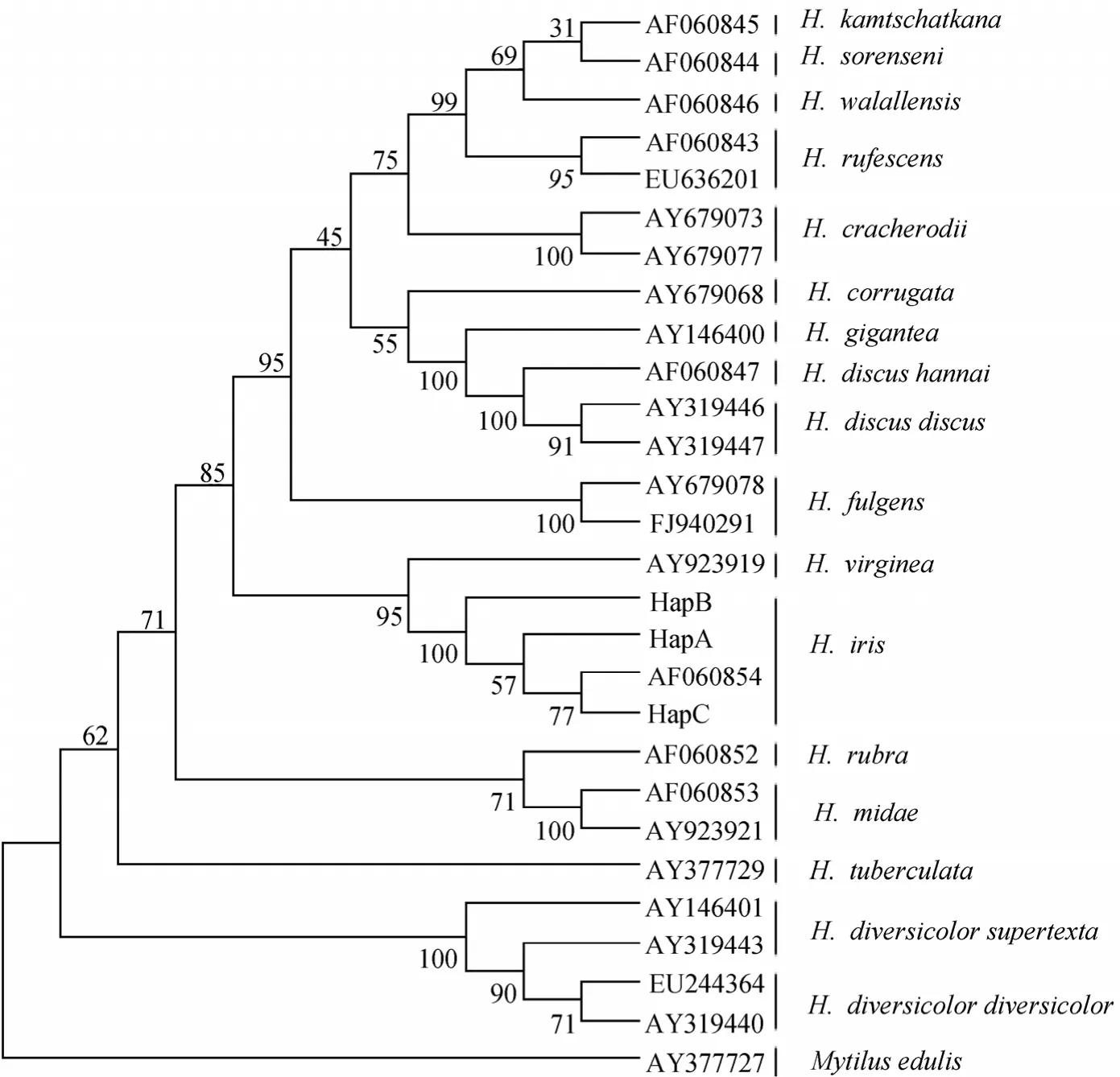
图2 黑足鲍与同属其他鲍的COⅠ系统发育树(NJ树)Fig. 2 Neighbor-Joining Tree of Phylogenetic Relationships of Blackfoot abalone Haplotypes in the experiment and other abalones submitted in GenBank based on COⅠ
在本实验的 37条新西兰黑足鲍的 524 bpCOⅠ序列中, 仅发现3个变异位点, 得到3个单倍型, 单倍型之间的碱基差异只有 1~2个。同时遗传多样性指数也都较低, 其中单倍型多样性和核苷酸多样性分别为 0.249和 0.000 79, 平均核苷酸差异数为0.411。由以上实验数据可以看出, COⅠ基因在37个野生黑足鲍中表现出了较低的序列差异, 反映了 37个个体 COⅠ基因片段进化速率具有一致性以及COⅠ基因片段在进化过程中具一定的保守性, 不能很好地揭示种内的遗传分化, 可能更适用于种以上阶元的遗传多样性分析。
致谢:实验工作得到陶翠花, 刘莹莹, 赵琦, 张婷,张文学, 妙星, 许敏的帮助, 论文写作得到周灿的帮助, 谨此一并致谢。
[1] 侯旭光. 世界重要经济鲍的生物学特征[J]. 齐鲁渔业, 1998, 15(4): 20-22.
[2] 杨建敏, 郑小东, 王如才, 等. 3种鲍16S rRNA基因片段序列的初步研究[J]. 青岛大学学报, 2003, 33(1):36-40.
[3] Breen P A, Andrew N L, Kendrick T H. The 2000 stock assessment of paua (Haliotis iris) in PAU 5B and PAU 5D using a new length-based model [R]. Ministry of Fisheries. New Zealand Fisheries Assessment Report,2000.
[4] Breen P A, Kim S W. The 2004 stock assessment of paua (Haliotis iris) in PAU 4 [R]. Ministry of Fisheries.New Zealand Fisheries Assessment Report, 2004.
[5] Breen P A, Kim S W. The 2005 stock assessment of paua (Haliotis iris) in PAU 7 [R]. Ministry of Fisheries.New Zealand Fisheries Assessment Report, 2005.
[6] Breen P A, Smith Adam N H. The 2007 assessment for paua (Haliotis iris) stock PAU 5B (Stewart Island) [R].Ministry of Fisheries. New Zealand Fisheries Assessment Report , 2008.
[7] Diggles B K, Nichol J, Hine P M, et al. Pathology of cultured paua Haliotis iris infected with a novel haplosporidian parasite, with some observations on the course of disease [J]. Diseaes of Aquatic Organisms,2002, 50: 219-231.
[8] 吴永沛, Shiel D R. 不同蛋白源饵料对虹鲍生长的影响[J]. 海洋科学, 2000, 24(1): 2-6.
[9] Hye-Suck A, Young-Ju J, Kwang-Sik M, et al. Phylogenetic analysis of six species of Pacific Abalone(Haliotidae) based on DNA sequences of 16S rRNA and cytochrome c oxidase subunit Ⅰ mitochondrial genes [J]. Marine Biology, 2005: 373-380.
[10] 李联泰, 安贤惠. 几种海水经济贝类 DNA提取方法探讨[J]. 淮海工学院学报(自然科学版), 2005, 14(4):66-69.
[11] Hamm D E, Burton R S. Population genetics of black abalone, Haliotis cracherodii, along the central california coast [J]. Journal of Experimental Marine Biology and Ecology, 2000, 254: 235-247.
[12] Gruenthal K M, Burton R S. Genetic diversity and species identification in the endangered white abalone(Haliotis sorenseni) [J]. Conservation Genetics, 2005,6: 929-939.
[13] Mather P B, Bruyn M. Genetic diversity in wild stocks of the giant freshwater prawn (Macrobrachium rosenbergii): Implications for aquaculture and conservation[J]. WorldFish Center Quarterly, 2003, 26(4): 4 - 7.
[14] 冯建彬, 孙悦娜, 程熙, 等. 我国五大淡水湖日本沼虾线粒体 COⅠ基因部分片段序列比较[J]. 水产学报, 2008, 32(4): 517-525.
Received: Dec., 12, 2009
Key words:Blackfoot abalone (Haliotis iris); COI; genetic diversity; phylogeny
Abstract:Genetic analysis of 37 Blackfoot abalones from New Zealand was studied based on cytochrome c oxidase subunit I (COⅠ) of mitochondrial DNA (mtDNA). In 524 base pairs, three variable sites and three haplotypes were detected. The haplotype and nucleotide diversity were 0.249 and 0.000 79, respectively, indicating that the genetic diversity of Blackfoot abalone was very low. Blackfoot abalone lies in the middle position of the phylogenetic tree constructed with Mussel (Mytilus edulis) as outgroup. It was expected that these results can be used for further studies of genetic background and COⅠgene of Blackfoot abalone.
(本文编辑:梁德海)
Genetic analysis of blackfoot abalone (Haliotis iris) based on COⅠgene fragment
GAO Hai-xia1, HOU Xu-guang1, ZHU Qian1,2
(1. Ocean College, Shandong University at Weihai, Weihai 264209, China; 2. Third Institute of Oceanography,State Oceanic Administration, Xiamen 361005, China)
Q812
A
1000-3096(2010)11-0014-04
2009-12-12;
2010-03-11
国家海洋局项目(HC10301-10, HD10301-10); 国家海洋局第三海洋研究所项目(HE09701(1), HE09702(1))
高海霞(1984-), 女, 山东滨州人, 硕士研究生, 主要从事保护生物学研究, E-mail: bluocean200@yahoo.com.cn; 祝 茜, 通信作者,教授, 博士, 主要从事海洋动物保护生物学和视觉生物学研究, 电话:0631-5688004, E-mail: qianzhu@sdu.edu.cn
