生物医学异构数据库集成的研究进展
2010-09-18张正国
张 智 张正国
(中国医学科学院基础医学研究所 北京协和医学院基础学院,北京 100005)
生物医学异构数据库集成的研究进展
张 智 张正国*
(中国医学科学院基础医学研究所 北京协和医学院基础学院,北京 100005)
随着生物医学的快速发展,不断地涌现出许多生物医学数据库。将这些相互独立的数据库有机地组织在一起,对于提高一个学科或领域的整体知识水平,以及对该学科或领域进行更深入、更全面的理解是十分重要的。数据集成可以实现数据更广泛的共享和更有效的利用,已经成为生物信息学的核心研究内容之一。介绍生物医学异构数据库集成的方法,综述生物医学异构数据库集成领域内最新的研究进展,并且讨论和总结各种方法的特点及使用条件。
异构数据库;数据集成;数据仓库;联邦数据库;中间件
引言
随着生物医学知识的快速增长、计算成本的降低以及互联网在传播媒介上的优势,生物医学数据已呈现海量规模,并且其数据量还在加速增长。截止到2010年1月,权威的“核酸研究在线分子生物学数据库集合”(Nucleic Acids Research online Molecular Biology Database Collection)共收录了1 230个分子生物学数据库[1]。但是,这些生物医学数据库大多相互隔离,形成了所谓的“信息孤岛”,不能实现数据的共享和更有效的利用。将这些相互独立的数据库有机地组织在一起,对于提高一个学科或领域的整体知识水平,以及对该学科或领域进行更深入、更全面的理解是十分重要的[2]。生物医学由于其复杂性,既需要多学科、多方位进行地研究,又需要综合多方面的观察进行分析。生物医学数据库常是细分的,而需求常是综合的,因此数据集成对于生物医学具有必要性和紧迫性。现在,数据集成已经成为生物信息学(bioinformatics)的核心研究内容之一,也是一项需要长期进行的工作[3]。
数据集成(data integration)是把不同来源和不同格式的数据在逻辑上或物理上有机地集中,从而实现全面的数据共享。数据集成的核心任务是将相互关联的异构数据源集成到一起,使用户能够以透明的方式访问[4]。集成是指维护数据源在整体上的数据一致性,为用户提供统一的数据访问接口,提高数据共享的效率;透明的访问方式是指用户无需关心所需数据所属数据源的位置以及如何访问数据源,只需关注对数据的需求和操作。
由于生物医学数据库固有的特点,其数据集成工作困难重重[5]。第一,生物医学数据具有多样性和复杂性。数据的实体类型包括基因表达、序列、结构和图像等多种,而且这些实体之间通常还有复杂的关系。第二,生物医学数据的数据量通常很大。第三,生物医学数据库具有异构性。不同数据库之间可能同时存在系统性异构、技术性异构和语义性异构等多种异构。第四,生物医学数据库具有自治性。绝大多数的生物医学数据库可以自由地删除数据和修改数据库模式,而不必考虑其他与其相关的数据库。其数据也按照各种自定的格式或标准进行存储。
近年来,越来越多的研究者致力于生物医学异构数据库集成的研究。这些研究面向的领域也更加广泛,涉及基因组学、蛋白质组学、相互作用组学、医学图像和临床医学等多个方面,并产生了丰硕的研究成果。文中介绍了生物医学异构数据库集成的方法,综述了生物医学异构数据库集成领域内最新的研究进展,并且讨论和总结了各种方法的特点及使用条件。
1 生物医学数据库的异构性
数据库的异构是生物医学数据库集成的难点和重点。数据库的异构体现在以下三个方面:系统性异构(systematic heterogeneity)、技术性异构(technical heterogeneity)[6]和语义性异构 (semantic heterogeneity)[6]。
系统性异构包括:一是数据库所依赖的计算机体系结构不同,如大型机、小型机和 PC服务器等;二是数据库所依赖的操作系统不同,如 Linux、Windows和Unix等;三是数据库所依赖的网络平台的不同,如ATM(异步传输模式)、Ethernet(以太网)和FDDI(光纤分布式数据接口)等。
技术性异构包括:一是数据存储方式不同,如普通文本文件,XML文件,关系型、层次型、面向对象型、网络型或函数型数据库等;二是数据访问方法不同,可以通过HTTP等网络协议访问,也可以通过JDBC、ODBC和 SOAP等编程接口访问;三是数据查询语言不同,如SQL、OQL和XPath/XQuery等。特别地,不同数据库支持相同查询语言的标准和程度也不尽相同。
语义性异构包括:一是数据库模式(schema)不同,不同数据库字段的数据类型不同、名称不同以及存在语义差异等;二是实体命名不同,不同数据库使用了形式不同,但彼此等价的规范化词表(controlled vocabulary)和本体(ontology)作为实体的标识符。
在实际情况中,生物医学数据库之间往往同时存在多种异构,这更加造成了数据集成工作的困难与复杂程度。因此,解决异构问题,屏蔽各局部数据源的异构性,构建与平台和系统无关的查询平台,是数据集成最重要的任务。
2 生物医学异构数据库集成解决方案
2.1 基于数据仓库的解决方案
基于数据仓库(data warehouse)的数据集成是对异构数据源的物理式集成,其系统结构如图1所示。该系统主要是使用ETL(extract-transform-load)工具,对各个异构数据源中的数据进行抽取、转换,并在通过集成器进行消除数据异构性后,将数据物理地装载到数据仓库中。特别地,在将数据装载到数据仓库之前,需要经过严格的数据清洗(data cleansing),以提高数据的质量。数据仓库的构建,实际上就是各个异构数据源模式转换为公共数据模式(common data schema)的过程。当用户直接面向数据仓库进行查询时,各个数据源的异构性已经消除,这使得检索过程更加简单和快速。为了确保数据仓库中的信息与各个数据源中的信息保持一致,必须定期更新数据仓库。在实际应用中,EnsEMBL、NCBI和UniProtKB等大型数据集成系统都是根据基于数据仓库的方法构建的。
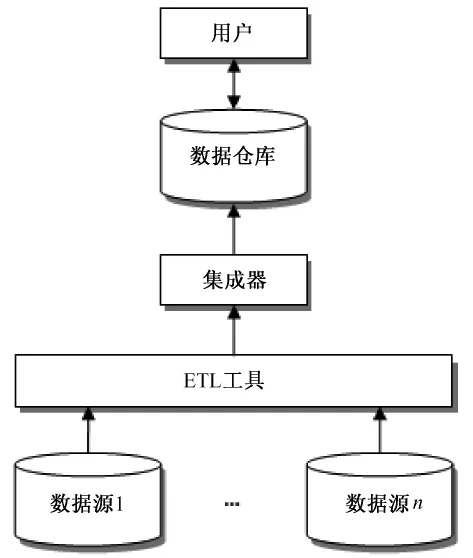
图1 基于数据仓库方法的数据集成系统架构Fig.1 The architecture of data integration system based on data warehouse
Trissl等人基于数据仓库的方法集成若干蛋白质结构信息相关数据库,建立了 Columba系统[7]。Columba通过物理的方式集成了12个异构数据库,构成了一个数据仓库,其数据涵盖了蛋白质基于结构和序列的分类信息、蛋白质功能注释信息、蛋白质二级结构信息和代谢通路信息。Columba已经被证明在许多蛋白质结构的相关研究中发挥了重要作用,是一个成功和成熟的异构数据集成系统。在此基础上,Columba已经准备继续集成 LIGAND,MEDLINE和OMIM等数据库,拓展该数据仓库的应用。Chaurasia等人建立了用于查询、分析和可视化人类蛋白质相互作用组的数据仓库 UniHI[8]。UniHI集成了14个异构蛋白质相互作用数据库以及GO和OMIM等辅助数据库。UniHI系统结构分为数据集成、数据仓库和网络应用程序3个部分。数据集成部分负责通过解析器,将异构数据源的数据转存到临时数据库,此时已经通过应用全局数据模式消除了数据的异构性;然后,对临时数据库中的数据进行数据清洗、ID转换等修饰性工作;最终,将修饰好的数据导入数据仓库中。用户通过网络应用程序接口访问该数据仓库,获取相应信息。UniHI通过数据仓库的方式完整、一致地描述了人类蛋白质相互作用组领域的数据,其数据规模很大、数据质量很高,是一个成功的数据集成案例。Tarcea等人基于数据仓库的方法构建了分子相互作用的数据集成系统 MiMI[9]。MiMI采取了深度集成(deep-merging)的策略,即在消除数据异构性的基础上,还要进一步消除冗余的数据,消除相互矛盾的数据,将相关的数据进行连接并追踪数据的来源。其他数据集成方法由于是在程序运行阶段进行集成,因此无法做到如此程度的深度集成。由此可见,只有使用基于数据仓库的方法,才能在构建数据仓库的过程中进行如此程度的深度集成,进而提高数据集成的质量。MiMI成功地集成了10个分子相互作用异构数据库。但并不急于寻找新的数据集成到数据仓库中,而是通过完善深度集成的策略,强调增强对已知数据的更完善的使用。
2.2 基于联邦数据库系统的解决方案
联邦数据库系统(Federated Database System,FDBS)是一种元数据库管理系统,负责将多个自治数据库以透明、虚拟的方式集成到一个联邦数据库。各自治数据库通过计算机网络互联,相互提供访问接口,相互分享数据。对这些自治数据库的操作进行控制和协调的软件,称为联邦数据库管理系统(federated database management system,FDBMS)。FDBMS是一个管理中心,它能把各成员数据库模式映射到一个公共联邦模式,负责各种模式之间的转换工作,自动解决网络传输问题和异构数据库操纵问题,接受联邦用户的数据请求,把这个请求翻译后送到各个目的数据库引擎,并将收到的结果数据集成后返回给用户。其一般结构如图2所示,它通过包装器与各异构数据源进行通信。包装器从FDBMS接受数据访问指令,进而转换为各异构数据源所支持的数据访问指令,然后通过各异构数据源的服务器提交执行,最后将结果返回给 FDBMS进行异构数据集成处理。除了包装器之外,FDBMS还有两个核心部分。一个是全局的元数据字典,用于描述数据集成的公共联邦模式;另一个是配置数据表,其中包括各异构数据源的配置信息、模式、字段数据类型、相应的索引、数据分布的统计信息,以及CPU、网络、I/O等系统资源情况。

图2 基于联邦数据库系统的数据集成系统架构Fig.2 The architecture of data integration system based on federated database system
Muilu等人使用联邦数据库,将欧洲600 000个孪生双胞胎的基因组和表型组数据进行了集成,构建了TwinNET系统[10],结构如图3所示。TwinNET采用IBM的DB 2数据库和IBM的联邦服务器Discovery Link作为异构数据集成系统的基础。8个国家的表型组数据库通过虚拟专用网络(virtual private network,VPN),连接到表型数据库集线器(Hub)。表型数据库集线器负责将各个表型组数据库的数据提交到联邦数据库系统中的信息集成器,完成数据集成,基因型数据库也是以此种形式得到集成。用户可以通过网络程序,访问联邦数据库系统,获取集成后的数据。TwinNET十分注重系统的安全性,各个成员数据库通过VPN或安全外壳协议(secure shell protocol/SSH)的方式连接到联邦数据库。联邦数据库与互联网之间有前端防火墙、VPN网关和非军事区3个屏障,联邦数据库服务器位于 TwinNET非军事区(demilitarized zone,DMZ)内,因此系统安全性很好。
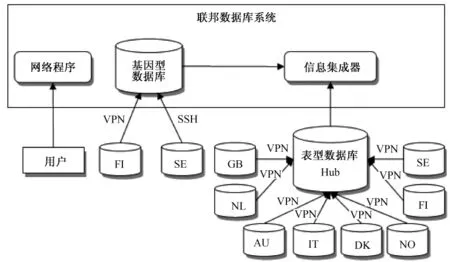
图3 TwinNET系统架构[10]Fig.3 The architecture of TwinNET system[10]
Androulakis等人构建了集成X射线衍射图像异构数据的联邦数据库系统[11],采用了第三方软件框架Fedora成功地构建了该联邦数据库系统。由于图像数据量十分庞大,达到数百GB,因此从技术上和经济上考虑将这些异构图像数据集中存储在本地数据库里是不适合的。同时,由于图像数据量比较大以及处理过程耗时等特点,基于中间件的方法也无法高效地处理数据图像的集成。因此,Androulakis等人选择了基于联邦数据库系统的方法,既考虑了图像数据分布式存储的便利,又兼顾了数据集成的效率问题。
2.3 基于中间件的解决方案
基于中间件(middleware)的数据集成方法通过统一的全局数据模式来访问异构数据库,是模型层的数据集成方案,数据集成中间件位于各异构数据源(数据层)和应用系统(应用层)之间。这种方法的核心是全局模式(global schema):数据集成系统通过一个全局模式,将各异构数据源的数据集成起来,而数据仍存储在局部数据源中。通过各异构数据源的包装器对数据进行转换,使之符合全局模式。用户提交的查询是针对全局模式的,所以用户不必知道数据源的位置、模式及访问方法。按照实现方式的不同,基于中间件的数据集成方法可以分为基于包装器/中介器、基于本体、基于网格和基于XML共4种具体方法。
2.3.1 基于包装器/中介器的方法
基于包装器/中介器(wrapper/mediator)方法的体系结构如图4所示。中介器为应用系统提供统一的全局数据模式和通用的数据访问接口,同时负责协调各异构数据源,为用户提供全局的查询服务。中介器将基于全局数据模式的查询转换为基于各局部异构数据源的子查询,通过针对各异构数据源的包装器执行该子查询并获取查询结果,最终将结果集成后返回给用户。

图4 基于包装器/中介器的数据集成系统架构Fig.4 The architecture of data integration system based on wrapper/mediator
Hwang等使用基于包装器/中介器的方法成功地集成了果蝇相关的蛋白质相互作用数据、基因组数据和其他辅助数据[12]。该研究将蛋白质相互作用数据存储在本地数据库,而使用基于包装器/中介器的方法集成与该数据库中信息相关的基因组数据(GenBank,FlyBase和GadFly)和其他辅助数据(SwissProt和GO),所构建的系统分为包装器模块、中介器模块和应用模块等3个部分。包装器模块根据查询关键词,应用 HTML、XML和 HTML-XML包装器,从待集成的5个数据库的网页上分析并获取所需的信息。中介器模块提供中介服务,将包装器模块和应用模块连接起来,实现数据集成;中介器分析用户的查询请求,将查询任务分派给各个包装器,在得到包装器的返回结果并进行集成后,返回给用户。应用模块负责结果显示和用户交互。这项研究成功地将5个公共数据库和蛋白质相互作用的数据进行了集成。具有将蛋白质组数据集成入该系统的扩展能力,从而形成一个以蛋白质相关研究数据为中心的综合性生物学数据集成系统。
Marenco等人创建了基于包装器/中介器模式的生物医学数据库集成框架 QIS[13],其体系结构如图5所示,核心是3个功能单元:集成服务器、数据源服务器和本体服务器。这3个功能单元的有机统一体现了用户、数据和知识的三位一体结构:集成服务器作为中介器,负责调控另外两个功能单元,进行数据集成以及与客户端的交互;数据源服务器作为包装器,负责对各个异构数据源进行结构化查询;本体服务器负责管理UMLS本体,为集成服务器提供语法式查询,为数据源服务器提供相关数据与本体之间映射的信息。QIS具有很好的泛化能力,可以应用于不同领域的数据集成。在实际应用中,QIS已经成功地应用于神经科学和基因组学。QIS通过引入本体,为数据集成系统提供了在不同粒度下的应用。例如:数据源服务器可以通过本体服务器的辅助,掌握哪一个数据源中含有神经元细胞的相关信息,而哪一个数据源中含有某一种特定的神经细胞,如小脑浦肯野细胞的相关信息。特别地,通过引入UMLS本体,对于临床医学信息学数据的集成有着重大的意义。
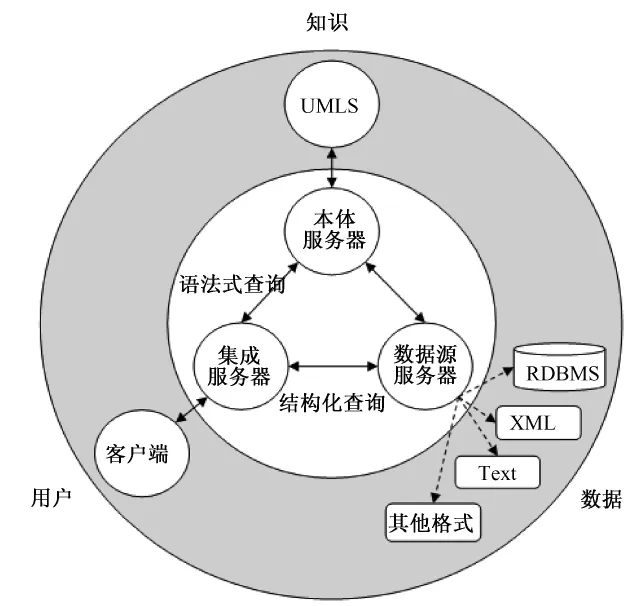
图5 QIS框架的系统架构[13]Fig.5 The architecture of QIS framework[13]
Blankenburg等人构建了用于集成异构分子相互作用数据的 DASMI系统[14],该系统基于分布式注释系统(distributed annotation system,DAS)[15]。DAS封装了包装器/中介器方法,用户只需要按照DAS规范调用即可实现包装器/中介器方法。DASMI系统由数据交换规范和提供相互作用数据集成的客户端两个部分组成,采用非集中化的(decentralized)系统架构(见图6),提供在线的从分布式异构数据库中获取的最新数据。各个相互作用数据服务器提供相互作用的数据,可信度评分服务器提供对于相互作用数据的评分。DASMI客户端查询相互作用数据服务器,并集成查询结果;DAS客户端分为基于网络程序的客户端和基于软件插件的客户端两种类型。DAS注册表用于维护互联网上可用的 DAS服务器列表,通过注册新的 DAS服务器,就可以方便快捷地实现 DASMI系统的扩展。DAS服务器与客户端的数据交换需要符合DAS的 URL规范和 XML规范。DAS客户端通过HTTP协议向DAS URL对应的DAS服务器发送查询请求,DAS服务器通过DASINT XML协议响应该请求并返回数据,从而完成一次数据交换。因此,DAS的 URL规范和 XML规范是 DASMI系统实现分布式异构数据集成的基础和关键,DASMI系统可以对所有实现了DAS服务的相关相互作用数据库进行有效集成。由于采用了非集中化的系统架构和数据交换规范,DASMI系统本身并不需要存储任何相关数据,有效地降低了系统的维护成本,同时提高了系统的灵活性和可配置性。Blankenburg等人在DASMI系统的基础上,又开发了在线集成、分析和评估分布式异构数据库的 DASMIweb系统[16]。该系统采用基于网络程序的DAS客户端,专门面向蛋白质相互作用异构数据的集成;集成了35个蛋白质相互作用的相关数据库,具有灵活、高效的特点,并具备一定的扩展能力。但是,由于采用了DAS系统的方式进行集成,所以该系统集成的对象只能是实现了DAS服务的数据库,对于其他没有实现该服务的数据库无能为力。DASMIweb系统扩展能力的高低,取决于科学共同体对DAS服务的支持力度。
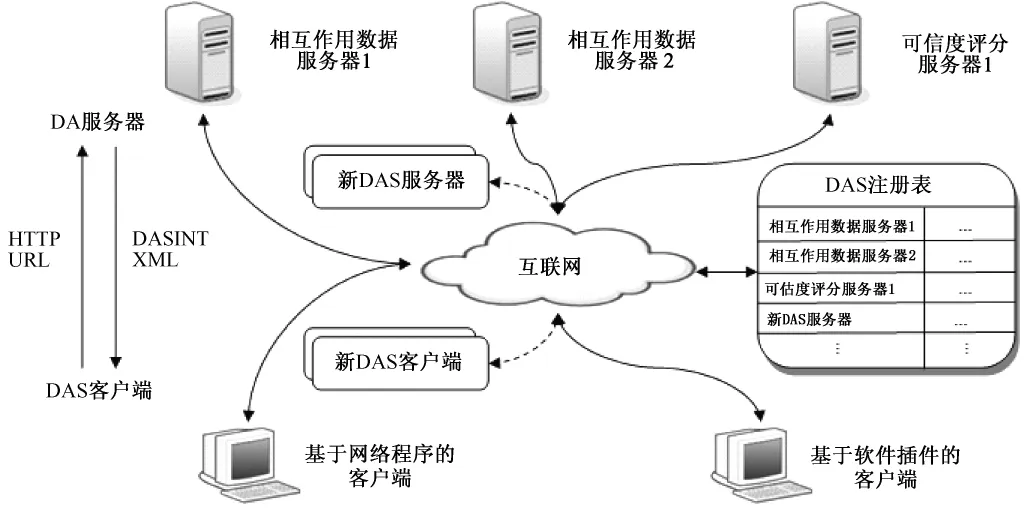
图 6 DASMI系统架构[14]Fig.6 The architecture of DASMI system[14]
2.3.2 基于本体的方法
本体对特定领域的实体给出名字和描述,使用谓词来表示这些实体间的关系。本体具有描述数据源的语义和解决数据源异构问题的潜力,因此可以用于异构数据集成。基于本体的数据集成中间件通过将各异构数据库的模式映射到本体,实现了不同数据源间的语义一致,完成了语义层次上的数据集成。基于本体的数据集成的基本思想:一是在领域专家的帮助下,建立相关领域的本体;二是收集数据源的数据模式,并参照已建立的本体,把数据源模式与本体间的映射信息按规定格式存储在元数据库中;三是对用户的查询请求,查询转换器按照本体把查询请求转换成规定的格式,在本体的帮助下从元数据库中匹配出符合条件的数据源集合,进而实施查询;四是将查询结果经过定制处理,返回给用户。
Köhler等建立了一个基于本体实现生物学数据库语义集成的系统SEMEDA[17],其总体策略是将数据库的表和属性映射到一个本体,而本体则需要实现“is a”的层次结构,图7显示了通过所建立的本体集成 Enzyme和 Vertebrate两个数据库的思路。首先,将两个数据库中表和属性映射到本体,用本体提纲挈领地通过语义方式将所有表和属性串联到一起,形成语义网。同时,通过规范词表(controlled vocabulary),对数据库表的属性值,进行语义规范,以进行一致性的语义查询,如图7中所示的ec nr和ec_nr两个属性被规范化为规范词表中的“ECID”项。基于本体的语义集成方法特别适用于大量异构数据库的集成,能够通过本体对数据集成系统进行管理,十分灵活,可以方便地添加和删除数据源。同时,该方法不需要维护复杂的集成模式,避免了相应的问题。
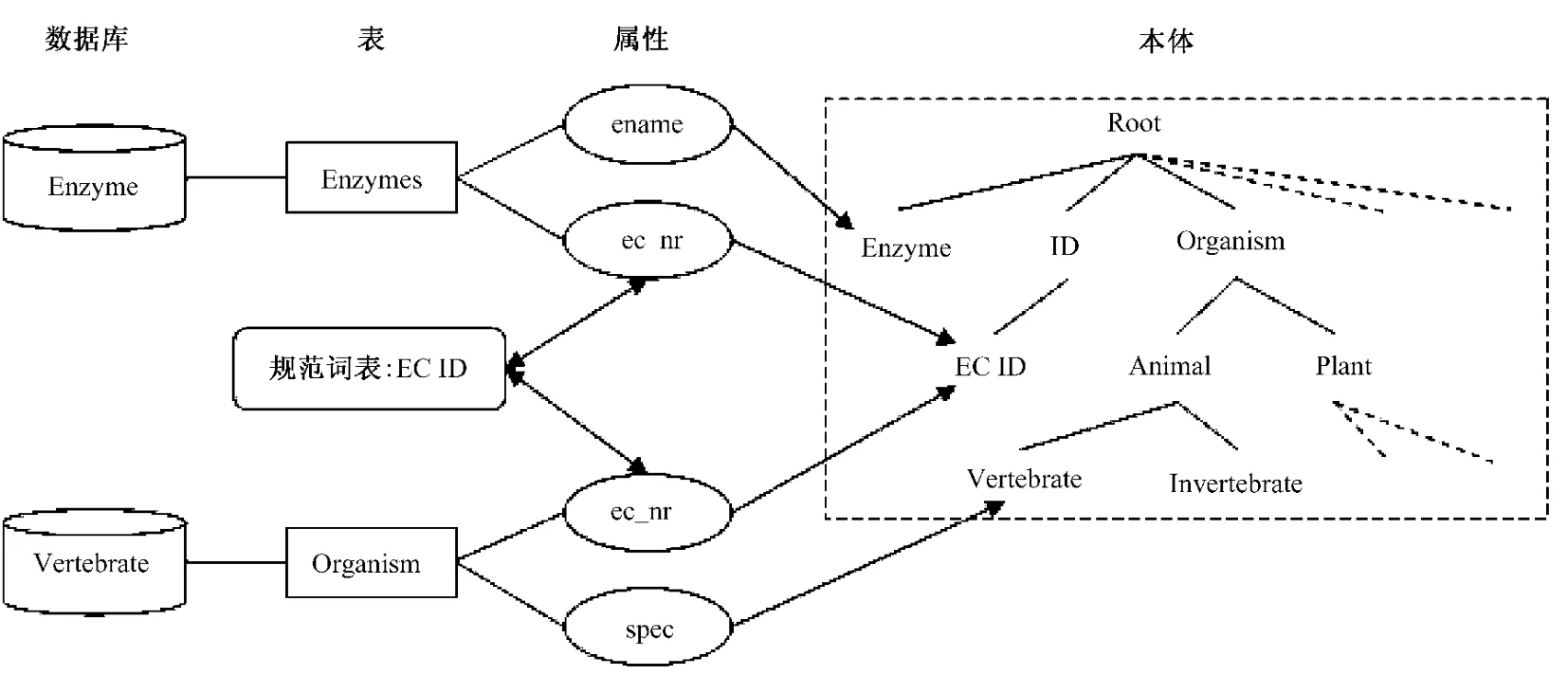
图7 SEMEDA系统的集成思路[17]Fig.7 The integration way of SEMEDA system[17]
Alonso-Calvo等人建立了基于本体和代理(agent)的基因、蛋白质和疾病相关异构数据库集成系统 OntoFusion[18],结构如图 8 所示。OntoFusion系统构建在多agent系统JADE基础上,共有4个模块:用户界面、词表服务模块、中介器模块和数据访问模块。系统的核心模块是中介器模块,负责提供对各异构数据库的一致性访问。词表服务模块负责维护和提供医学和遗传学本体,数据访问模块实现对公共和私有生物医学数据库的查询,用户界面模块包括用户接口和管理模块。OntoFusion系统采用了多agent架构,使得其各个模块可以运行于不同的计算机上,增强了系统的并行处理能力和灵活性。OntoFusion系统提供了独立的词表服务模块,既可以用于数据集成,完成各异构数据库模式之间的映射,又可以直接为用户提供本体数据,使词表服务模块的复用性得到充分体现。
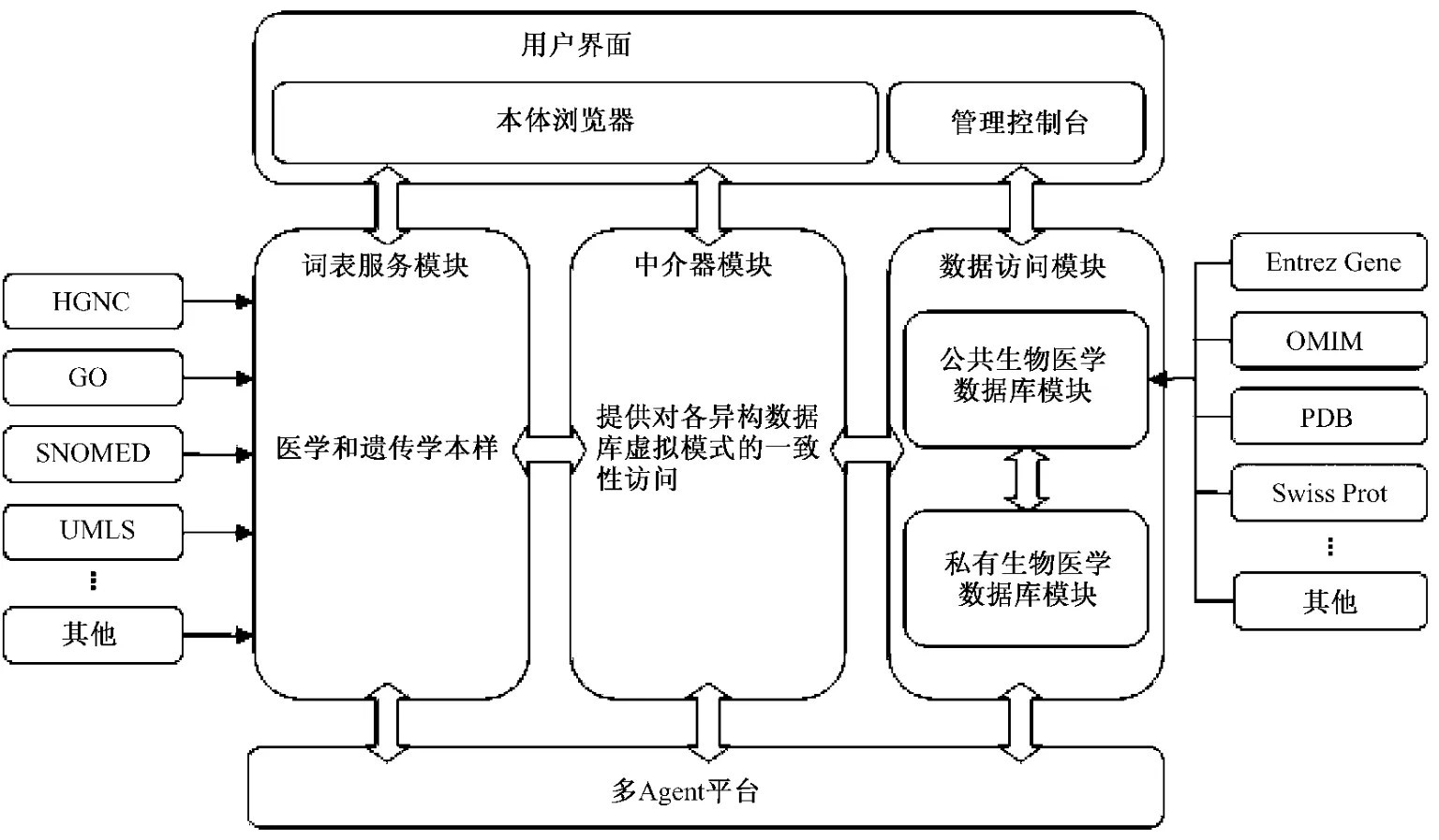
图8 OntoFusion系统架构[18]Fig.8 The architecture of OntoFusion system[18]
Noy等建立了通过本体仓库(repository)集成生物医学数据的系统 BioPortal[19]。BioPortal提供两个主要功能:一是开放的生物医学本体仓库,二是使用该本体仓库进行生物医学数据源的集成。BioPortal的生物医学本体仓库包含134个本体,极大地拓展了数据集成的领域。特别地,BioPortal为编程者提供了用于访问该本体仓库的网络服务(web service),可以方便地为需要该项功能的用户提供服务。通过丰富的本体信息,BioPortal集成了诸如 ArrayExpress,DrugBank,OMIM,PubChem 和UniProtKB等20个不同领域内著名的异构生物医学数据源,体现了强大的适应能力和扩展性。Min等人创立了前列腺癌本体(prostate cancer ontology,PCO),并以此为基础开发了前列腺癌数据集成系统PCIS[20]。PCIS利用PCO为美国著名的福克斯·蔡斯癌症中心(Fox Chase Cancer Center,FCCC),成功地集成了与两个前列腺癌相关的数据库系统。该系统的关键是通过创建PCO与两个异构数据库模式之间的映射关系以达到消除语义性异构的目的。特别地,PCIS使用了一种语义查询语言SPARQL进行查询的构造,这样可以更好地利用PCO语义方面的优势。PCIS是基于本体的数据集成方法在临床医学领域内的成功应用之一。
2.3.3 基于网格的方法
开放网格服务架构-数据访问与集成(open grid services architecture-data access and integration,OGSA-DAI),是一种成熟的基于网格技术(grid technology)的分布式异构数据集成中间件。OGSADAI通过定义数据访问的接口,隐藏数据库驱动、数据传输等技术细节,使用户可以通过统一的网络服务接口连接和使用分布式数据源,使得数据共享和使用更加方便。特别是在互联网飞速发展的今天,基于网格的OGSA-DAI数据集成方法非常适合用于公开在互联网上发布的生物医学数据库的集成。
Crompton等使用基于网格的OGSA-DAI技术进行了生物信息学领域的数据集成[21]。OGSA-DAI客户端按照用户的查询请求,创建网格数据服务,同时调用 Jones等开发的 BDW[22]中间件的DWQueryActivity服务进行查询。BDWQueryActivity使用包装器查询目标数据库,获取查询结果。然后,XSL转换器将结果转换为BDW格式文件,并经过数据集成模块处理,将集成后的结果返回给OGSI-DAI客户端,进而返回给用户。BDW是一个建立生物多样性方面网格系统的中间件,但是Crompton等人将 BDW应用在生物信息学领域,使BDW中间件实现了基于网格的异构数据查询和集成,并取得了成功。Luo等建立了一种基于网格的、用于分布式医学数据库集成的模型[23],所支持的异构数据源类型包括关系型数据库(MySQL、Oracle和SQL Server)及文件数据源,其系统结构如图9所示。该模型使用OGSA-DAI组件提供的网格数据服务(grid data service)作为本系统的核心组件,通过网格系统来访问各分布式数据库,隐藏了这些数据库之间的异构性和动态性。该模型还提供了其他的功能组件用于实现数据集成和数据查询功能。最终,通过应用程序接口可以实现基于网格技术的网络应用程序,供用户使用。该模型使用了网格技术及OGSA-DAI,实现了基于标准框架的分布式数据源的访问和集成。同时,该模型提供了成熟的服务接口,以数据源相互独立的方式支持了对医学数据库的一致性访问。
2.3.4 基于XML的方法
XML(eXtensible Markup Language)可扩展标记语言,以一种开放的自我描述方式定义数据结构,在描述数据内容的同时又能突出对结构的描述,从而体现出数据之间的关系。XML有很强的数据结构表达能力及扩展性,非常适合于解决数据集成中关键的语义异构性问题。可以将异构数据源的数据模式描述为 Schema或 DTD文档,进而转换为XML文档结构,从而实现局部异构数据源的数据模式到全局数据模式的映射与数据的转换。同样具有跨平台特性的XML数据描述技术与Java编程技术的结合,可以很好地解决系统异构性的问题。同时,可以利用基于成熟的XPath技术的XQuery查询语言来访问XML数据。XQuery之于XML相当于SQL之于关系型数据库,可以完成复杂的查询任务。
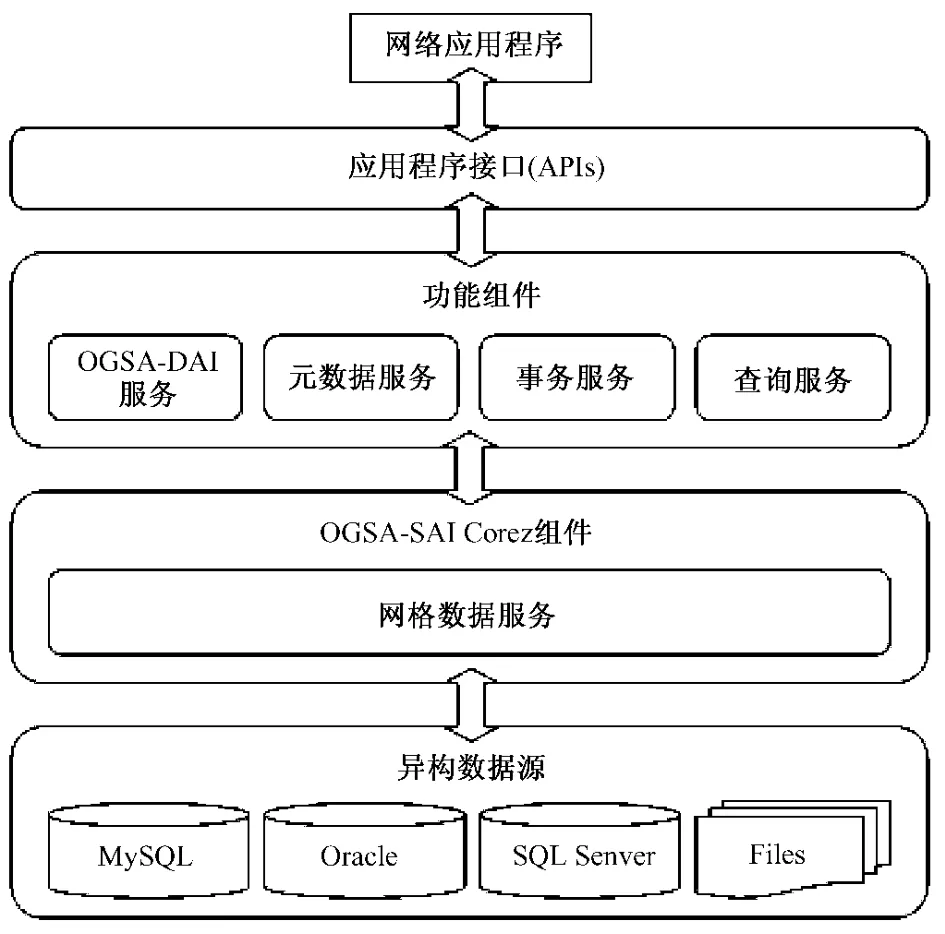
图9 基于网格的数据集成系统架构[23]Fig.9 The architecture of data integration system based on grid[23]
Huang等构建了一个基于XML方法的生物数据集成系统 JXP4BIGI[24]。JXP4BIGI是一个独立于系统的通用框架中间件,其功能包括对异构数据的访问、提取、转换和集成。JXP4BIGI有4个核心组件,即XML生物实体模板,查询/逻辑提取组件,包装器和 JXP处理器。XML生物实体模板也称为Java XML页面(JXP),是用于表示生物实体的数据结构。包装器按照查询/逻辑提取组件的要求,在从异构数据源获取到所需数据后,就将其存储到JXP中。然后,JXP处理器就来分析处理这些 JXP,以抽取出用户所需的数据,返回给用户。由于定义目标生物实体的模板是XML文件,因此可以灵活地定义非常复杂的描述目标实体的结构,为解决数据的异构性奠定了坚实的语义描述基础。JXP4BIGI是基于Java技术开发的,因此具有平台独立性,使其中间件的特点更加鲜明,适用范围更广。Bales等人提出了基于XML的生物医学数据集成框架XBrain[25],该系统结构如图 10 所示。XBrain 所集成的异构数据源分为3种数据模型:关系型(如CSM)、XML类型(如 IM)和本体类型(如OQAFMA)。XBrain采用 XQuery,在分布式条件下的扩展应用 XQueryD,实现数据的获取;利用 XML技术,实现异构数据的集成。XQueryD处理器负责根据用户提交的查询需求,从各个异构数据源获取数据。其中,XQueryD处理器使用 Silkroute[26]完成XML和关系型数据库之间的映射,使用 StruSQL包装器完成XML和本体之间的映射。XBrain可以提供CSV、HTML和XML3种格式的查询结果返回给用户。XBrain使用XML技术集成的数据模型基本涵盖了生物医学数据可能的数据模型,具有数据集成对象的全面性。XBrain使用了成熟的XQueryD技术,构建的应用程序具有很好的性能,特别是稳定性和查询速度。
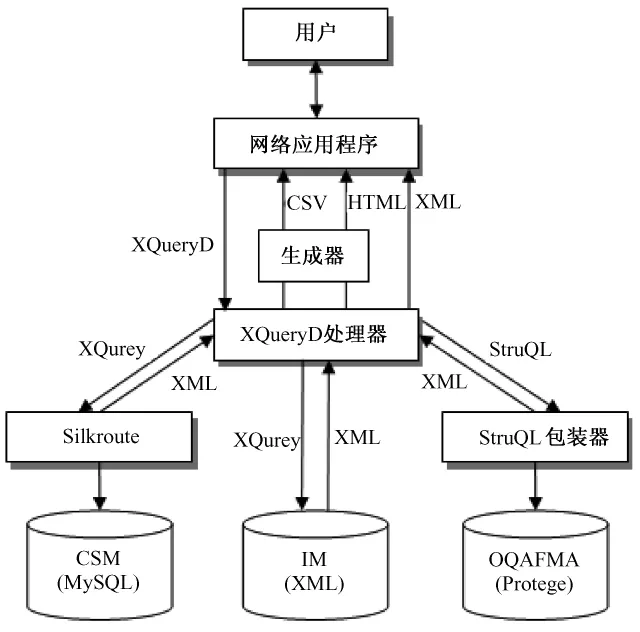
图10 XBrain系统架构[25]Fig.10 The architecture of XBrain system[25]
3 结论
本文综述了生物医学异构数据库集成领域内的最新研究进展,对基于数据仓库、联邦数据库系统和中间件的3种方法及其相关研究进行了介绍和讨论。下面将对这些方法的特点和适用性进行总结。
数据仓库通常利用成熟的关系型数据库,因此处理海量数据具有优势。利用数据仓库还可以对数据进行预处理,特别是数据清洗,以提高数据的质量。可以利用数据仓库提供的工具进行联机分析处理(OLAP)和数据挖掘,对数据进行多层次、全方位的有效利用。数据仓库通常在本地存储,而且对其中的数据进行了预处理,因此实现了以磁盘的空间换取了用户查询的时间,提高了数据存储和查询的效率。数据仓库增加了数据的可获取性,当使用其他两种方法时,如果某一个外部数据源暂停数据访问服务时,数据集成系统的数据来源就减少了一个,而数据仓库则不会发生这种情况。但是,数据仓库也存在一些不足:其架构不够灵活,不能及时反映所集成数据源的改变;建立数据仓库的成本较高;随着数据量的不断增长,存储设备不断增加,数据更新和维护逐渐困难。
联邦数据库系统着重实现各异构数据源之间的互操作,其最常用的方法是将每个数据库模式分别和其他所有数据库模式进行映射。如果有n个自治数据库,则联邦中需要建立 n(n-1)个模式映射规则。但是,当参与联邦的数据库很多(n值很大)时,建立映射规则的任务将变得很复杂而难于实现和维护。所以,联邦数据库集成系统适用于自治数据库的数量比较少、各数据库有较好的自治性、允许用户单独查询、各数据库间能够彼此联合回答查询的情况。
中间件是目前比较流行的数据集成方法。由于每次查询都要连接到各数据源,因此该方法可以很好地反映所集成数据的实时性。该方法可以集成非数据库数据源,如半结构化数据。基于中间件的数据集成系统中的数据源的查询能力可以是受限的,如可以不支持SQL查询。因为这些数据源是完全自治的,所以很容易对系统中数据源进行添加或删除,这使得系统的数据维护工作大大减少、系统的灵活性大大加强。该方法也存在一些不足之处。基于该方法建立的数据集成系统通常是只读的,而数据仓库和联邦数据库系统既可读也可写;不支持各异构数据源之间的互操作;因为该方法是基于程序运行时的数据集成方法,所以受程序运行效率的限制,很难完成在线的数据清洗等维护数据质量的工作;该方法可能引发原始数据源非故意的拒绝访问,此时系统所集成的数据源就会减少。例如NCBI严格限制用户每天访问的次数,如超限,可能会被暂时禁止访问。当数据源的查询能力受限时,如何处理查询和进行优化也是亟需完善的工作。
[1] Cochrane GR and Galperin MY.The 2010 Nucleic Acids Research Database Issue and online Database Collection: a community of data resources[J].Nucleic Acids Research,2010,38(Database issue):D1-D4.
[2] Karasavvas KA, Baldock R, Burger A. Bioinformatics integration and agent technology[J].J Biomed Inform,2004,37(3):205-219.
[3] Goble C,Stevens R,Hull D,et al.Data curation + process curation= data integration + science [J]. Briefingsin Bioinformatics,2008,9(6):506-17.
[4] Maurizio L.Data integration:a theoretical perspective[A].In:Proceedings the ACM Symposium on Principles of Database Systems[C].New York:ACM Press,2002.233 -246.
[5] Hernandez T and Kambhampati S. Integration of biological sources:current systems and challenges ahead [J].ACM SIGMOD Record,2004,33(3):51-60.
[6] Köhler J. Integration of life science databases[J]. Drug Discovery Today,2004,2(2):61-69.
[7] Trissl S,Rother K,Müller H,et al.Columba:an integrated database of proteins,structures,and annotations [J].BMC Bioinformatics,2005,6:81.
[8] Chaurasia G,Malhotra S,Russ J,et al.UniHI 4:new tools for query,analysis and visualization of the human protein-protein interactome[J].Nucleic Acids Res,2009,37(Database issue):D657-660.
[9] Tarcea VG,Weymouth T,Ade A,et al.Michigan molecular interactions r2:from interacting proteins to pathways[J].Nucleic Acids Res,2009,37(Database issue):D642-646.
[10] Muilu J,Peltonen L,Litton JE.The federated database-a basis for biobank-based post-genome studies,integrating phenome and genome data from 600,000 twin pairs in Europe [J].Eur J Hum Genet,2007,15(7):718-723.
[11] Androulakis S,Schmidberger J,Bate MA,et al.Federated repositories of X-ray diffraction images[J].Acta Crystallogr D Biol Crystallogr,2008,D64(Pt 7):810 -814.
[12] Hwang DS,Fotouhi F,Son YJ.A case study:development of an organism-specific protein interaction database and its associated tools [J]. InternationalJournalofCooperative Information Systems,2003,12(2):15.
[13] Marenco L,Wang TY,Shepherd G,et al.QIS:A framework for biomedical database federation[J].J Am Med Inform Assn,2004,11(6):523-34.
[14] Blankenburg H,Finn RD,Prlic'A,et al.DASMI:exchanging,annotating and assessing molecularinteraction data [J].Bioinformatics,2009,25(10):1321-1328.
[15] Dowell RD, Jokerst RM,Day A, et al. The distributed annotation system [J].BMC Bioinformatics,2001,2:7.
[16] Blankenburg H,Ramírez F,Büch J,et al.DASMIweb:online integration,analysis and assessmentofdistributed protein interaction data[J].Nucleic Acids Res,2009,37(Web Server issue):W122-128.
[17] Köhler J,Philippi S,and Lange M.SEMEDA:ontology based semantic integration of biological databases[J].Bioinformatics,2003,19(18):2420-2427.
[18] Alonso-Calvo R,Maojo V,Billhardt H,et al.An agent-and ontology-based system for integrating public gene,protein,and disease databases[J].J Biomed Inform,2007,40(1):17-29.
[19] Noy NF,Shah NH,Whetzel PL,et al.BioPortal:ontologies and integrated data resources at the click of a mouse [J].Nucleic Acids Res,2009,37(Web Server issue):W170-173.
[20] Min H,Manion FJ,Goralczyk E,et al.Integration of prostate cancer clinical data using an ontology[J].J Biomed Inform,2009,42(6):1035-1045.
[21] Crompton S,Matthews B,Gray A,et al.Data integration in bioinformaticsusing OGSA-DAI[EB/OL]. http://www.allhands.org.uk/2005/proceedings/papers/500.pdf,2005/2009-12-18.
[22] Jones AC,White RJ,Gray WA,et al.Building a Biodiversity GRID[A].In Konagaya A and Satou K,eds.:Grid Computing in Life Science[M].Berlin:Springer-Verlag,2005.140-151.
[23] Luo Y,Jiang L,Zhuang TG.A grid-based model for integration of distributed medicaldatabases [J]. JournalofDigital Imaging,2008,22(6):579-588.
[24] Huang Y,Ni T,Zhou L,et al.JXP4BIGI:a generalized,Java XML-based approach for biological information gathering and integration[J].Bioinformatics,2003,19(18):2351-2358.
[25] Bales N,Brinkley J,Lee ES,et al.A framework for XML-based integration ofdata,visualization and analysisin a biomedical domain [A].Database and XML Technologies[M].Berlin Heidelberg:Springer-Verlag,2005.207-221.
[26] Fernandez M,Kadiyska Y,Morishima A,et al.SilkRoute:a framework for publishing relational data in XML [J].ACM Transactions on Database Technology,2002,27(4):438-493.
Progress in Biomedical Heterogeneous Database Integration
ZHANG ZhiZHANG Zheng-Guo*
(Institute of Basic Medical Sciences,Chinese Academy of Medical Sciences,Peking Union Medical College,Beijing 100005,China)
With the rapid development of biomedicine,many biomedical databases have constantly emerged.How to methodically organize these independent databases is fundamental to increase the overall knowledge and understanding of a specific subject.Data integration can implement wider data sharing and more effective utilization of data,and it has become the core research content of bioinformatics.In this paper,the methods of the integration of biomedical heterogeneous database were introduced,and the newest progress in this field was reviewed.The characteristics and the use conditions of all methods were discussed and summarized as well.
heterogeneous database;database integration;data warehouse;federated database;middleware
R318
A
0258-8021(2010)03-0454-10
10.3969/j.issn.0258-8021.2010.03.022
2009-12-25,
2010-01-08
中华医学基金(CMB03-787)
*通讯作者。 E-mail:zhangzg126@126.com
