百合种质资源间亲缘关系及RAPD指纹图谱分析
2010-03-17童巧珍周日宝刘湘丹盛孝邦王朝晖
童巧珍,周日宝,刘湘丹,盛孝邦,王朝晖
(1.湖南农业大学,湖南 长沙 410128;2.湖南中医药大学,湖南 长沙 410208)
百合,作为药用植物、观赏植物和食物在中国的栽培已经有很长一段历史,其种质资源丰富,常见有以下种或品种。
药用百合有百合科植物卷丹 Lilium lancifolium Thunb.、百合L.brownii F.E.Brown var.viridulum Baker、细叶百合L.pumilum DC.的干燥肉质鳞叶[1];观赏百合在商业花卉栽培学上其主要分为以下三大类型:一是东方百合 (Oriental hybrids)种群类型;二是亚洲百合 (Asiatic hybrids)种群类型;三是麝香百合(L.longiflorum)种群类型[2]。至今在切花栽培中仍负盛名的“康涅锹格王子”就是卷丹、毛百合(L.dauricum Ker.Gawl)与荷兰百合(L. enchantment)杂交所获得的中世纪百合杂交种中的优良品种,王百合(L.regale)、麝香百合亦为名贵切花[3];食用百合主要有百合、卷丹、川百合(L.davidii Duch.)、山丹(L.pumilum DC.)、百合、毛百合及沙紫百合(L.sargentiae Wils.)。
中国作为中药资源大国,尽管种质资源丰富,但由于种种原因,一些野生资源遭到严重破坏。尽管自20世纪80年代以来我国对百合属植物的研究己取得了一些初步成果,但从现有资料分析中可看出,有关百合的研究工作主要集中在分类学、繁殖及栽培生物学、细胞学、育种等4个方面,对其种质资源描述评价方面却少有涉及[4-5]。而对百合种质资源及其亲缘关系的研究,可为其栽培生产提供适宜的种类,为其育种提供丰富的种源,从而对百合产业的可持续发展有着重要意义。笔者利用RAPD技术对不同来源的百合种质资源进行了研究,其DNA提取及RAPD条件优化方法均已作了报道[6-7],现将其种质资源间亲缘关系及RAPD指纹图谱分析方法与结果报道如下。
1 材料与方法
1.1 百合种源概况
2005年7~11月从江苏、江西、广西、湖南、甘肃5省(区)采集16个百合种质资源各10~16株(表1),种植于湖南中医药大学药用植物园内,以备采集新鲜鳞叶和其他试验用。每一产地来源的百合样品采集10株地下新鲜肉质鳞叶,分别提取其DNA,然后按照等量方式将来自10株的DNA混合后,作为该地百合DNA的样本,用于RAPD分析。
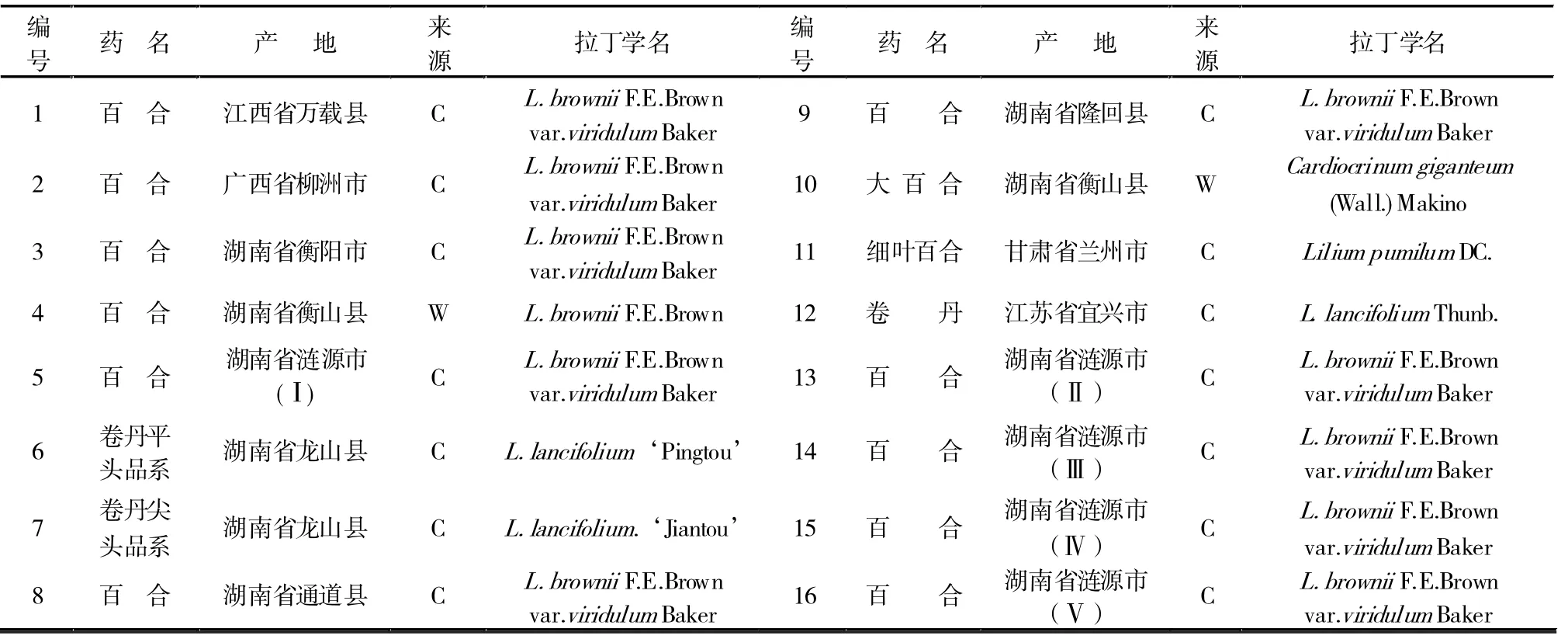
表1 百合样品来源一览表
1.2 DNA提取及RAPD扩增反应
参见文献[6-8]中DNA提取及RAPD扩增反应条件优化的方法。
1.3 数据分析
将电泳图谱上清晰且可重复出现的条带赋值为“1”,同一位置无带的赋值为“0”,由此生成“0”和“1”原始矩阵。统计每个引物扩增处的总带数和多态性带数。数据采用NTSYS软件进行UPGMA(Unweighted Pair Group Method with Arithmetic Mean)聚类分析,建立样品间的亲缘关系聚类树型图。
2 结果与分析
2.1 RAPD随机扩增多态DNA标记引物筛选与PCR扩增结果
本法提取的百合基因组DNA的样品,测得其OD260/ OD280比值范围为1.53~2.00,以1.5%琼脂糖凝胶电泳检测,结果表明,提取的基因组DNA为一条清晰完整明亮的带,迁移率与λDNA相当,没有拖尾或弥散现象(图1)。凝胶的点样槽中未发现明显的荧光信号。因而,该实验提取的DNA纯度高,完全符合实验的要求,可用于进一步进行RAPD扩增。
随机用5号样品对120条随机引物进行筛选,从中选出35条能获得清晰谱带、反应稳定、存在多态性和重复性好的引物用于PCR扩增和群体遗传变异的分析(表2,图1)。
2.2 百合供试材料基因组的指纹图谱分析
利用引物B2、G4、M14、B3对16份百合供试材料进行RAPD扩增产物电泳图分析,得出各样品相对应引物扩增产物的DNA指纹图谱及指纹峰(表3)。结果表明指纹图谱能反应出样品间差异,可为鉴别药材提供分子水平指纹图谱。图2为11号样品的DNA指纹图谱,图3为B2、G4、M14、B3扩增产物电泳图谱的三维图谱。
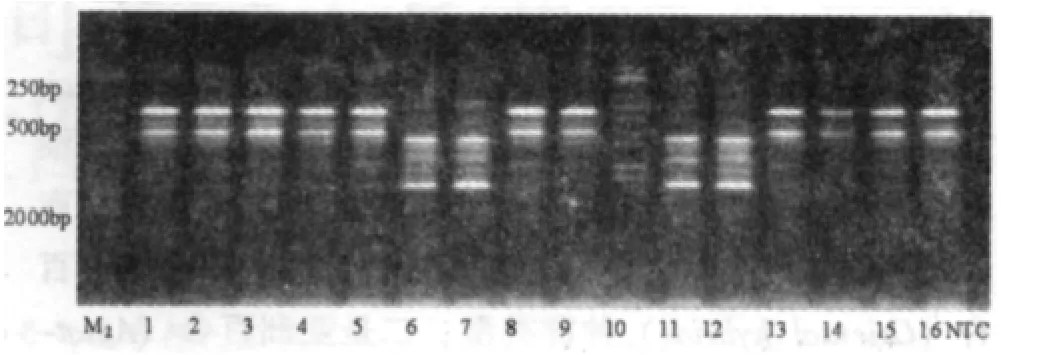
图1 引物M19扩增结果图
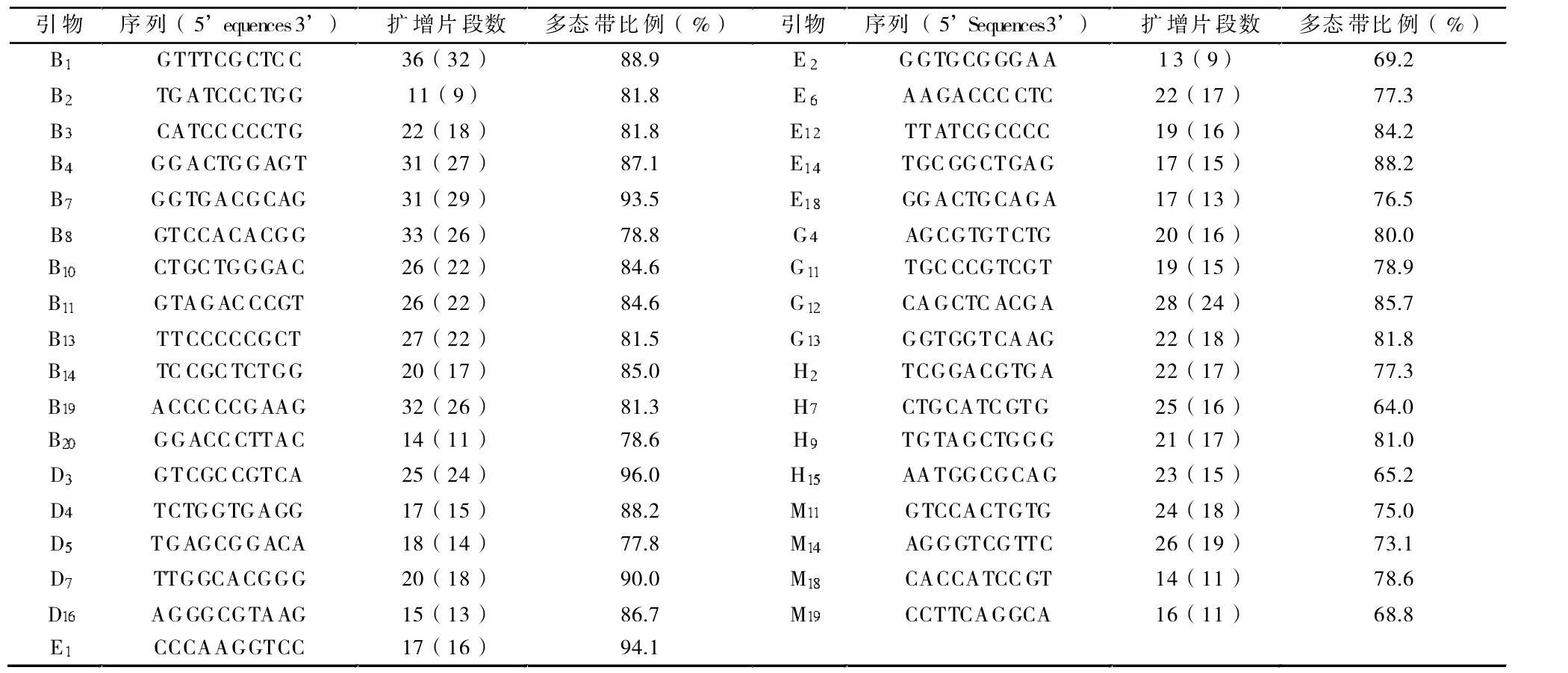
表2 百合种质资源35条有效引物序列及其DNA片段数

表3 引物B2、G4、M14、B3对各样品扩增的指纹峰

图2 引物B2、G4、M14、B3对样品11扩增的指纹图谱

图3 引物B2、G4、M14、B3扩增产物电泳图谱三维图谱
2.3 百合供试材料的亲缘关系及遗传距离分析
RAPD扩增结果表明,35条有效引物共扩增出769条谱带,其中多态带有628条,多态性位点比率为81.7%,每条引物扩增的带数在11~36条之间,平均为22条。其中扩增带数最多的为B1引物,有36条扩增带,且不同的引物扩增片段的数目及其多态性程度不同,表明供试材料具备丰富的遗传多样性。利用统计分析软件将16份不同百合种质资源的RAPD标记进行聚类分析(图4),参照表4列出的不同百合种质资源间的遗传距离可看出,16份样品之间的遗传距离为0~0.940 7。其中,样品6与样品7之间的遗传距离最小达到0.008 9,两者间亲缘关系最近,为卷丹从江苏省引种到湖南省龙山县栽培过程中形成的2个农家品系(尖头系和平头系)。样品10与样品11之间的遗传距离最大为2.209 1,表明这两个样品间有较大的遗传背景差异,它们之间的亲缘关系最远,为百合科两个近缘百合属和大百合属植物。
在聚类分析的基础上,16份供试品以遗传距离0.940 7为阈值可分为2个RAPD群(RAPD groups,简称RGs),即RG1和RG2。其中RG2为样品10,而其他样品为RG1。这为百合科的不同两个属,RG1为百合属 (lilium),RG2为大百合属(cardiocrinum)。
RG1在阈值为0.579 0处又被分为两大亚群:第一大亚群包括样品6、7、11、12,其余样品为第二大亚群。第一大亚群在阈值为0.316 7处又被分为两类:一类为6、7、12号样品卷丹L.lancifolium Thunb.;第二类为11号样品细叶百合L.pumilum DC.。第二大亚群在阈值为0.298 2样品分为两类,第一小类为野生种样品4,除10号样品外,其余为第二小类,均为栽培品种,说明同属同种植物因为生存环境不同,存在着较大的遗传差异。聚类结果与生长环境亦有趋异效应,故第二小类同属同种不同产地的百合存在较小的分类。以上聚类分析结果与传统鉴别的结论一致,说明RAPD分析可以从分子水平上鉴定不同属不同种不同遗传背景的药材,为中药材百合种质资源的研究和利用提供了一种新的方法。
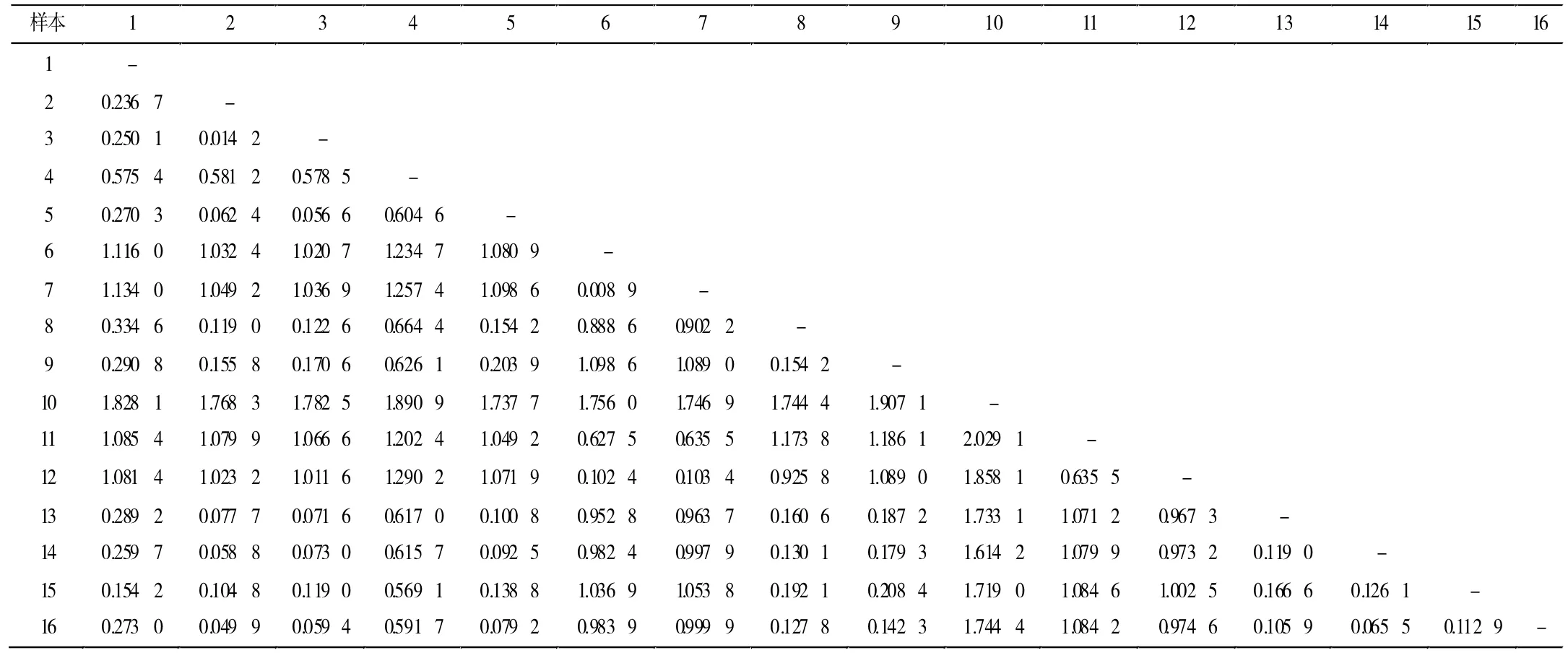
表4 百合样品间遗传距离
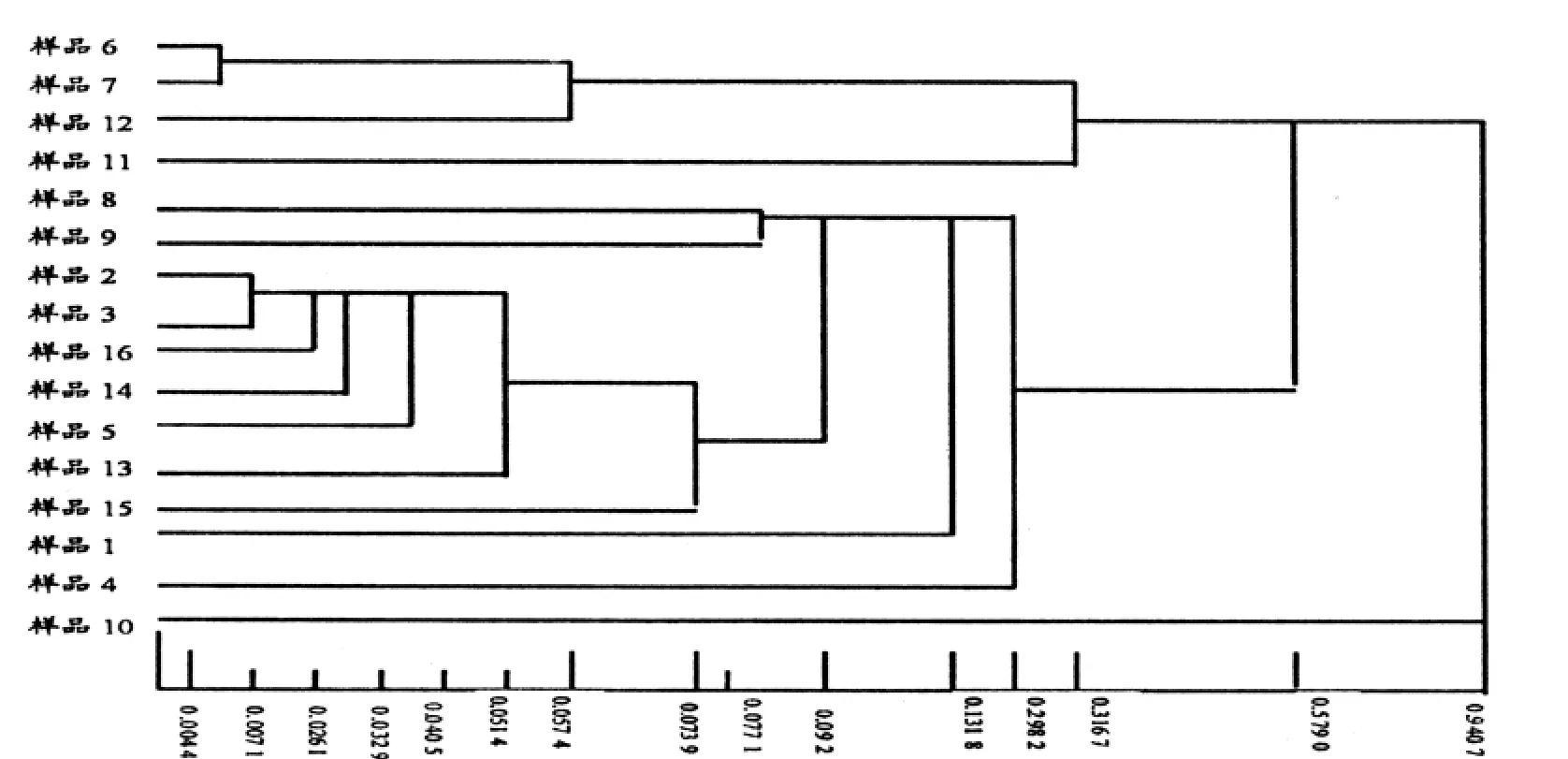
图4 基于RAPD标记结果的实验材料聚类分析图
3 讨论
3.1 指纹图谱及三维图谱分析
应用凝胶成像分析系统对RAPD扩增产物电泳图进行分析可以得出以泳道中各谱带的吸收度值A为纵坐标、谱带位置Pix为横坐标的指纹图谱及三维图谱。从图谱中可以看出不同样品同一引物扩增产物指纹图谱峰有相同位置,也存在不同位置,能体现不同样品之间基因组序列存在的差异,说明不同样品基因组序列中与引物互补的相同序列片断位点不同。不同样品同一谱带位置峰峰高(吸收度值)不同,说明不同样品基因组序列中与引物相互补的相同序列片断数目不同。指纹图谱和电泳图谱都能说明样品之间的遗传背景差异,但指纹图谱将扩增产物电泳条带的有无及条带的强弱转化为指纹峰形式,这可与化学指纹图谱一起建立鉴别中药材优良种质的多元鉴别体系,具有较大的研究意义。
3.2 遗传距离和聚类分析
从分析结果可知不同属百合之间遗传距离较大,亲缘关系较远,同属百合之间遗传距离相对较小,亲缘关系较近,但属内不同种百合之间亦存在一定的遗传距离,证明种内亦存在遗传差异;另同属同种植物之间也存在一定的遗传距离,这说明地域差异也会引起植物产生趋异效应而具遗传差异;野生种与经过驯化的栽培品种在遗传距离也较大,表明野生种与栽培种在遗传背景上存在一定的差异;药材具有明显的地域性,地域上偏北或偏南的同属同种植物之间遗传距离相对较小,而地域偏北与偏南的同属同种植物之间遗传距离相对较大。
通过对16个样品进行ΜPGMA聚类分析得到树状图表明所研究的百合品种分为两大类群,继而又将一些亚群明显区分开来。实验结果与我们经验鉴定的结果一致,说明RAPD技术具有较高的分辨力,完全能够将在形态、生药性状及化学成分等特征上具有高度相似性、来源一致或十分相近的药材区别开来,可正确鉴别百合科不同属及种间群体。而且对种内等级亦能进行有效的区别,为百合药材的鉴定、百合不同品系的鉴定以及筛选优良品系提供分子生物学依据。RAPD分子遗传标记的应用,从居群和分子水平上阐明药材道地性产生的生物学本质成为可能,因而该方法可以作为不同中药百合品系鉴定的依据,为中药百合的鉴定提供了一种行之有效的分子水平方法。
[1]中华人民共和国国家药典委员会.中华人民共和国药典[S].北京:中国医药科技出版社,2005:88.
[2]神农氏.神农本草经[M].北京:人民卫生出版社,1982:67.
[3]苏 敬,李 绩.唐本草[M].合肥:安徽科技出版社,1981:223-224.
[4]卢之颐.本草乘雅半偈[M].北京:人民卫生出版社,1986:279-280.
[5]李时珍.本草纲目[M].北京:人民卫生出版社,1982:1 680-1 682.
[6]童巧珍,周日宝,刘湘丹,等.药用百合鳞叶中总DNA提取方法的研究[J].中国药房,2008,19(6):406-408.
[7]童巧珍,盛孝邦,刘湘丹,等.药用百合DNA提取和RAPD条件的优化[J].湖南中医药大学学报,2008,28(2):33-36.
[8]易庆平,罗正荣,张青林.植物总基因组DNA提取方法综述[J].安徽农业科学,2007,35(25):7 789-7 791.
