双组分系统分析预测共生机制
2010-03-12崔艳华曲晓军
崔艳华,曲晓军,马 莺
(1.哈尔滨工业大学食品科学与工程学院,哈尔滨150090,yhcui@hit.edu.cn; 2.黑龙江省科学院微生物研究所,哈尔滨150010)
德氏乳杆菌保加利亚亚种(Lactobacillus delbrueckii subsp. bulgaricus,文 中 简 称 L. delbrueckii)和 嗜 热 链 球 菌 (Streptococcus thermophilus)是最具经济价值的同型发酵乳酸菌,在世界上广泛应用于酸奶和瑞士型干酪等发酵乳品的生产[1-2].二者在牛奶发酵过程中彼此促进生长和酸化,但互作机制尚不十分明确[1].在牛奶发酵过程中,L.delbrueckii和S.thermophilus需对变化的环境进行感知和反应.在细菌中通常由双组分信号转导系统(Two-component system,TCS)来完成这一功能.典型的TCS包括组氨酸蛋白激酶(Histidine protein kinase,HK)和应答调节蛋白(Response regulator protein,RR).
当前L.delbrueckii和S.thermophilus TCS系统研究鲜有报道,且二者共生机制尚不十分明确[3-5].为此,利用生物信息学手段对二者的TCS系统进行预测,并进行结构和功能分析,旨在从TCS系统的角度揭示二者的协同关系.
1 实验
1.1 L.delbrueckii和S.thermophilus基因组序列
L.delbrueckii ATCC 11842、ATCC BAA-365;S.thermophilus LMG18311、LMD-9和CNRZ1066基因组序列信息来自NCBI(www.ncbi.nih.gov/genomes/Bacteria/).
1.2 双组分信号系统的预测
上述基因组序列用于TCS的预测.保守蛋白序列数据库(Pfam:www.sanger.ac.uk/software/ Pfam/)中的 HisKA(PFAM00512)、HATPase-c (Pfam02518)、Response-reg(PFAM00072)序列用于HMMER搜索.HisKA HMM和HATPase-c HMM用于扫描组氨酸蛋白激酶中高度保守的磷酸基团接受区域和HATPase区域,Response-reg HMM用于扫描应答调节蛋白中保守的磷酸基团接受区域.
1.3 序列分析
功能域采用SMART(simple modular architecture research tool)分析(http://smart.embl-heidelberg.de).应用TMHMM Server(http://www.cbs. dtu.dk/services/TMHMM-2.0)分析蛋白跨膜结构区域.序列比对采用BLASTP程序(www.ncbi. nlm.nih.gov/blast/).采用ClustalW进行多重序列比对,利用TREECON软件建立系统发育树.
2 结果与分析
2.1 双组分信号转导系统的分布
使用Pfam HMMs HisKA、HATPase-c和Response-reg信息对 L.delbrueckii ATCC 11842、ATCC BAA-365及S.thermophilus CNRZ1066、LMG18311和LMD-9等5株菌全基因组的TCS系统进行预测分析.L.delbrueckii中,HK为6~7个,RR为6~7个;而S.thermophilus中发现了6~10个HK和8~10个RR(见表1).

表1 L.delbrueckii和S.thermophilus中TCS的分布情况
L.delbrueckii ATCC11842与BAA-365菌株,具有5个高度同源的TCS.ATCC11842中含有单独的HK(YP-618959)和单独的RR(YP-618256),但却未发现二者相应的 RR和 HK. ATCC11842和BAA-365菌株来源上存在差异,推测调控基因在进化上存在一定差异[3-4].
S.thermophilus CNRZ1066与LMG18311菌株的TCS相似,而LMD-9菌株的TCS数目明显少于上述两个菌株.CNRZ1066分离自法国酸奶,LMG18311于1974年分离自英国的商业酸奶,LMD-9也分离自酸奶[4-5].通过比较基因组杂交分析发现,S.thermophilus不同菌株的核心基因具有较大差异,推测部分基因在稳定的环境中退化和消失[6-7].
将预测的HK进行聚类分析,建立系统发育树,以大肠杆菌的HK PhoR作参比,以更好显示L.delbrueckii与S.thermophilus各菌株HK之间的亲缘关系.研究表明HK分为11组(I-XI).使用TMHMM、Pfam、SMART工具对各组HK蛋白结构区域进行了分析.I-XI组呈现了不同的结构区域特征(见图1,2).
所预测的HK大多数是跨膜蛋白,可直接感受外界环境的变化,而部分HK,如IV、X、XI组的HK缺乏跨膜区域.缺少跨膜区域的HK,可能感受细胞内信号(如氧化还原电位的变化)或者通过特有的信号传导蛋白获得外界环境信号. HAMP结构域主要发现于HK、热激蛋白HSP90等ATP结合蛋白中,但具体功能未知,推测与细菌渗透敏感性有关[8].III、IV、VI、XI组的蛋白均具有HAMP结构区域.PAS结构域(PER-ARNTSIM domain)能够感受光、氧、氧化还原电位、小分子配体和细胞总能量的变化,是重要的信号感受结构域[9].PAC结构区域通常出现在PAS结构域的C端,推测与PAS结构域的折叠有关.VI、VII组中的蛋白均具有PAS结构区域.VI组中蛋白PAS结构域的C端还伴有PAC结构域.
将预测的RR进行聚类分析,建立系统发育树,以大肠杆菌的应答调节蛋白KdpE作为参比.研究表明,RR分为11组(I-XI),与HK聚类结果相类似.使用TMHMM、Pfam、SMART工具对各组的RR蛋白结构区域进行分析(见图3,4).与HK多样的结构区域相比,RR的结构区域较单一,大多数RR的输出区域为典型的Trans reg-C结构域(III组,V组至XI组).
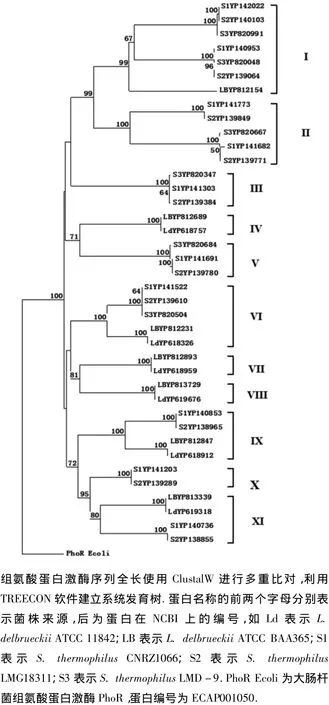
图1 L.delbrueckii与S.thermophilus组氨酸蛋白激酶的系统发育图谱
来自 L.delbrueckii的 YP-812153和 YP-618256及 S.thermophilus的 YP-142021、YP-140102、YP-820990、YP-820049、YP-140954、YP-139065聚为一类,上述蛋白含有LytTR型输出区域.LytTR结构域中的DNA结合区域是一种新型的DNA结合区域,不同于常见的HTH(helix -turn-helix)或WH(winged-helix)型的DNA结合区域[10].LytTR型输出区域广泛地分布于低G+C含量的革兰氏阳性菌中,参与毒力、细菌素和胞外多糖合成等多种代谢调控[10].
来自 S.thermophilus的 YP-820749、YP-141772、YP-139848、YP-820666、YP-141681、YP-139770的输出区域为HTH-LuxR结构区域. HTH-LuxR结构域由65个氨基酸组成,具有螺旋-转角 -螺旋(helix-turn-helix,HTH)结构,为DNA结合区域,常见于LuxR/FixJ族应答调节蛋白的输出区域.大多数HTH-LuxR结构区域充当转录的激活因子,但有时抑制转录或呈现双重作用[11].L.delbrueckii中RR未发现此类结构域.
所预测的RR大多数具有信号输出区域,而S.thermophilus的YP-141734和YP-139811较为特殊,仅具有接受区域,缺乏DNA结合区域即信号输出区域.通常输出区域是DNA结合模件,充当转录因子,激活目标基因的表达[12].无输出区域的RR,可通过其他方式作用于目标蛋白,如E.coli调控趋化作用的CheB磷酸化后改变对靶蛋白的亲和力,促使化学感应器蛋白MCPs去甲基化[13].有一部分此类RR,仅作为信号级联放大系统中的一个上游组分,在复杂的磷酸转移系统中传递磷酸基团,如B.subtilis孢子形成系统中的Spo0F即充当一个磷酸基团传递的元件[14].
2.2 组氨酸蛋白激酶和应答调节蛋白的分类
典型的HK具有传感器区域和组氨酸激酶核心区域.激酶核心区域序列高度保守,存在着5个由5~10个氨基酸组成的保守区域,分别为H、N、G1、F、G2-box.这些高度保守的氨基酸残基在底物结合、催化上具有重要作用[12,15],根据保守区域特点,Grebe将HK划分为11亚族[15].
以已知类型的HK为参照,将预测HK的保守HisKA和HATPase-c区域序列进行聚类分析.结果发现53.8%的HK属于HK1a亚族(见表2).HK1a亚族是HK较大的一个亚族,参与多种代谢调控,如磷酸盐代谢、藻酸盐的生物合成等. YP-141691和YP-139780属于HK3i亚族,其保守的组氨酸残基(His,H)上游第二位为色氨酸(Trp,W)残基.YP-812154等10个蛋白属于HK10亚族,此类HK多参与细菌的群体感应[16].
RR位于细胞质中,由N端的接受区域和C端的输出区域组成.通常输出区域是DNA结合模件,充当转录因子,具有典型的HTH结构.根据输出区域的差异,可将RR分为CheB、CitB、LytR和OmpR等11个亚族[15].聚类分析表明,大多数RR属于OmpR亚族(见表2).YP-618256、YP-812153等蛋白具有LytTR结构区域,属于LytR亚族.YP-141681、YP-141772等蛋白具有HTHLuxR结构区域,属于CitB亚族.

图2 L.delbrueckii与S.thermophilus组氨酸蛋白激酶的结构域简图

图3 L.delbrueckii与S.thermophilus应答调节蛋白的系统发育图谱

图4 L.delbrueckii与S.thermophilus应答调节蛋白的结构域简图
2.3 功能的预测
使用NCBI BLASTP对预测的TCS系统进行了功能预测,其中有部分TCS与已知生物功能的TCS系统相似(见表2),功能涉及全局调控、细菌素合成等.尽管S.thermophilus在生境上与乳杆菌、乳球菌相近,但在遗传进化上却与病原链球菌如 Streptococcus pyogenes、S. pneumoniae、Streptococcus mutans关系密切.BLAST研究发现,S.thermophilus中多个TCS系统与病原菌中毒力因子调控系统高度同源(见表 2).S. thermophilus与进化关系密切的病原菌的全基因组比较研究发现,病原菌中的致病力相关基因在该菌中已缺失或为假基因[5],因此上述毒力调控相关的TCS无实际功能.

表2 L.delbrueckii和S.thermophilus TCS系统的功能预测
L.delbrueckii中发现了与 Lactobacillus acidophilus中调控蛋白水解能力的LBA1524HK/ LBA1525HK系统同源的YP-619318/YP-619319和YP-813339/YP-813340,推测这两个系统与细菌的蛋白水解能力相关[17].而在S.thermophilus中没有发现此类TCS.该结果从TCS调控的角度,支持了Gilbert等人观点,即L.delbrueckii具有较高蛋白水解能力,为S.thermophilus提供肽和氨基酸营养[18].同时LBA1524HK/LBA1525HK也与细菌的耐酸能力密切相关,因此推测 YP-619318/YP-619319和 YP-813339/YP-813340调控着细菌的耐酸能力.在牛奶发酵过程中,细菌需耐受不断酸化的低pH环境.耐酸能力是评价酸奶发酵剂的一个重要指标.同时研究发现L. delbrueckii的 YP-618912/YP-618911、YP-812847/YP-812846系统与L.acidophilus中调控细菌耐胆汁能力的YP-194286/YP-194287同源性很高[19],推测与耐胆汁能力相关.益生菌在胃肠道中生存和定殖的最大挑战是酸性环境(pH 2.5~3.5)和胆汁对其生长的抑制作用.因此调控耐酸和耐胆汁能力的TCS系统将有助于益生菌在人胃肠道中生存和其益生功能的发挥.
有些细菌在生长过程通过分泌细菌素抑制其他微生物的生长.目前,已在S.thermophilus菌株中发现了包括thermophilin ST-1、T、110等在内的8种细菌素[20].而L.delbrueckii细菌素报道较少[21].BLAST发现,S.thermophilus CNRZ1066、LMG18311和LMD-9 3个菌株均含有两个TCS参与细菌素合成(见表 2).YP-141303/YP-141302、YP-139384/YP-139383、YP-820347/ YP-820346分别来自 3个菌株且同源,与Bacillus subtilis、L. lactis 和 Carnobacterium divergens中 SpaK/SpaR、NisK/NisR 和 DvnK/ DvnR系统高度同源,这些系统分别调控着I型细菌素 subtilin、nisin、divercin V41的合成[21-24]. YP-142022/YP-142021、YP-140103/YP-140102和YP-820991/YP-820990系统与 Streptococcus pneumoniae中调节细菌素合成的BlpH/BlpR系统相似[24].近来研究证实S.thermophilus LMD-9中YP-820991/YP-820990系统调控细菌素的合成,而CNRZ1066、LMG18311中的同源系统,由于细菌素ABC转运基因blpA和转运加工基因blpB基因发生无义突变而没有相应的功能[25-26].在 L.delbrueckii ATCC 11842、BAA-365基因组中没有发现调控细菌素合成的基因.因此,推测S.thermophilus通过分泌细菌素抑制其他细菌的生长,为自身和L.delbrueckii的生长创造有利条件.同时,S.thermophilus产生的细菌素也可能对L.delbrueckii产生拮抗作用.
S.thermophilus中发现了与S.pneumoniae调控细胞感受态建立的CiaH-CiaR同源的TCS系统 YP-141203/YP-141200和 YP-139289/ YP-139286[27].建立细胞感受状态是自然遗传转化的必要条件.而自然遗传转化是实现细菌种间基因交流即基因水平转移的重要机制.全基因组分析表明S.thermophilus存在着10余处基因水平转移区域,涉及细菌素、胞外多糖合成等等[5].因此,推测 YP-141203/YP-141200和 YP-139289/YP-139286与细胞感受态建立调控相关,进而促进种间基因交流.而L.delbrueckii中未发现此类系统.
Bolotin等人研究发现,S.thermophilus和L. delbrueckii之间存在基因水平转移现象.S. thermophilus pepD基因中含有一个发生基因水平转移的G+C含量异常区域.该区域内的3.6 kb大小片段,与L.delbrueckii的DNA区域同源性高达95%,编码了独特的metC基因,该基因为甲硫氨酸合成所必须,而牛奶中缺乏此种氨基酸[5].因此推测S.thermophilus通过YP-141203/YP-141200和YP-139289/YP-139286调控建立细胞感受态,进而获得L.delbrueckii提供的相关基因,实现种间基因的转移,从而实现与 L. delbrueckii subsp.bulgaricus共生,适应生境的目的.同时,本研究通过YP-141203/YP-141200和YP-139289/YP-139286系统的上下游基因扫描发现,这两个TCS邻近区域存在着IS3型转座酶基因子.推测通过转座酶基因实现外源基因在基因组的定位.
3 结语
通过生物信息学分析方法对L.delbrueckii和S.thermophilus的TCS系统进行预测和结构分析,并对其功能进行了预测.L.delbrueckii和S. thermophilus因长期适应营养丰富的牛奶环境而缺少一些代谢系统,因此TCS数目较少,在5~9个之间.这些TCS系统与全局调控、抗生素抗性及外界压力相关,为进一步通过实验确定其功能奠定了基础.其中部分TCS与细菌素合成、蛋白水解能力和细菌感受态的建立相关,推测德氏乳杆菌保加利亚亚种和嗜热链球菌通过以上双组分系统相互协作,进行共生.本研究从TCS调控信号转导的角度,分析了 L.delbrueckii和 S. thermophilus的共生关系,为明确二者共生机制提供了旁证.
[1]ADOLFSSON O,MEYDANI S N,RUSSELL R M.Yogurt and gut function[J].Am J Clin Nutr,2004,80: 245-256.
[2]GARCÍA-ALBIACH R,JOSÉ M,DE FELIPE P,et al. Molecular analysis of yogurt containing Lactobacillus delbrueckii subsp. bulgaricus and Streptococcus thermophilus in human intestinal microbiota[J].Am J Clin Nutr,2008,87(1):91-96.
[3]VAN DE GUCHTE M,PENAUD S,GRIMALDI C,et al.The complete genome sequence of Lactobacillus bulgaricus reveals extensive and ongoingreductive evolution[J].Proc Natl Acad Sci USA,2006,103(24): 9274-9279.
[4]MAKAROVAA K,SLESAREVB A,WOLFA Y,et al. Comparative genomics of the lactic acid bacteria[J]. Proc Natl Acad Sci USA,2006,103(42):15611-15616.
[5]BOLOTIN A,QUINQUIS B,RENAULT P,et al.Complete sequence and comparative genome analysis of the dairy bacterium Streptococcus thermophilus[J].Nat Biotechnol,2004,22(12):1554-1558.
[6]GALIA W,PERRIN C,GENAY M.Variability and molecular typing of Streptococcus thermophilus strains displaying different proteolytic and acidifying properties[J].Int Dairy J,2009,19:89-95.
[7]RASMUSSEN T B,DANIELSEN M,VALINA O,et al. Streptococcus thermophilus core genome:Comparative genome hybridization study of 47 strains[J].Appl Environ Microbiol,2008,74(15):4703-4710.
[8]ARAVIND L,PONTING C P.The cytoplasmic helical linker domain of receptor histidine kinase and methyl-accepting proteins is common to many prokaryotic signalling proteins[J].FEMS Microbiol Lett,1999,176: 111-116.
[9]TAYLOR B L,ZHULIN I B.PAS domains:Internal sensors of oxygen,redox potential,and light[J].Microbiol Mol Biol Rev,1999,63:479-506.
[10]NIKOLSKAYA A N,GALPERIN M Y.A novel type of conserved DNA-binding domain in the transcriptional regulators of the AlgR/AgrA/LytR family[J].Nucleic Acids Res,2002,30:2453-2459.
[11]FUQUA W C,WINANS S C,GREENBERG E P.Quorum sensing in bacteria:The LuxR-LuxI family of cell density-responsive transcriptional regulators[J].J Bacteriol,1994,176:269-275.
[12]PARKINSON J S,KOFOID E C.Communication modules in bacterial signaling proteins[J].Annu Rev Genet,1992,26:71-112.
[13]MOWBRAY S L,SANDGREN M O J.Chemotaxis receptors:A progress report on structure and function[J].J Struct Biol,1998,124:257-275.
[14]TZENG Y L,HOCH J A.Molecular recognition in signal transduction:The interaction surfaces of the Spo0F response regulator with its cognate phosphorelay proteins revealed by alanine scanning mutagenesis[J].J Mol Biol,1997,272:200-212.
[15]GREBE T W,STOCK J B.The histidine protein kinase superfamily[J].Adv Microb Physiol,1999,41:139-227.
[16]STURME M H J,FRANCKE C,SIEZEN R J,et al. Making sense of quorum sensing in lactobacilli:A special focus on Lactobacillus plantarum WCFS1[J].Microbiology,2007,153:3939-3947.
[17]AZCARATE-PERIL M A,MCAULIFFE O,ALTERMANN E,et al.Microarray analysis of a two-component regulatory system involved in acid resistance and proteolytic activity in Lactobacillus acidophilus[J].Appl Environ Microbiol,2005,71(10):5794-5804.
[18]GILBERT C,ATLAN D,BLANC B,et al.A new cell surface proteinase:Sequencing and analysis of the prtB gene from Lactobacillus delbruekii subsp.bulgaricus[J].J Bacteriol,1996,178:3059-3065.
[19]PFEILER E A,AZCARATE-PERIL M A,KLAENHAMMER T R.Characterization of a novel bile-inducible operon encoding a two-component regulatory system in Lactobacillus acidophilus[J].J Bacteriol,2007,189(13):4624-4634.
[20]GILBRETH S E,SOMKUTI G A.Thermophilin 110:A bacteriocin of Streptococcus thermophilus ST110[J]. Curr Microbiol,2005,51:175-182.
[21]MEER van der J R,POLMAN J,BEERTHUYZEN M M,et al.Charaterization of the Lactococcus lactis nisin A operon genes nisP,encoding a subtilisin-like serine protease involved in precursor processing,and nisR,encoding a regulatory protein involved in nisin biosynthesis[J].J Bacteriol,1993,175:2578-2588.
[22]CHUNG Y J,HANSEN N J.Determination of the sequence of spaE and identification of a promoter in the subtilin(spa)operon of Bacillus subtilis[J].J Bacteriol,1992,174:6699-6702.
[23]METIVIER A,PILET MF,DOUSSET X,et al.Divercin V41,a new bacteriocin with two disulphide bonds produced by Carnobacterium divergens V41:Primary structure and genomic organization[J].Microbiology,1998,144:2837-2844.
[24]SAIZIEU de A,GARDES C,FLINT N,et al.Microarray-based identification of a novel Streptococcus pneumoniae regulon controlled by an autoinduced peptide[J].J Bacteriol,2000,182:4696-4703.
[25]FONTAINE L,BOUTRY C,GUÉDON E,et al.Quorum-sensing regulation of the production of Blp bacteriocins in Streptococcus thermophilus[J].J Bacteriol,2007,189(20):7195-7205.
[26]FONTAINE L,HOLS P.The inhibitory spectrum of thermophilin 9 from Streptococcus thermophilus LMD-9 depends on the production of multiple peptides and the activity of BlpGSt,a thiol-disulfide oxidase[J].Appl Environ Microbiol,2008,74(4):1102-1110.
[27]GIAMMARINARO P,SICARD A M,GASC A M.Genetic and physiological studies of the CiaH-CiaR twocomponent signal-transducing system involved in cefotaxime resistance and competence of Streptococcus pneumoniae[J].Microbiology,1999,145:1859-1869.
