分离自扎龙湿地的一株降解纤维素细菌的鉴定
2010-03-07许修宏李洪涛
徐 莹,许修宏,李洪涛
(东北农业大学资源与环境学院,哈尔滨 150030)
扎龙湿地位于黑龙江省西部松嫩平原,是乌裕尔河尾闾湖退化而成的苇草湖沼,总面积2100 km2,其中湿地(湖泊、沼泽)面积1240 km2,属中温带大陆性季风气候,年平均气温2.0~4.2℃,是我国珍稀鸟类和湿地生态类型的国家级自然保护区[1]。其得天独厚的自然条件,孕育了丰富的植被、吸引了各种珍贵鸟类和野生动物前来栖息,我国对扎龙湿地的动植物有了全面的研究,但是目前对该地的微生物资源报道还属鲜见。
扎龙湿地植被分为草甸草原植被、草甸植被、沼泽植被、水生植被4种类型,其中高等植物有525种,隶属70科,以芦苇、羊草为优势种[2],含有大量的纤维素类物质,因此在扎龙湿地取样筛选降解纤维素的微生物,具有可行性。本研究对从扎龙湿地底泥分离出来的一株可以在4℃生长的具有纤维素降解功能的菌株进行了形态学观察、生理生化反应和16S rDNA序列分析鉴定,从而为扎龙湿地微生物资源的开发提供基础理论依据。
1 材料与方法
1.1 材料
1.1.1 供试菌株
菌株ZL-5分离自黑龙江省扎龙自然保护区湿地底泥,现于东北农业大学资源与环境学院黑土实验室保存。
1.1.2 培养基
菌株培养采用牛肉膏蛋白胨培养基,鉴定采用纤维素刚果红培养基。
1.1.3 主要试剂
rTaq酶、dNTPs、pMD-18T载体均购于TAKARA公司;感受态细胞(Escherichia coli TOP 10)购于北京全式金生物工程有限公司;引物合成由上海生物工程公司完成;其余药品、试剂均为国外或国内生产的分析纯产品。
1.2 方法
1.2.1 菌株的活化与生理生化特征鉴定
将-20℃保存的甘油菌悬液接种于液体牛肉膏蛋白胨培养基,于30℃、160 r·min-1培养24 h,每隔2 h取菌悬液,测定菌株生长曲线,选取对数级菌悬液稀释涂布于固体牛肉膏蛋白胨培养基平板,镜检无杂菌后,挑取单菌落接种于纤维素刚果红培养基,30℃培养3 d,每隔12 h观察水解圈大小;菌株的形态观察及生理生化特征鉴定参照文献[3]。
1.2.2 菌株基因组DNA的提取
取培养至饱和状态的菌液1.5mL,6000 r·min-1离心5 min,沉淀物加入467μL的TE缓冲液重悬,再加入30μL 10%的SDS和3μL的蛋白酶K(20 mg·mL-1),混匀,于37 ℃温育1 h;加入100μL NaCl(5 mol·L-1),充分混匀,再加入80μL CTAB/NaCl(10%CATB/0.7 mol·mL-1NaCl)溶液,混匀,于65℃温育10 min;加入等体积的氯仿/异戊醇(24∶ 1),混匀,12000 r·min-1离心 5 min;取上清液加入等体积的酚/氯仿/异戊醇(25∶ 24∶ 1),混匀,12000 r·min-1离心5 min;取上清加入0.6倍体积的异丙醇,轻轻混合至DNA沉淀,12000 r·min-1离心10 min;将DNA沉淀用预冷的70%乙醇洗涤,晾干后TE溶解。
1.2.3 16S rDNA的基因扩增与克隆测序
以基因组DNA为模板,采用细菌通用引物27F/1492R[4]进行PCR扩增。反应体系为:2.5μL 10×PCR Buffer、2.5μL dNTPs、15μL引物27f(10 pmol·L-1)、15μL 引 物 1492r(10 pmol·L-1)、1μL rTaq DNA polymerase、1μL 模板 DNA,加无菌水补足至25μL扩增。反应条件:94℃5 min;94℃40 s,52℃45 s,72℃ 90 s,共30个循环;72℃10 min。
扩增的PCR产物经凝胶回收试剂盒纯化后,与pMD-18T载体连接并转化克隆到E.coil TOP10中,将确定转化成功的新鲜菌液送至南京金思特科技有限公司测序。
1.2.4 构建系统发育树
登录NCBI网站,使用BLAST将测得序列与GenBank中已有的核酸序列进行比较,选取部分相似性较高且已注明种类的16S rDNA序列,应用ClustalX 1.83软件进行多序列比对,利用MEGA 4.0软件,采用NJ法构建该微生物的系统发育树。
2 结果与分析
2.1 纤维素刚果红培养基定性
菌株ZL-5在纤维素刚果红培养基上培养72 h后,水解圈明显(见图1),测定水解圈直径为2.8 cm,初步判定该菌株具有较好的纤维素分解能力。
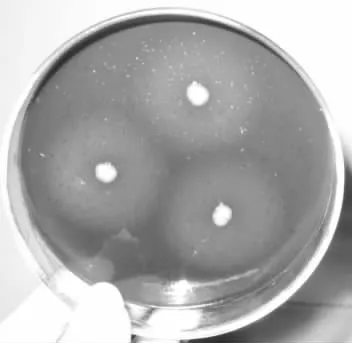
图1 在纤维素刚果红培养基上产生的水解圈Fig.1 Hydrolyzed circle in cellulose congo red culture medium
2.2 形态观察、生理生化鉴定
菌株ZL-5在牛肉膏蛋白胨培养基上形成的菌落为乳白色,呈规则圆形,凹状,光滑,干燥,不易挑取。光学显微镜下观察为两端钝圆的短杆菌,大小约(0.3~1.0)μm×(1.0~3.5)μm,无芽孢,革兰氏染色反应为阴性,且无运动性(见图2)。
菌株ZL-5葡萄糖发酵试验阳性,氧化酶检测阳性,0 mol·L-1NaCl能够生长,氧化酶检测为阳性,且能够利用甘露醇、麦芽糖、蔗糖、半乳糖,淀粉水解、明胶水解阳性,根据文献[3]可以初步判断供试菌株为气单胞菌属(Aeromonas)。
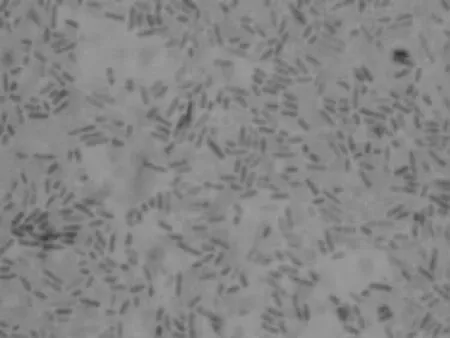
图2 菌株的光学显微镜照片(1000×)Fig.2 Morphology of ZL-5 by optical microscope(1000×)
2.3 生长条件
菌株ZL-5的适宜生长温度范围为17~32℃,最适生长温度为30℃,但在4℃环境下能够缓慢生长;最佳生长pH范围为6.0~10.0,最适生长pH为7.0,当pH低于6.0、高于10.0时,无明显生长。
2.4 PCR反应扩增结果
将提取的基因组DNA经电泳检验后作为模板进行PCR扩增,结果见图3。由图3可见,PCR反应得到的扩增产物片断大小为1.5 kb左右,与16S rDNA片断大小接近,说明扩增的片断是16S rDNA片断。
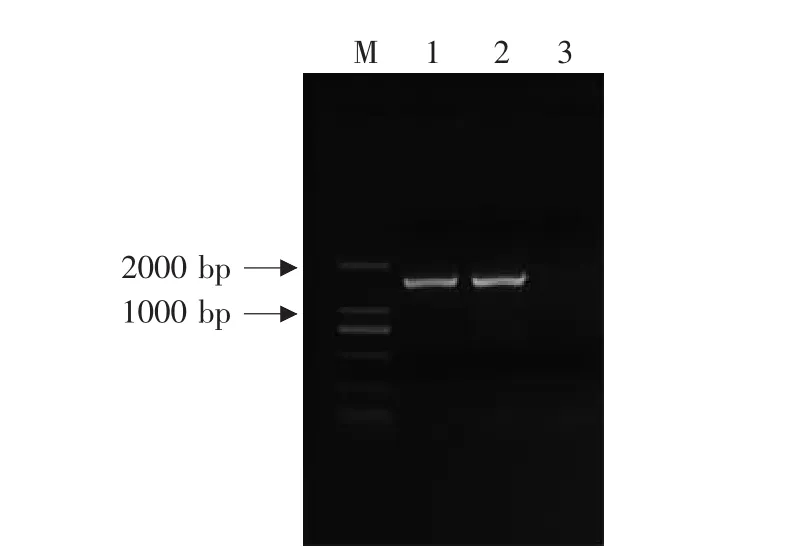
图3 菌株ZL-516S rDNA序列的PCR扩增结果Fig.3 16S rDNA-sequence PCR of ZL-5
2.5 16S rDNA测序结果
将转化成功的菌液送至南京金思特科技有限公司测序,测得16S rDNA大部分序列共1467bp,(见图4)。

图4 菌株ZL-516S rDNA碱基序列Fig.4 16S rDNA base sequence of ZL-5
2.6 系统发育树的构建
选取GenBank中与菌株ZL-5相似性99%以上的18个不同种属的16S rDNA序列,利用MEGA 4.0,采用NJ法构建菌株ZL-5的系统发育树,如图5所示。可以看出,菌株ZL-5与Aeromonas salmonicida subsp.Salmonicida的亲缘关系最近,因此可以鉴定菌株为Aeromonas salmonicida sp.Salmonicida。
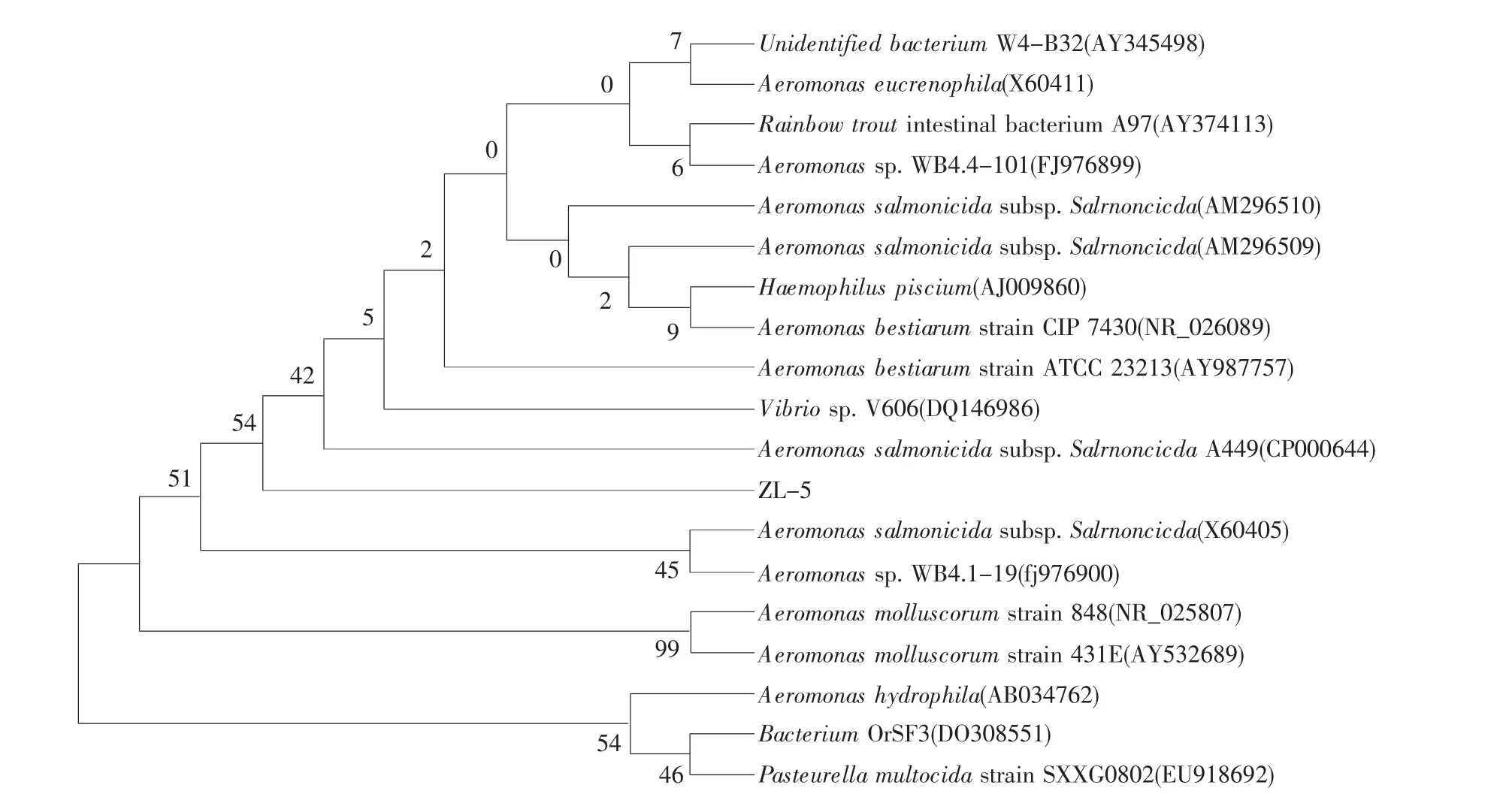
图5 菌株ZL-516S rDNA全序列的系统发育树Fig.5 16S rDNA-sequence phylogenetic tree of ZL-5
3 讨论
16S rRNA普遍存在于一切细胞中,且含量较高,其信息量大、相对分子量适中、易于分析,成为细菌分类系统研究中的理想研究材料。因其在结构与功能上的高度保守性,所以素有“细菌活化石”之称[5]。目前,以16S rRNA/DNA为基础的分子生物学技术已经成为普遍接受的方法[6],传统的微生物分类鉴定仅局限于形态结构观察、生理生化反应等,且试验周期较长,而16S rRNA/DNA分子生物技术主要利用不同微生物在16S核糖体RNA(rRNA)基因序列上的差异来进行微生物种类的鉴定和定量分析[7],在分析、鉴定细菌进化过程及亲缘性方面具有突出特点,其最大的优势是检测速度快,这是常规的细菌鉴定和培养方法无法比拟的[8]。
本研究采用细菌通用引物进行PCR扩增16S rDNA全序列,与GenBank中相似性较高的18个已知菌株进行序列比对后,采用NJ法进行系统发育分析,表明菌株与Aeromonas salmonicida sp.Salmonicida亲缘关系最近。目前,对于气单胞菌属(Aeromonas)降解纤维素功能的研究还属鲜见,韩韫等从汕尾一鲍鱼场采集的样品中分离到120株菌,经过刚果红染色,初筛得到产纤维素酶酶活较高的8株菌,并进行了纤维素酶和滤纸分解的研究,其中两株在分泌纤维素酶活力及降解滤纸能力方面均很强,经鉴定分别为Aeromonas sobria与Vibrio alginolyticus,可用于海洋菌株产纤维素酶的进一步研究[9]。石鹏君从饲喂黑麦草的草鱼肠道中分离出多株产纤维素酶的细菌,其中气单胞菌属、芽孢杆菌属和肠杆菌属是已报道的草鱼肠道主要的优势菌,经16S rDNA序列相似性分析,该气单胞菌株与Aeromonas veronii bv.Sobria strain RK 77443(AY987763)相似性为99%[10]。目前研究尚未报道Aeromonas salmonicida sp.Salmonicida的降解纤维素功能,本研究筛选出来的这株可以在4℃生长的降解纤维素细菌恰恰填补了这个空白,但其功能的后续研究还有待进一步深入,如能利用其在低温环境下能够生长的特性来进行寒地纤维素降解试验的研究,那么其应用前景也将是广阔的。
4 结论
利用16S rDNA序列和传统生理生化指标鉴定菌株ZL-5,初步鉴定该菌株为Aeromonas salmonicida sp.Salmonicida。
[1]殷志强,秦小光,刘嘉麒,等.扎龙湿地的形成背景及其生态环境意义[J].地理科学进展,2006,25(3):32-38.
[2]盖赫莉,王淑梅,王欣.对扎龙湿地生态环境的分析[J].黑龙江环境通报,2002,26(3):94-96.
[3]东秀珠,蔡妙英.常见细菌系统鉴定手册[M].北京:科学出版社,2001.
[4]martin-Laurent F,Philippot L,Hallet S,et al.DNA extraction from soils:Old bias for new microbial diversity analysis methods[J].Applied and Environmental Microbiology,2001,67(5):2354-2359.
[5]刘毅,韩金祥.16S rRNA基因在脑脊液细菌鉴定中的应用[J].临床检验杂志,2002(4):245-246.
[6]Zhang T,Fang H H P.Digitization of DGGE(denaturing gradient gel electrophoresis)profile and cluster analysis of microbial communities[J].Biotechnol Lett,2000,22:399-405.
[7]张彤,方汉平.微生物分子生态技术:16S rRNA/DNA方法[J].微生物学通报,2003,30(2):97-101.
[8]杨霞,陈陆,王川庆.16S rRNA基因序列分析技术在细菌分类中应用的研究进展[J].西北农林科技大学学报:自然科学版,2008,36(2):55-60.
[9]韩韫,蔡俊鹏.产纤维素酶海洋菌株的筛选及鉴定[J].现代食品科技,2005,21(3):36-44.
[10]石鹏君.山羊瘤胃和草鱼肠道微生物分子生态研究及β-葡萄糖苷酶基因的克隆[D].北京:中国农业科学院,2007.
