10个绵羊种群的微卫星变异和群体遗传结构分析
2010-01-30乔利英刘文忠周忠孝
乔利英,刘文忠,周忠孝
(山西农业大学动物科技学院,太谷 030801)
微卫星被用于包括人在内的各种动物基因作图的首选标记。在畜禽品种资源分类、遗传多样性评估、保种和品种的培育等方面,也是重要的标记之一。利用微卫星制作DNA指纹图,通过分析进行个体、品种(系)鉴定是微卫星的最早应用之一[1]。同时,在定位功能基因、个体与亲缘关系的鉴定、数量性状基因座(quantitative trait loci,QTL)定位及连锁分析、标记辅助选择等方面起着重要作用,尤其在群体遗传结构和遗传变异、构建系统发生树等方面有广泛应用。利用微卫星标记已对某些绵羊品种间的进化关系和遗传距离,品种内的遗传变异等进行了研究。所涉及品种对于家用绵羊可以是起源于某一品种的具有较近亲缘关系的品种[2],或者对于野生原始绵羊可以是同一物种的若干地理亚种[3],但绝大多数研究则集中在纯种绵羊品种上。
本研究拟以3个引入品种、1个本地品种和用于山西肉用绵羊母本品系培育的这些品种间的6个杂交组合为对象,研究微卫星变异和群体遗传结构,旨在为杂交育种和新品系选育提供科学依据。
1 材料与方法
1.1 材料 本研究所用的219个个体血液样品均采自山西省沁水示范牧场,采集的10个品种(系)包括南非肉用美利奴羊(43只)、考力代羊(28只)、夏洛来羊(4只)、本地绵羊(23只)、(夏洛来羊×考力代羊)F1(30只)、(夏洛来羊×考力代羊)F2(24只)、南×(夏×考)F1(28只)、南×(夏×考)F2(21只)、夏洛来羊×本地绵羊(6只)、南非肉用美利奴羊×本地绵羊(12只)。颈静脉采血5 mL,肝素钠抗凝(1 mL),-70℃条件下保存。
1.2 方法 参考《分子克隆实验指南》,进行血液DNA提取。选出多态信息含量高、杂合度高、符合孟德尔遗传规律的微卫星基因座进行PCR扩增,并对PCR产物进行检测、电泳分型。以ΦX174DNA/Hinf I为分子量标准,用凝胶成像系统携带的Alphamager(v 5.1)软件计算微卫星等位基因片段大小。
1.3 数据处理与统计方法 采用FSTAT(Version 2.9.3)软件[4]计算有效等位基因数(ne)、等位基因丰度(rs)、多态信息含量(PIC)、观察的杂合度(Ho)、基因多样性(Hs)以及Wright的F-统计量;用GeneClass2软件[5]进行个体的分配测定。
2 结果与分析
2.1 品种内遗传变异 4个微卫星基因座在10个绵羊种群中共检测到41个等位基因,其中,BP33、CSSM018、IDVGA46和MCM148基因座分别检测到15、7、10和9个等位基因数。说明所选择微卫星标记的遗传信息比较丰富。各基因座的有效等位基因数(ne)、等位基因丰度(rs)和基因多态性信息含量(PIC)见表1。
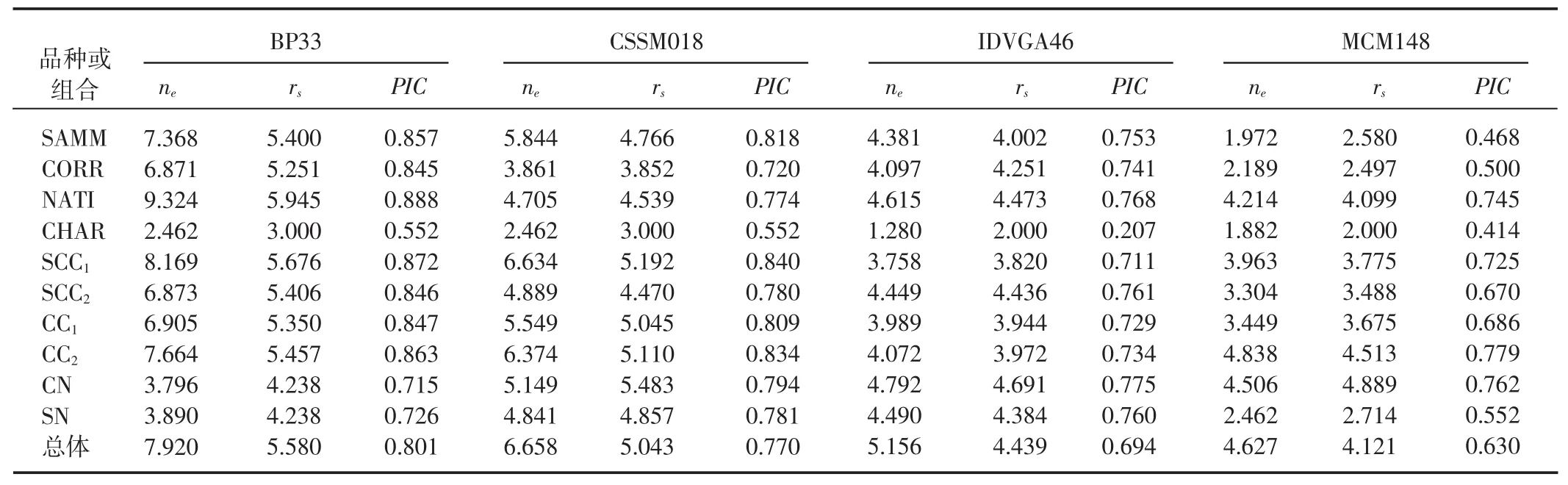
表1 不同群体中微卫星基因座的有效等位基因数(ne)、等位基因丰度(rs)和多态信息含量(PIC)
总群体的平均杂合度(Ht)和经样本校正的平均杂合度(Ht′)、种群内平均杂合度(Hs)以及平均观察杂合度(Ho)见表 2。Ht、Ht′、Hs和 Ho 的总体均数分别为 0.833、0.838、0.789和0.477。从各基因座来看,以BP33的杂合度最大,而MCM148的杂合度最小。
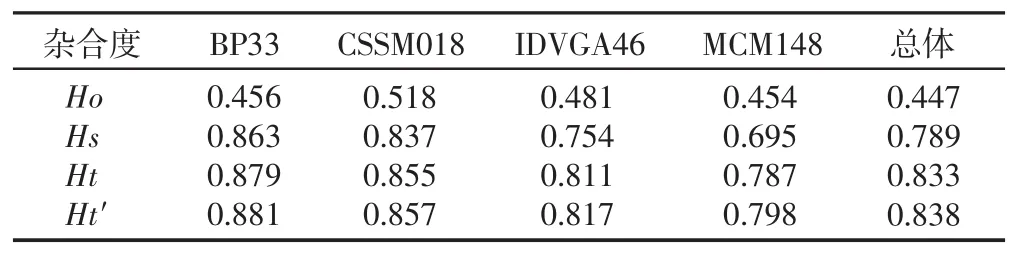
表2 微卫星基因座的杂合度估值
2.2 种群间的遗传分化 用来衡量群体间遗传分化程度的F-统计量估计值列于表3。就不同基因座而言,Fst介于0.069~0.106之间,总体均值0.092,说明群体的总变异有9.2%来自种群间的差异,而剩余的90.8%来自种群内。Fis介于0.311~0.460间,总体均值0.359,表明各种群均存在较高程度的非随机交配;Fit介于0.391~0.478间,总体均值0.424,则反映群体有较高的总体近交系数。
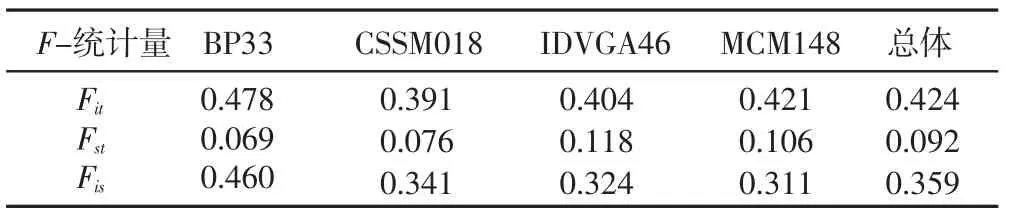
表3 各微卫星基因座Wright的F-统计量
2.3 分配测定 分配测定是将某些个体分配到它最可能所在群体的一种方法。利用4个微卫星的信息用3类方法进行个体分配测定的结果见表4。由表4可见,正确分配率介于19.18%~32.42%。以DA距离法和绳距法最为准确,正确分配率均为32.42%,两种贝叶斯方法,正确分配率也在30%以上。
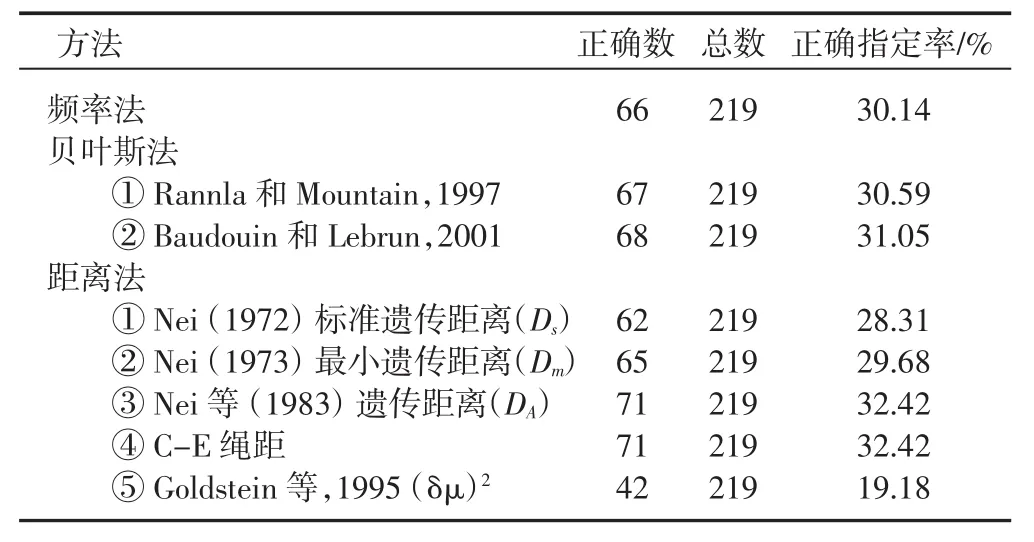
表4 利用4个微卫星进行分配测定的准确性
当利用单标记信息用上述正确分配率最高的方法进行分配测定时,发现测定的准确性因标记基因座的不同而有所不同(表5)。对于BP33和IDVGA46,贝叶斯方法较好,其它两个标记以距离法为优。
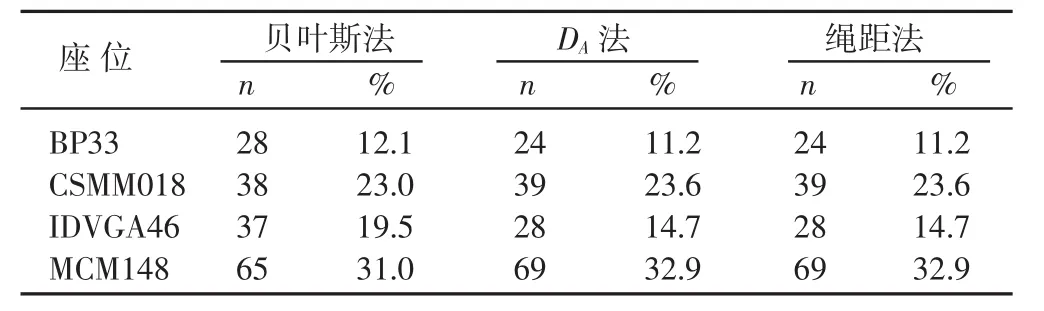
表5 利用单个微卫星进行分配测定的准确性
3 讨论与结论
3.1 群体遗传多样性 多态性信息含量(PIC)是衡量基因多态性的较好指标。Botstein等首先提出了衡量基因变异程度高低的多态性信息含量指标[6]:当PIC>0.50时,该位点为高度多态位点;0.25<PIC<0.50时,为中度多态性位点;PIC<0.25时,为低度多态位点。本研究所选4个微卫星标记在10个绵羊种群中的PIC介于0.630~0.801之间,均值为0.72,均属于高度多态,表明所选的微卫星标记可用于绵羊品种之间遗传多样性和群体遗传结构分析。Tomasco等[7]的研究结果与本研究所得到的PIC值相近;而 Buchanan等[8]和贾斌等[9]的研究中 PIC 值均较本研究低。本文中PIC值较大的原因可能有:①所选标记基因座与其它研究不同;②所选种群只有4个纯种,而其它都是杂种。
杂合度又称为基因多样性,反映各群体在多个基因座上的遗传变异,一般认为它是度量群体遗传变异的一个最适参数。本研究根据Nei[10]估计的整个群体的平均杂合度Ht和Ht′分别为0.833和0.838,种群内平均杂合度Hs和平均观察杂合度Ho分别为0.789和0.477。其中,表示基因多样性的Hs较Gutierrez-Espeleta等利用10个微卫星标记估计的沙漠大角羊 (desert bighorn sheep)的基因多样性(0.51)[11]明显大,但与 Farid 等用10个微卫星基因座研究10个绵羊品种的结果(0.74)[12]、Grigaliunaite等利用15个微卫星标记研究7个绵羊品种的结果(0.712)[13]相近。沙漠大角羊属于野生种群,由于种群间的交流有限甚至会出现严重近交的情况,因而遗传多样性较低。而其他群体均为家养绵羊,而且本研究中还有多个杂种,所以遗传多样性较高。
3.2 群体遗传分化 Fst称为固定指数,又称为基因分化系数,是反映各亚群间遗传分化的重要指标。Fst总是非负的。Wright认为:若Fst在0~0.05之间,表明各亚群间不存在分化;若Fst介于0.05~0.15间,为中度分化;若Fst在 0.15~0.25 之间,则为高度分化[14]。本研究中,Fst的总体均值为0.092,即群体的总变异中,大约有9.2%是来自种群间的差异,而剩余的90.8%则来自种群内。与Soheir等对埃及家养绵羊品种Fst估值(0.037)[15]和耿岩等对中国蒙系 6 个绵羊品种 Fst估值(0.039)[16]相比,本研究中各种群间的分化程度要大。主要原因可能是本研究中有多个杂种群体,由此导致了群体间的遗传分化增大。
Fis也称为内近交系数,用来衡量各种群内因非随机交配引起的个体杂合度的降低;Fit则反映相对于总群体个体的总体近交程度。Fit和Fis可以为正值,也可以为负值[14],前者表示群体内存在近交,杂合度缺失;后者则表示远交,杂合度过量。本研究估计的Fis、Fit均为正值,总体上来说,各种群内存在一定程度的近交。
3.3 绵羊群体的分配测定 本研究基于4个微卫星用3类方法进行的分配测定表明,以DA距离法和绳距法最为准确,但正确分配率仅为30%多,远低于Arranz等利用18个微卫星用同样的方法所得到的正确分配率(98%以上)[17]。当用3种正确分配率最高的方法,用单标记信息进行分配测定时,仅MCM148一个标记的正确分配率就达32.9%,甚至高出利用4个标记信息的正确分配率。这说明正确分配率的高低与标记的选取和数目有关。
[1]Ali S,Muller C R,Eppkn J T.DNA fringarprinting by oligonucletide probes specific for simple repeats[J].Hum Genet,1986,74:239-243.
[2]Arranz J J,Bayón Y,San Primitivo F.Differentiation among Spanish sheep breeds using microsatellites[J].Genet Sel Evol,2001,33:529-542.
[3]Arranz J J,Bayón Y,San Primitivo F.Genetic relationships among Spanish sheep using microsatellite[J].Anim Genet,1998,29:435-440.
[4]Goudet J.FSTAT,a program to estimate and test gene diversities and f zixation indices(Version 2.9.3)[EB/OL].2001.http://www.unil.ch/izea/softwares/fstat.html.
[5]Piry S,Alapetite A,Cornuet J M,et al.GENECLASS2:a software for genetic assignment and first-generation migrant detection[J].J Hered,2004,95:536-539.
[6]Bostein D,White R L,Skollnick M,et al.Construction of a genetic linkage map in man using restriction fragment length polymorphism[J].Am J Hum Genet,1980,32:314-331.
[7]Tomasco I,Wlasiuk G,Lessa E P.Evaluation of polymorphism in ten microsatellite loci in Uruguayan sheep flocks[J].Genet Mol Biol,2002,25:37-41.
[8]Buchanan F C,Thue T D.Intrabreed polymorphic information content of microsatellites in cattle and sheep[J].Can J Anim Sci,1998,78:425-428.
[9]贾斌,陈杰,赵茹茜,等.新疆8个绵羊品种遗传多样性和系统发生树关系的微卫星分析[J].遗传学报,2003,30:847-854.
[10]Nei M.Molecular Evolutionaly Genetics[M].New York:Columbia University Press,1987.
[11]Gutierrez-Espeleta G A,Kalinoeski S T,Boyce W M,et al.Genetic variation and population structurein desertbighorn sheep:implications for conservation[J].Conserv Genet,2000,1:3-15.
[12]Farid A,O’Reilly E,Dollard C,et al.Genetic analysis of ten sheep breeds using microsatellite markers[J].Can J Anim Sci,2000,80:9-17.
[13]Grigaliunaite I,Tapio M,Viinalass H,et al.Microsatellite variation in the Baltic sheep breeds[J].Veterinarija Ir Zootechnika,2003,21:66-73.
[14]Wright S.Evolution and the genetics of populations.vol.4.Variability within and among natural populations[M].Chicago:University of Chicago Press,1978.
[15]Soheir M E,Nahas A A,Hassan A A,et al.Analysis of genetic variation in different sheep breeds using microsatellites[J].Afr J Biotechnol,2008,7(8):1060-1068.
[16]耿岩,杨章平,常洪,等.中国蒙系6个绵羊品种的遗传分化和基因流[J].扬州大学学报:农业与生命科学版,2007,28(3):22-26.
[17]Arranze J J,Bayon Y,Primitivo F S.Differentiation among Spanish sheep breeds using microsatellites[J].Genet Sel Evol,2001,33:529-542.
